Agréger et visualiser les données épidémiologiques
Dernière mise à jour le 2025-11-11 | Modifier cette page
Vue d'ensemble
Questions
- Comment agréger et résumer les données épidémiologiques ?
- Comment visualiser les données agrégées ?
- Quelle est la répartition des cas dans le temps, l’espace, en fonction du sexe, l’âge, etc ?
Objectifs
- Simuler des données synthétiques sur les épidémies
- Convertir un tableau de données des cas individuels (linelist) en tableau d’incidence des cas au cours du temps
- Créer des courbes épidémiques à partir de données d’incidence
Introduction
Dans un pipeline d’analyse de données, l’analyse exploratoire des données (AED) est une étape importante avant la modélisation mathématique. L’AED aide à de déterminer les relations entre les variables et de résumer leurs principales caractéristiques, souvent en visualisant les données.
Dans cet épisode, nous allons explorer les techniques d’analyse
exploratoire des données épidémiologiques en utilisant des librairies de
R.
L’un des aspects clés de l’analyse des données épidémiologiques est la notion de “personne, lieu et temps”. Il est important d’identifier comment les événements observés - tels que les nombres de cas confirmés, d’hospitalisations, de décès et de guérisons - évoluent au cours du temps et comment ils varient en fonction des différents localités et facteurs démographiques, notamment le sexe, l’âge, etc.
Commençons par charger la librairie simulist pour
simuler des données épidémiologiques à analyser. Nous utiliserons
ensuite la librairie incidence2 permettant d’agréger un
tableau des données individuelles en fonction de caractéristiques
spécifiques et de visualiser les courbes épidémiques (épicourbes) qui en
résultent. Le tableau de données agrégées obtenu représente le nombre de
nouveaux événements (c’est-à-dire l’incidence des cas au fil du temps).
Pour le formatage des figures, nous utiliserons la librairie
{tracetheme}. Nous utiliserons l’opérateur pipe
(%>%) pour connecter certaines de leurs fonctions, y
compris celles des packages dplyr et
ggplot2, ferons donc faire appel à la librairie
{tidyverse}.
R
# Charger les librairies
library(incidence2) # Pour aggréger et visualiser les données
library(simulist)
library(tracetheme)
library(tidyverse)
L’opérateur double deux-points
(::)
L’opérateur :: de R permet d’accéder aux fonctions ou
aux objets d’un package spécifique sans attacher l’intégralité du
package (sans faire appel à la functionlibray()). Il offre
plusieurs avantages, notamment :
- Indiquer explicitement le package d’origine d’une fonction, réduisant ainsi les ambiguïtés et les conflits potentiels lorsque plusieurs packages possèdent des fonctions portant le même nom.
- Permettre d’appeler une fonction depuis un package sans charger
l’intégralité du package avec
library().
Par exemple, la commande dplyr::filter(data, condition)
signifie que nous appelons la fonction filter() depuis la
librairie dplyr.
Données synthétiques sur les épidémies
Pour illustrer le processus d’analyse exploratoire des données épidémiologiques, nous allons générer un tableau de données d’une épidémie hypothétique à l’aide de la librairie simulist. simulist génère des données simulées pour une épidémie selon la configuration définie. Sa configuration par défaut permet de générer un tableau de données individuelles comme le montre le morceau de code ci-dessous.
Pour rappelle, vous pouvez désormais utiliser la librairie {readepi} pour importer vos données portant sur les cas individuels á partir des systèmes de gestion de base de données relationnelles, de DHIS2 et SORMAS.
R
# initialiser le générateur de nombres aléatoires pour la reproducibilité
set.seed(1)
# simuler un tableau de données
# pour une épidémie de la taille de 1000 à 1500 cas
sim_data <- simulist::sim_linelist(outbreak_size = c(1000, 1500)) %>%
dplyr::as_tibble()
AVERTISSEMENT
Warning: Number of cases exceeds maximum outbreak size.
Returning data early with 1546 cases and 3059 total contacts (including cases).R
# Voir un aperçu des donnees simulees
sim_data
SORTIE
# A tibble: 1,546 × 13
id case_name case_type sex age date_onset date_reporting
<int> <chr> <chr> <chr> <int> <date> <date>
1 1 Zahra al-Masri probable f 37 2023-01-01 2023-01-01
2 3 Waleeda al-Muhammad probable f 12 2023-01-11 2023-01-11
3 6 Rhett Jackson confirmed m 53 2023-01-18 2023-01-18
4 8 Sunnique Sims confirmed f 36 2023-01-23 2023-01-23
5 11 Danielle Griggs probable f 77 2023-01-30 2023-01-30
6 14 Mohamed Parker probable m 37 2023-01-24 2023-01-24
7 15 Melissa Eriacho probable f 67 2023-01-31 2023-01-31
8 16 Maria Laughlin probable f 80 2023-01-30 2023-01-30
9 20 Phillip Park confirmed m 70 2023-01-27 2023-01-27
10 21 Dewarren Newton probable m 87 2023-02-09 2023-02-09
# ℹ 1,536 more rows
# ℹ 6 more variables: date_admission <date>, outcome <chr>,
# date_outcome <date>, date_first_contact <date>, date_last_contact <date>,
# ct_value <dbl>Ce tableau de données contient des enregistements simulés concernant des individus répertoriés lors d’une épidémie.
Ce qui précède est la configuration par défaut de
simulist. Elle suppose un certain nombre d’hypothèses sur
la transmissibilité et la gravité de l’agent pathogène. Si vous
souhaitez en savoir plus sur la fonction
simulist::sim_linelist() et les autres fonctions de cette
librairie, consultez le site avec la
documentation.
Vous pouvez également télécharger des jeux de données
épidémiologiques réelles concernant des épidémies antérieures dans la
librairie {outbreaks}
de R.
Agrégation des données
Nous voudrons généralement analyser et visualiser le nombre de cas
enregistrés en un jour ou une semaine, plutôt que de nous concentrer sur
chaque cas individuel. Pour ce faire, il est nécessaire de regrouper le
tableau des données individuelles en tableau de données d’incidence. La
fonction incidence2::incidence() de la librairie
[{incidence2}] (([https://www.reconverse.org/incidence2/articles/incidence2.html)
permet de regrouper les données individuelles, généralement en fonction
d’événements datés et/ou d’autres caractéristiques. Le morceau de code
fourni ci-dessous montre la procédure de création d’un objet de la
classe incidence2 qui contiendra le nombre agrégé de cas à
partir de la date d’apparition de leurs symptômes en utilisant le
tableau de données d’Ebola simulé.
R
# creer un objet de classe incidence
# en agregeant le nombre de cas
# en fonction de la date d'apparition des symptomes
daily_incidence <- incidence2::incidence(
sim_data,
date_index = "date_onset",
interval = "day" # Aggreger les données de façon journalière
)
# Voir un aperçu des données d'incidence
daily_incidence
SORTIE
# incidence: 232 x 3
# count vars: date_onset
date_index count_variable count
<date> <chr> <int>
1 2023-01-01 date_onset 1
2 2023-01-11 date_onset 1
3 2023-01-18 date_onset 1
4 2023-01-23 date_onset 1
5 2023-01-24 date_onset 1
6 2023-01-27 date_onset 2
7 2023-01-29 date_onset 1
8 2023-01-30 date_onset 2
9 2023-01-31 date_onset 2
10 2023-02-01 date_onset 1
# ℹ 222 more rowsLa librairie incidence2 vous permet de varier l’intervalle de temps à utiliser lorsque vous agrégez vos données (par exemple, les données peuvent être agrégées de façon journalière, hebdomadaire, mensuelle, etc). Elle permet également de classer les cas selon un ou plusieurs facteurs. L’exemple ci-dessous est un extrait de code calculant le nombre hebdomadaire de cas regroupés selon la date d’apparition des symptômes, le sexe et le type de cas.
R
# calculer l'incidence hebdomadaire
# des cas regroupés selon le sexe et le type de cas
weekly_incidence <- incidence2::incidence(
sim_data,
date_index = "date_onset",
interval = "week", # obtenir le nombre hebdomadaire de cas
groups = c("sex", "case_type") # regrouper par sexe et type de cas
)
# voir un aperçu de l'incidence hebdomadaire des cas
weekly_incidence
SORTIE
# incidence: 201 x 5
# count vars: date_onset
# groups: sex, case_type
date_index sex case_type count_variable count
<isowk> <chr> <chr> <chr> <int>
1 2022-W52 f probable date_onset 1
2 2023-W02 f probable date_onset 1
3 2023-W03 m confirmed date_onset 1
4 2023-W04 f confirmed date_onset 2
5 2023-W04 f probable date_onset 1
6 2023-W04 m confirmed date_onset 1
7 2023-W04 m probable date_onset 1
8 2023-W05 f confirmed date_onset 3
9 2023-W05 f probable date_onset 4
10 2023-W05 f suspected date_onset 1
# ℹ 191 more rowsCompléter les dates manquantes
Lorsque les cas sont regroupés en fonction de différents facteurs, il
est possible que les événements impliquant ces groupes se produisent à
des dates différentes dans l’objet de type incidence2 qui
en résulte. La fonction incidence2::complete_dates() permet
de insérer les dates manquantes pour chaque groupe de l’objet de la
classe incidence. Par défaut, le nombre de cas de ces groupes sera
représenté par 0 pour cette date (où aucun membre de ces groupe n’avait
été enregistré). Cette fonctionnalité peut également être activées
durant l’appel à la fonction incidence2::incidence() en
donnant la valeur TRUE à l’argument
complete_dates.
R
# creer un objet de la classe incidence
# contenant l'incidence journalière du
# nombre de cas, groupé par sexe
daily_incidence_2 <- incidence2::incidence(
sim_data,
date_index = "date_onset",
groups = "sex",
interval = "day", # Agreger par interval journalier
complete_dates = TRUE # Compléter les dates manquantes
)
# voir un aperçu de l'incidence journaliere des cas
daily_incidence_2
SORTIE
# incidence: 534 x 4
# count vars: date_onset
# groups: sex
date_index sex count_variable count
<date> <chr> <chr> <int>
1 2023-01-01 f date_onset 1
2 2023-01-01 m date_onset 0
3 2023-01-02 f date_onset 0
4 2023-01-02 m date_onset 0
5 2023-01-03 f date_onset 0
6 2023-01-03 m date_onset 0
7 2023-01-04 f date_onset 0
8 2023-01-04 m date_onset 0
9 2023-01-05 f date_onset 0
10 2023-01-05 m date_onset 0
# ℹ 524 more rowsDéfi 1 : Pouvez-vous le faire ?
-
Tâche : calculer l’incidence
bihebdomadaire des cas dans le tableau de donnèes
sim_dataen fonction de leur date d’admission et de leur revenu. Sauvegarder le résultat dans un objet appelébiweekly_incidence.
Visualisation
Les object de la classe incidence2 peuvent être
visualisés à l’aide de la fonction plot() de la librairie
de base de R. Le graphe qui en résulte est appelé courbe épidémique, ou
épi-courbe. Le code ci-dessous génère des épi-courbes pour lee objets
daily_incidence et weekly_incidence créés
ci-dessus.
R
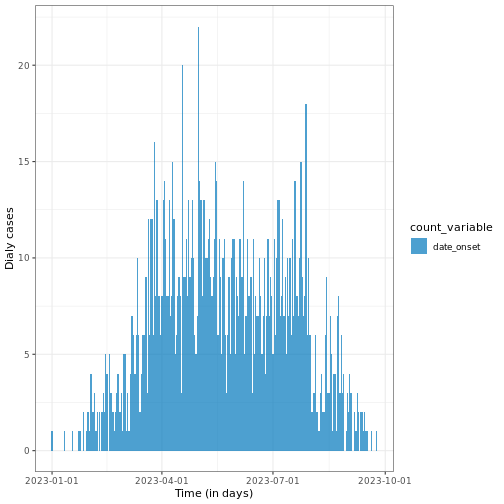
# visualiser l'incidence journaliere
base::plot(daily_incidence) +
ggplot2::labs(
x = "Time (in days)",
y = "Dialy cases"
) +
tracetheme::theme_trace() # appliquer le theme personnalise de epiverse
R
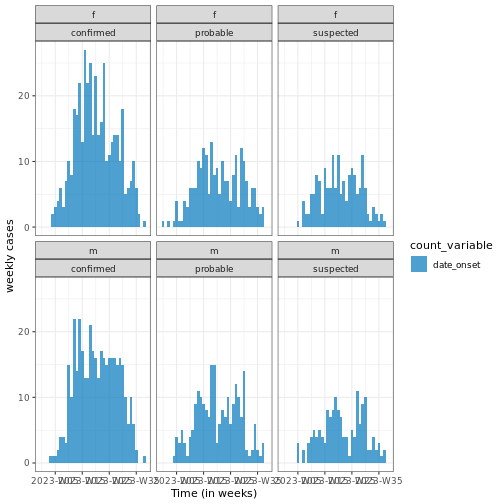
# visualiser l'incidence hebdomadaire
base::plot(weekly_incidence) +
ggplot2::labs(
x = "Time (in weeks)",
y = "weekly cases" #
) +
tracetheme::theme_trace()
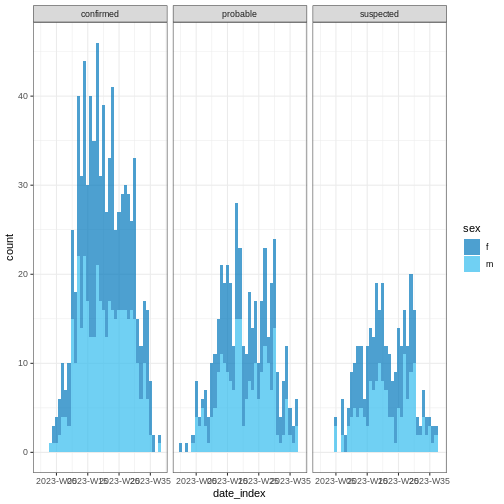
Esthétique simple
Nous vous invitons à parcourir la Vignette
de la librairie incidence2. Découvrez comment vous pouvez
utiliser les arguments de la fonction plot() pour donner de
l’esthétique lors de la visualisation de vos objets de la classe
incidence2.
R
base::plot(weekly_incidence, fill = "sex")
Vous y trouverez entre autre argument:
show_cases = TRUE, angle = 45 et
n_breaks = 5. Essayer de les utiliser pour voir comment ils
changent l’apparence du graphe qui en découle.
Défi 2 : Pouvez-vous le faire ?
-
Tâche visualiser l’objet
biweekly_incidence.
Courbe du nombre cumulé de cas
Le nombre cumulé de cas peut être calculé à l’aide de la fonction
incidence2::cumulate() à partir d’un objet de la classe
incidence2 et peut être visualisé de la même façon que dans
les exemples précédents.
R
# calculer l'incidence cumulée
cum_df <- incidence2::cumulate(daily_incidence)
# visualiser l'incidence cumulée avec {ggplot2}
base::plot(cum_df) +
ggplot2::labs(
x = "Time (in days)",
y = "weekly cases" #
) +
tracetheme::theme_trace()
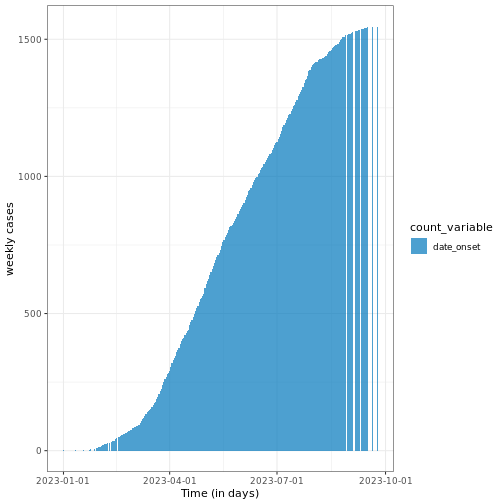
Notez que cette fonction préserve le regroupement, c’est-à-dire que
si l’objet de la classeincidence2 contient des groupes,
elle accumulera les cas en conséquence.
Défi 3 : Pouvez-vous le faire ?
-
Tâche visuliser le nombre cumulé de cas de l’objet
biweekly_incidence.
Estimation la date du pic de l’épidémie
Vous pouvez estimer le pic - le moment où le nombre de cas
enregistrés est le plus élevé - à l’aide de la fonction
incidence2::estimate_peak() de la librairie {incidence2}.
Cette fonction utilise une méthode de bootstrap pour déterminer la
période de pic du nombre de cas (c’est-à-dire en rééchantillonnant les
dates avec remplacement, ce qui donne une distribution des périodes de
pic estimés).
R
# estimer le pic de l'incidence journaliere
peak <- incidence2::estimate_peak(
daily_incidence,
n = 100, # nombre de simulations à utiliser pour trouver le pic
alpha = 0.05, # niveau de significativite pour l'interval de confiance
first_only = TRUE, # retourner la valeur du premier pic trouvé
progress = FALSE # Désactiver les messages de progression
)
# voir un aperçu du pic estime
print(peak)
SORTIE
# A tibble: 1 × 7
count_variable observed_peak observed_count bootstrap_peaks lower_ci
<chr> <date> <int> <list> <date>
1 date_onset 2023-05-01 22 <df [100 × 1]> 2023-03-26
# ℹ 2 more variables: median <date>, upper_ci <date>Cet exemple montre comment estimer la date du pic de l’épidémie (date
à laquelle le nombre de cas le plus élevé a été répertorié) à l’aide de
la fonction incidence2::estimate_peak() avec un interval de
confiance de \(95%\) et en
ré-échantillonnant 100 fois.
Défi 4 : Pouvez-vous le faire ?
-
Tâche Estimation la période du pic de l’épidémie à
partir de l’objet
biweekly_incidence.
Visualisation avec ggplot2
incidence2 permet d’obtenir des graphes basiques pour les épi-courbes, mais un travail supplémentaire est nécessaire pour créer des graphiques bien annotés. Cependant, en urilisant la librairie ggplot2 vous pouvez générer des graphes plus sophistiqués et des épi-courbes avec plus de flexibilité dans leur annotation. ggplot2 est un package qui offre de nombreuses fonctionnalités. Cependant, nous nous focaliserons sur trois éléments clés pour la production d’épi-courbes : les histogrammes, la mise à l’échelle des axes des variables de type Date et de leurs étiquettes, et l’annotation générale du thème du graphe. L’exemple ci-dessous montre comment configurer ces trois éléments pour un simple objet de la classe incidence2.
R
# definir comment les dates seront reparties sur l'axe des x
breaks <- seq.Date(
from = min(as.Date(daily_incidence$date_index, na.rm = TRUE)),
to = max(as.Date(daily_incidence$date_index, na.rm = TRUE)),
by = 20 # chaque 20 jours
)
# creation de l'histogramme
ggplot2::ggplot(data = daily_incidence) +
geom_histogram(
mapping = aes(
x = as.Date(date_index),
y = count
),
stat = "identity",
color = "blue", # couleur de bordure de l'histogramme
fill = "lightblue", # couleur de remplissage des bars de l'histogramme
width = 1 # l'epaisseur des bars de l'histogramme
) +
theme_minimal() + # utiliser un theme minimal
theme(
plot.title = element_text(face = "bold",
hjust = 0.5), # centrer le titre du gaphe en gras
plot.subtitle = element_text(hjust = 0.5), # centrer le sous-titre
plot.caption = element_text(face = "italic",
hjust = 0), # mettre la legende en italique
axis.title = element_text(face = "bold"), # mettre les noms des axes en gras
axis.text.x = element_text(angle = 45, vjust = 0.5) # tourner des 45 degrés
) +
labs(
x = "Date", # definir le nom de l'axe des x
y = "Number of cases", # definir le nom de l'axe des y
title = "Daily Outbreak Cases", # definir le titre du graphe
subtitle = "Epidemiological Data for the Outbreak",
caption = "Data Source: Simulated Data" # definir la legende du graphe
) +
scale_x_date(
breaks = breaks, # definir les limites personnalisee pour l'axe des x
labels = scales::label_date_short() # Raccourcir les noms sur l'axe x
)
AVERTISSEMENT
Warning in geom_histogram(mapping = aes(x = as.Date(date_index), y = count), :
Ignoring unknown parameters: `binwidth`, `bins`, and `pad`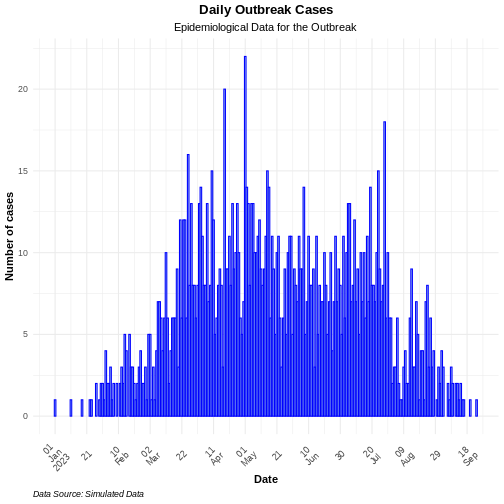
Utilisez l’option group dans la fonction mapping pour
visualiser une courbe épidémique avec différents groupes. Si plusieurs
variables ont été utilisées pour regrouper les cas, utilisez l’option
facet_wrap() comme le montre l’exemple ci-dessous :
R
# visualiser l'incidence journaliere avec des facettes liées au sexe
ggplot2::ggplot(data = daily_incidence_2) +
geom_histogram(
mapping = aes(
x = as.Date(date_index),
y = count,
group = sex, # grouper les bars par sexe
fill = sex
),
stat = "identity"
) +
theme_minimal() +
theme(
plot.title = element_text(face = "bold", hjust = 0.5),
plot.subtitle = element_text(hjust = 0.5),
plot.caption = element_text(face = "italic", hjust = 0),
axis.title = element_text(face = "bold"),
axis.text.x = element_text(angle = 45, vjust = 0.5)
) +
labs(
x = "Date",
y = "Number of cases",
title = "Daily Outbreak Cases by Sex",
subtitle = "Incidence of Cases Grouped by Sex",
caption = "Data Source: Simulated Data"
) +
facet_wrap(~sex) + # creer un panel separant les groupes de la variable sexe
scale_x_date(
breaks = breaks,
labels = scales::label_date_short()
) +
scale_fill_manual(values = c("lightblue",
"lightpink")) # couleur personnalisees
AVERTISSEMENT
Warning in geom_histogram(mapping = aes(x = as.Date(date_index), y = count, :
Ignoring unknown parameters: `binwidth`, `bins`, and `pad`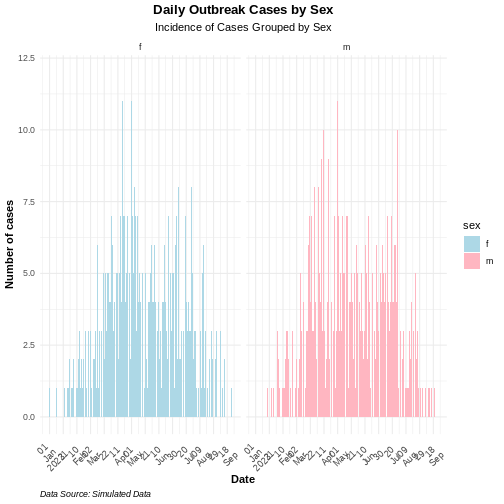
Défi 5 : Pouvez-vous le faire ?
-
Tâche: produire une figure annotée pour l’objet
contenant les incidences bihebdomadiares
biweekly_incidenceà l’aide de la librairie ggplot2.
Points clés
- Utiliser simulist pour générer des données épidémiologiques synthétiques
- Utilisez incidence2 pour agréger les données épidémiologiques en fonction d’événements datés et d’autres variables pour produire des épi-courbes.
- Utilisez ggplot2 pour produire des courbes épidémiques mieux annotées.
