Nettoyage des données épidémiologiques
Dernière mise à jour le 2025-11-11 | Modifier cette page
Durée estimée : 30 minutes
Vue d'ensemble
Questions
- Comment nettoyer et standardiser les données épidémiologiques ?
Objectifs
- Expliquez comment nettoyer, organiser et standardiser les données épidémioloques en utilisant la librairie cleanepi
- Appliquer les opérations de nettoyage de données sur des données épidémiologiques simulées
Pré-requis
Pour cet épisode, nous utiliserons les données simulées portant sur des individus atteints de la fièvre hémorargique ébola que vous devez:
- d’abord télécharger à partir d’ici,
- puis l’enregistrez-le dans le répertoire
data/.
Introduction
Lors de l’analyse de données épidémiologiques, il est essentiel de s’assurer que les des données sont nettoyées, organisées, standardisées et validées pour garantir la précision (vous analysez ce que vous pensez analyser) et la reproductibilité (si quelqu’un veut revenir en arrière et répéter vos étapes d’analyse avec votre code, vous pouvez être sûr qu’il obtiendra les mêmes résultats) de l’analyse. Cet épisode est consacré au nettoyage de données relatives aux épidémies à l’aide du package R {cleanepi}. Pour illustrer l’utilisation des fonctionalités de ce package, nous les appliquerons sur des données simulées de cas d’Ebola.
En plus du package {cleanepi}, nous utiliserons d’autres fonctions qui proviennent des packages R suivants durant cette procédure de nettoyage des données :
- here pour faciliter le référencement des fichiers,
- rio pour importer les données dans R,
- dplyr pour effectuer certaines opérations de traitement de données,
-
magrittr pour utiliser son opérateur pipe
(
%>%).
R
# charger les librairies
library(tidyverse)
library(rio)
library(here)
library(cleanepi)
L’opérateur double deux-points
(::)
L’opérateur :: de R permet d’accéder aux fonctions ou
aux objets d’un package spécifique sans attacher l’intégralité du
package (sans faire appel à la functionlibray()). Il offre
plusieurs avantages, notamment :
- Indiquer explicitement le package d’origine d’une fonction, réduisant ainsi les ambiguïtés et les conflits potentiels lorsque plusieurs packages possèdent des fonctions portant le même nom.
- Permettre d’appeler une fonction depuis un package sans charger
l’intégralité du package avec
library().
Par exemple, la commande dplyr::filter(data, condition)
signifie que nous appelons la fonction filter() depuis la
librairie dplyr.
La première étape consiste à importer le jeu de données dans
l’environnement de travail. Pour ce faire, nous suivrons les
instructions décrites dans l’épisode Importer
les données de cas épidémiologiques pour importer les données dans
R et visualiser sa structure et son contenu.
R
# Importer les données
# supposons que le chemin d'accès au ficher est: data/simulated_ebola_2.csv
raw_ebola_data <- rio::import(
here::here("data", "simulated_ebola_2.csv")
) %>%
dplyr::as_tibble() # convertir en tibble
R
# Afficher les dix premières lignes du data frame
raw_ebola_data
SORTIE
# A tibble: 15,003 × 9
V1 `case id` age gender status `date onset` `date sample` lab region
<int> <int> <chr> <chr> <chr> <chr> <chr> <lgl> <chr>
1 1 14905 90 1 "conf… 03/15/2015 06/04/2015 NA valdr…
2 2 13043 twenty… 2 "" Sep /11/13 03/01/2014 NA valdr…
3 3 14364 54 f <NA> 09/02/2014 03/03/2015 NA valdr…
4 4 14675 ninety <NA> "" 10/19/2014 31/ 12 /14 NA valdr…
5 5 12648 74 F "" 08/06/2014 10/10/2016 NA valdr…
6 5 12648 74 F "" 08/06/2014 10/10/2016 NA valdr…
7 6 14274 sevent… female "" Apr /05/15 01/23/2016 NA valdr…
8 7 14132 sixteen male "conf… Dec /29/Y 05/10/2015 NA valdr…
9 8 14715 44 f "conf… Apr /06/Y 04/24/2016 NA valdr…
10 9 13435 26 1 "" 09/07/2014 20/ 09 /14 NA valdr…
# ℹ 14,993 more rowsDiscussion
Jettons un coup d’oeil sur ce jeu de données pour dressez la liste de toutes les anormalies qui pourraient poser problème durant l’analyse des données.
Certaines de ces anormalies vous sont-elles familières dans le cadre d’une analyse de données que vous avez déjà effectuée ?
Animez une brève discussion pour établir un lien entre les anormalies détectées et les opérations de nettoyage requises.
Vous pouvez utiliser les termes suivants pour désigner les anormalies qui ont été identifiées:
- Codification: codification des valeurs dans certaines colonnes, telles que ‘gender’, ‘age’, à l’aide de chiffres, de lettres et de mots. Également l’existence de plusieur formats de dates ((“jj/mm/aaaa”, “aaaa/mm/jj”, etc)) dans une même colonne telle que ‘date_onset’. Moins visibles, les noms des colonnes.
- Manquant: comment interpréter une entrée telle que “” dans la colonne ‘statut’ ou “-99” dans d’autres circonstances ? Disposons-nous d’un dictionaire de données provenant du processus de collecte des données ?
- Incohérences: avoir des incohérences comme lorsque la date d’échantillonnage est antérieure à la date d’apparition des symptomes.
- Valeurs non plausibles Les valeurs non plausibles, comme les observations aberrantes avec des dates en dehors de la période concernée.
- Les doublons Toutes les observations sont-elles uniques ?
Vous pouvez utiliser ces termes pour vous référer aux opérations de nettoyage:
- Normaliser le nom des colonnes
- Normaliser les variables catégorielles comme ‘gender’
- Normaliser les colonnes de type date
- Convertir des caractères en valeurs numériques
- Vérifier la séquence d’événements datés
Une inspection rapide
Une exploration et une inspection rapides des données sont
essentielles pour identifier les problèmes potentiels avant d’entamer le
processus d’analyse des données. Le package {cleanepi} simplifie ce
processus grâce à la fonction scan_data(). Voyons comment
vous pouvez l’utiliser.
R
cleanepi::scan_data(raw_ebola_data)
SORTIE
Field_names missing numeric date character logical
1 age 0.0646 0.8348 0.0000 0.1006 0
2 gender 0.1578 0.0472 0.0000 0.7950 0
3 status 0.0535 0.0000 0.0000 0.9465 0
4 date onset 0.0001 0.0000 0.9159 0.0840 0
5 date sample 0.0001 0.0000 0.9999 0.0000 0
6 region 0.0000 0.0000 0.0000 1.0000 0Le résultat donne un aperçu du contenu de toutes les colonnes de type charactère, y compris la structure de ces noms de colonnes et le pourcentage de certains types de données qu’on y retrouve. Vous pouvez constater que les noms des colonnes sont descriptifs mais manquent de cohérence. Certains sont composés de plusieurs mots séparés par des espaces. En outre, certaines colonnes contiennent plus d’un type de données, et il y a des valeurs manquantes dans d’autres.
Opérations courantes
Cette section montre comment effectuer quelques opérations courantes de nettoyage de données à l’aide de la librairie cleanepi.
Normalisation des noms de colonnes
Pour cet exemple de jeu de données, la normalisation des noms de
colonnes consiste généralement à supprimer les espaces et à relier les
mots avec "_". Cette pratique permet de maintenir la
cohérence et la lisibilité du jeu de données. Cependant, la fonction
utilisée pour normaliser les noms de colonnes offre plus d’options.
Entrez ?cleanepi::standardize_column_names dans la console
pour plus de détails.
R
sim_ebola_data <- cleanepi::standardize_column_names(raw_ebola_data)
names(sim_ebola_data)
SORTIE
[1] "v1" "case_id" "age" "gender" "status"
[6] "date_onset" "date_sample" "lab" "region" Si vous souhaitez conserver certains noms de colonnes sans les
soumettre au processus de normalisation, vous pouvez utiliser l’argument
keep de la fonction
cleanepi::standardize_column_names(). Cet argument accepte
un vecteur de noms de colonnes qui doivent rester inchangés.
Défi
Quelles différences pouvez-vous observer dans les noms de colonnes ?
Normalisez les noms des colonnes de l’ensemble de données d’entrée, mais conservez le nom de la première colonne tel quel.
Vous pouvez essayer
cleanepi::standardize_column_names(data = raw_ebola_data, keep = "V1")
Suppression des irrégularités
Les données brutes contiennent souvent des irrégularités telles que
des lignes vides et colonnes vides, ou des colonnes
constant (où toutes les entrées ont la même valeur).
Elles peuvent aussi contenir des doublons. Les fonctions de
cleanepi comme remove_duplicates() et
remove_constants() peuvent être utilisées pour supprimer
ces irrégularités, comme le montre le morceau de code ci-dessous.
R
# Supprimer les données invariables
sim_ebola_data <- cleanepi::remove_constants(sim_ebola_data)
Maintenant, afficher le résultat pour identifier les colonnes constantes que vous avez supprimé.
R
# Supprimer les doublons
sim_ebola_data <- cleanepi::remove_duplicates(sim_ebola_data)
SORTIE
! Found 5 duplicated rows in the dataset.
ℹ Use `attr(dat, "report")[["duplicated_rows"]]` to access them, where "dat" is
the object used to store the output from this operation.Vous pouvez obtenir le nombre et l’emplacement des lignes redondantes
qui ont été identifiées à l’aide de la function
cleanepi::print_report().
R
# les doublons identifiés
doublons <- print_report(sim_ebola_data, "found_duplicates")
doublons
Défi
Dans le jeu de données suivant :
SORTIE
# A tibble: 6 × 5
col1 col2 col3 col4 col5
<dbl> <dbl> <chr> <chr> <date>
1 1 1 a b NA
2 2 3 a b NA
3 NA NA a <NA> NA
4 NA NA a <NA> NA
5 NA NA a <NA> NA
6 NA NA <NA> <NA> NA Quelles sont les colonnes ou les lignes:
- contenant des doublons ?
- vides ?
- contenant des données constantes ?
Les données constantes se réfèrent principalement aux lignes et colonnes vides, mais aussi aux colonnes invariables.
R
cleaned_df <- df %>%
cleanepi::remove_constants() %>%
cleanepi::remove_duplicates()
SORTIE
! Constant data was removed after 2 iterations.
ℹ Enter `attr(dat, "report")[["constant_data"]]` for more information, where
"dat" represents the object used to store the output from
`remove_constants()`.
ℹ No duplicates were found.R
print_report(cleaned_df, "constant_data")
- indices des doublon : aucun
- colonnes vides :
- premiere itération: col5
- seconde itération: aucune
- lignes vides :
- premiere itération: 6
- seconde itération: 3, 4, 5
- colonnes constantes : col3, col4
Faites remarquer aux apprenants qu’ils peuvent obtenir des résultats
différents en variant la valeur de l’argument cutoff.
R
cleaned_df <- df %>%
cleanepi::remove_constants(cutoff = 0.5)
R
print_report(cleaned_df, "constant_data")
Remplacer les valeurs manquantes
Outre les irrégularités, les données brutes peuvent contenir des
valeurs manquantes, qui peuvent être representées par différentes
chaînes de caractères (par ex. "NA", "",
character(0)). Pour garantir une analyse solide, il est
conseillé de remplacer toutes les valeurs manquantes par NA
dans l’ensemble du jeu de données. Vous trouverez ci-dessous un extrait
de code démontrant comment vous pouvez remplacer les valeures
manquantes, représentées par une chaîne vide "", en
utilisant la librairie cleanepi:
R
sim_ebola_data <- cleanepi::replace_missing_values(
data = sim_ebola_data,
na_strings = ""
)
sim_ebola_data
SORTIE
# A tibble: 15,000 × 8
v1 case_id age gender status date_onset date_sample row_id
<int> <int> <chr> <chr> <chr> <chr> <chr> <int>
1 1 14905 90 1 confirmed 03/15/2015 06/04/2015 1
2 2 13043 twenty-five 2 <NA> Sep /11/13 03/01/2014 2
3 3 14364 54 f <NA> 09/02/2014 03/03/2015 3
4 4 14675 ninety <NA> <NA> 10/19/2014 31/ 12 /14 4
5 5 12648 74 F <NA> 08/06/2014 10/10/2016 5
6 6 14274 seventy-six female <NA> Apr /05/15 01/23/2016 7
7 7 14132 sixteen male confirmed Dec /29/Y 05/10/2015 8
8 8 14715 44 f confirmed Apr /06/Y 04/24/2016 9
9 9 13435 26 1 <NA> 09/07/2014 20/ 09 /14 10
10 10 14816 thirty f <NA> 06/29/2015 06/02/2015 11
# ℹ 14,990 more rowsValidation des identifiants des individus
Chaque entrée du jeu de données représente un individu (par exemple,
un patient ou un participant à une étude) et doit pouvoir être
distinguée par un identifiant unique formatée d’une certaine façon. Ces
identifiants peuvent contenir des nombres se situant dans un intervalle
bien déterminé, a préfixe et/ou suffixe, être formatés de façon à
contenir un nombre spécifique de caractères. La librairie
cleanepi propose la fonction
check_subject_ids() permettant de vérifier si les
identifiants des individus sont uniques et s’ils satisfont aux critères
établis par l’utilisateur.
R
# vérifier si les identifiants des individus comportent des nombres
# variant entre 0 et 15000
sim_ebola_data <- cleanepi::check_subject_ids(
data = sim_ebola_data,
target_columns = "case_id",
range = c(0, 15000)
)
SORTIE
! Found 1957 duplicated values in the subject Ids.
ℹ Enter `attr(dat, "report")[["duplicated_rows"]]` to access them, where "dat"
is the object used to store the output from this operation.
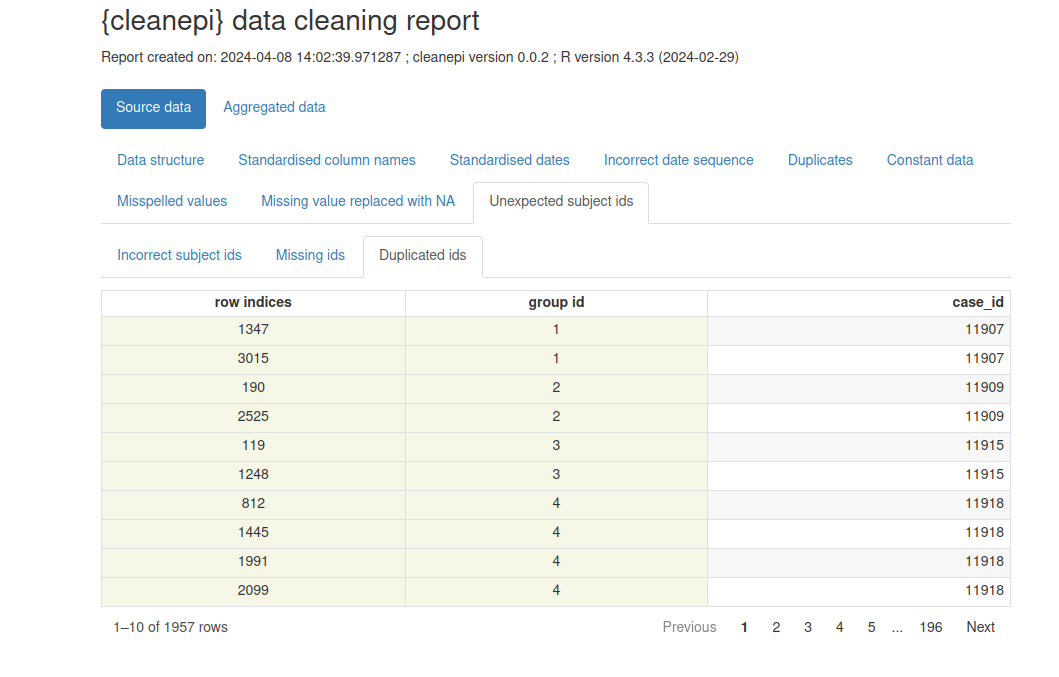
ℹ No incorrect subject id was detected.Notez que notre jeu de données simulé contient des doublons dans la colonne ayant les identifiants uniques des individus.
Visualisons un rapport préliminaire avec
cleanepi::print_report(sim_ebola_data). Concentrez-vous sur
l’onglet “Unexpected subject ids” pour identifier les identifiants qui
nécessitent une correction.
Après avoir terminé ce tutoriel, nous vous invitons à explorer le
guide de référence de la function cleanepi::check_subject_ids()
pour identifier la function qui peut remédier à cette situation.
Normalisation des dates
Un jeu de données épidémiques contient généralement des colonnes de type date pour différents événements, tels que la date d’infection, la date d’apparition des symptômes, etc. Ces dates peuvent apparaître sous différents formats, et il est bon de les normaliser afin de bénéficier des puissantes fonctionalités de R conçues pour traiter les valeurs de type date durant l’analyse des données. La librairies cleanepi fournit une fonctionnalité permettant de convertir les colonnes de type date dans les données épidémiologiques au format ISO8601, garantissant la cohérence des différentes colonnes de type dates. Voici comment vous pouvez l’utiliser sur notre jeu de données simulé :
R
sim_ebola_data <- cleanepi::standardize_dates(
sim_ebola_data,
target_columns = c("date_onset", "date_sample")
)
sim_ebola_data
SORTIE
# A tibble: 15,000 × 8
v1 case_id age gender status date_onset date_sample row_id
<int> <chr> <chr> <chr> <chr> <date> <date> <int>
1 1 14905 90 1 confirmed 2015-03-15 2015-06-04 1
2 2 13043 twenty-five 2 <NA> 2013-09-11 2014-03-01 2
3 3 14364 54 f <NA> 2014-09-02 2015-03-03 3
4 4 14675 ninety <NA> <NA> 2014-10-19 2031-12-14 4
5 5 12648 74 F <NA> 2014-08-06 2016-10-10 5
6 6 14274 seventy-six female <NA> 2015-04-05 2016-01-23 7
7 7 14132 sixteen male confirmed NA 2015-05-10 8
8 8 14715 44 f confirmed NA 2016-04-24 9
9 9 13435 26 1 <NA> 2014-09-07 2020-09-14 10
10 10 14816 thirty f <NA> 2015-06-29 2015-06-02 11
# ℹ 14,990 more rowsCette fonction convertit les valeurs dans les colonnes cibles au le format YYYY-mm-dd.
Conversion en valeurs numériques
Dans les données brutes, certaines colonnes peuvent contenir un
mélange de valeurs numériques et de caractères, et vous voudrez souvent
convertir les caractères en numériques. Par exemple, certaines valeurs
de la colonne de ‘age’ de notre jeu de données simulées sont écrites en
mots. Dans cleanepi la fonction
convert_to_numeric() permet d’effectuer la conversion de
ces mots en chiffres, comme illustré ci-dessous.
R
sim_ebola_data <- cleanepi::convert_to_numeric(
data = sim_ebola_data,
target_columns = "age"
)
sim_ebola_data
SORTIE
# A tibble: 15,000 × 8
v1 case_id age gender status date_onset date_sample row_id
<int> <chr> <dbl> <chr> <chr> <date> <date> <int>
1 1 14905 90 1 confirmed 2015-03-15 2015-06-04 1
2 2 13043 25 2 <NA> 2013-09-11 2014-03-01 2
3 3 14364 54 f <NA> 2014-09-02 2015-03-03 3
4 4 14675 90 <NA> <NA> 2014-10-19 2031-12-14 4
5 5 12648 74 F <NA> 2014-08-06 2016-10-10 5
6 6 14274 76 female <NA> 2015-04-05 2016-01-23 7
7 7 14132 16 male confirmed NA 2015-05-10 8
8 8 14715 44 f confirmed NA 2016-04-24 9
9 9 13435 26 1 <NA> 2014-09-07 2020-09-14 10
10 10 14816 30 f <NA> 2015-06-29 2015-06-02 11
# ℹ 14,990 more rowsLangues prise en charge
Il faut savoir que la fonction
cleanepi::convert_to_numeric() fait appel à la fonction
principale de la librairie numberize, qui permet de
convertir, en valeurs entières positives, les mots écrits en anglais,
français et espagnol.
Autres opérations liées à l’épidémiologie
Outre les tâches courantes de nettoyage des données, telles que celles évoquées dans les sections précédentes, la librairie cleanepi offre des fonctionnalités supplémentaires spécialement conçues pour le traitement et l’analyse des données relatives aux épidémies. Cette section couvre certaines de ces tâches spécialisées.
Vérification de la séquence des événements datés
Garantir l’ordre et la séquence correcte des événements datés est
crucial dans l’analyse des données épidémiologiques, en particulier lors
de l’analyse des maladies infectieuses, où la chronologie d’événements
tels que la date d’apparition des symptômes et la date de collecte
d’échantillons est essentielle. La librairie cleanepi
fournit une fonction utile appelée check_date_sequence()
précisément conçu dans ce but.
Voici un exemple de morceau de code démontrant l’utilisation de la
fonction check_date_sequence() dans les 100 premières
lignes de notre ensemble de données Ebola simulé.
R
cleanepi::check_date_sequence(
data = sim_ebola_data[1:100, ],
target_columns = c("date_onset", "date_sample")
)
SORTIE
! Detected 16 incorrect date sequences at lines: "10, 20, 22, 26, 29, 44, 46,
54, 60, 63, 70, 71, 73, 80, 81, 90".
ℹ Enter `attr(dat, "report")[["incorrect_date_sequence"]]` to access them,
where "dat" is the object used to store the output from this operation.Cette fonctionnalité est essentielle pour garantir l’intégrité des données et la précision dans les analyses épidémiologiques, car elle permet d’identifier toute incohérence ou erreur dans l’ordre chronologique des événements, ce qui vous permet d’y remédier de manière appropriée.
Substitution basée sur un dictionnaire
Dans le cadre du prétraitement des données, il est fréquent de rencontrer des scénarios dans lesquels certaines colonnes d’un jeu de données, comme la colonne “gender” dans notre jeu de données Ebola simulé, sont censées avoir des valeurs ou des facteurs spécifiques. Cependant, il est également fréquent que des valeurs inattendues ou erronées apparaissent dans ces colonnes, et doivent être remplacées par des valeurs de valeurs appropriées. La librairie cleanepi offre une fonction qui prend en charge la substitution de valeurs basée sur un dictionnaire données. Ainsi, vous permet de remplacer des valeurs dans des colonnes spécifiques sur la base de correspondances définies dans un dictionnaire de données.
Par ailleurs, la librairie cleanepi fournit un exemple de dictionnaire intégré spécialement conçu pour les données épidémiologiques. Le code ci-dessous montre les correspondances qui y sont établies pour la colonne “gender”.
R
test_dict <- base::readRDS(
system.file("extdata", "test_dict.RDS", package = "cleanepi")
) %>%
dplyr::as_tibble()
test_dict
SORTIE
# A tibble: 6 × 4
options values grp orders
<chr> <chr> <chr> <int>
1 1 male gender 1
2 2 female gender 2
3 M male gender 3
4 F female gender 4
5 m male gender 5
6 f female gender 6Nous pouvons maintenant utiliser ce dictionnaire pour substituer les
valeurs de la colonne “gender” selon des catégories prédéfinies. Vous
trouverez ci-dessous un exemple de code démontrant comment utiliser la
fonction clean_using_dictionary() de la librairie
{cleanepi} allant dans ce sens.
R
sim_ebola_data <- cleanepi::clean_using_dictionary(
data = sim_ebola_data,
dictionary = test_dict
)
sim_ebola_data
SORTIE
# A tibble: 15,000 × 8
v1 case_id age gender status date_onset date_sample row_id
<int> <chr> <dbl> <chr> <chr> <date> <date> <int>
1 1 14905 90 male confirmed 2015-03-15 2015-06-04 1
2 2 13043 25 female <NA> 2013-09-11 2014-03-01 2
3 3 14364 54 female <NA> 2014-09-02 2015-03-03 3
4 4 14675 90 <NA> <NA> 2014-10-19 2031-12-14 4
5 5 12648 74 female <NA> 2014-08-06 2016-10-10 5
6 6 14274 76 female <NA> 2015-04-05 2016-01-23 7
7 7 14132 16 male confirmed NA 2015-05-10 8
8 8 14715 44 female confirmed NA 2016-04-24 9
9 9 13435 26 male <NA> 2014-09-07 2020-09-14 10
10 10 14816 30 female <NA> 2015-06-29 2015-06-02 11
# ℹ 14,990 more rowsCette approche simplifie le processus de nettoyage des données, en garantissant que les variables catégorielles dans les jeux de données épidémiologiques sont catégorisées avec précision et prêtes pour une analyse plus ultérieures.
Calcul de l’intervalle de temps entre différentes dates
Durant l’analyse de données épidémiologiques, il est utile de suivre et d’analyser les événements se produisant au cours du temps, tels que la progression d’une épidémie (c’est-à-dire le temps écoulé entre la date de signalement du premier cas et aujourd’hui) ou la durée entre la durée entre la date de collection et d’analyse des échantillons (c’est-à-dire le temps écoulé entre la date de collecte des échantillons et aujourd’hui). L’exemple le plus courant est le calcul de l’âge des individus à partir de leur date de naissance (c’est-à-dire la différence de temps entre aujourd’hui et leurs dates de naissance).
La librairie cleanepi dispose d’une fonction
permettant de calculer le temps écoulé entre deux événements datés à
différentes échelles de temps. Par exemple, l’extrait de code ci-dessous
utilise la fonction cleanepi::timespan() pour calculer le
temps écoulé depuis la date à laquelle les échantillons ont été
prélevées jusqu’au 3 janvier 2025 ("2025-01-03").
R
sim_ebola_data <- cleanepi::timespan(
data = sim_ebola_data,
target_column = "date_sample",
end_date = as.Date("2025-01-03"),
span_unit = "years",
span_column_name = "years_since_collection",
span_remainder_unit = "months"
)
sim_ebola_data %>%
dplyr::select(case_id, date_sample, years_since_collection, remainder_months)
SORTIE
# A tibble: 15,000 × 4
case_id date_sample years_since_collection remainder_months
<chr> <date> <dbl> <dbl>
1 14905 2015-06-04 9 7
2 13043 2014-03-01 10 10
3 14364 2015-03-03 9 10
4 14675 2031-12-14 -6 -11
5 12648 2016-10-10 8 2
6 14274 2016-01-23 8 11
7 14132 2015-05-10 9 7
8 14715 2016-04-24 8 8
9 13435 2020-09-14 4 3
10 14816 2015-06-02 9 7
# ℹ 14,990 more rowsAprès avoir exécuté la fonction cleanepi::timespan(),
deux nouvelles colonnes nommées years_since_collection et
remainder_months ont été ajoutées à la base de données
sim_ebola_data. Ces colonnes contiennent respectivement
le temps écoulé depuis la date de prélèvement de l’échantillon de chaque
individu, mesuré en années, et le temps restant mesuré en mois.
Défi
Les données relatives à l’âge sont utiles dans beaucoup d’analyses qui seront menées après le nettoyage des données. Vous pouvez classer les âges en catégories pour générer des estimations stratifiées.
Utilisez le jeu de données test_df.RDS qui se trouve
dans la librairie cleanepi pour calculez l’âge en années
jusqu’au 1er mars à partir de la date de naissance des
sujets, et le temps restant en mois. Nettoyez et standardisez les
éléments nécessaires pour y parvenir.
R
# lire les donnees dans test_df.RDS
dat <- readRDS(
file = system.file("extdata", "test_df.RDS", package = "cleanepi")
) %>%
dplyr::as_tibble()
Avant de calculer l’âge, vous aurez besoin de:
- normaliser les noms des colonnes
- standardiser les colonnes de type date
- remplacer les valeurs manquantes par ‘NA’
Dans la solution, nous avons ajouter
date_first_pcr_positive_test parmi les colonnes de type
date à standardiser étant donné que c’est une variable qui est souvent
utilisée durant les analyse épidémiologiques.
R
dat_clean <- dat %>%
# normaliser les noms de colonnes et les colonnes de type date
cleanepi::standardize_column_names() %>%
cleanepi::standardize_dates(
target_columns = c("date_of_birth", "date_first_pcr_positive_test")
) %>%
# replacer les chaînes de caractères représentant les données
# manquantes avec NA
cleanepi::replace_missing_values(
target_columns = c("sex", "date_of_birth"),
na_strings = "-99"
) %>%
# calculer l'age en 'annees' and retourner le reste en 'mois'
cleanepi::timespan(
target_column = "date_of_birth",
end_date = as.Date("2025-03-01"),
span_unit = "years",
span_column_name = "age_in_years",
span_remainder_unit = "months"
)
SORTIE
! Found <numeric> values that could also be of type <Date> in column:
date_of_birth.
ℹ It is possible to convert them into <Date> using: `lubridate::as_date(x,
origin = as.Date("1900-01-01"))`
• where "x" represents here the vector of values from these columns
(`data$target_column`).Maintenant, comment définiriez-vous les classes d’une variable numérique ?
La solution la plus simple consiste à utiliser
Hmisc::cut2(). Vous pouvez également utiliser
dplyr::case_when(). Cependant cette solution nécessite plus
de lignes de code et est plus appropriée pour les catégorisations
personnalisées. Nous vous proposons ici une solution basée sur
base::cut():
R
dat_clean %>%
# selectionner les colonnes d'intéret
dplyr::select(
study_id,
sex,
date_first_pcr_positive_test,
date_of_birth,
age_in_years
) %>%
# categoriser la variable age [ajouter comme indice dans le challenge]
# replacer avec la valeur maximale si elle est connue
dplyr::mutate(
age_category = base::cut(
x = age_in_years,
breaks = c(0, 20, 35, 60, Inf),
include.lowest = TRUE,
right = FALSE
)
)
SORTIE
# A tibble: 10 × 6
study_id sex date_first_pcr_posit…¹ date_of_birth age_in_years age_category
<chr> <int> <date> <date> <dbl> <fct>
1 PS001P2 1 2020-12-01 1972-06-01 52 [35,60)
2 PS002P2 1 2021-01-01 1952-02-20 73 [60,Inf]
3 PS004P2… NA 2021-02-11 1961-06-15 63 [60,Inf]
4 PS003P2 1 2021-02-01 1947-11-11 77 [60,Inf]
5 P0005P2 2 2021-02-16 2000-09-26 24 [20,35)
6 PS006P2 2 2021-05-02 NA NA <NA>
7 PB500P2 1 2021-02-19 1989-11-03 35 [35,60)
8 PS008P2 2 2021-09-20 1976-10-05 48 [35,60)
9 PS010P2 1 2021-02-26 1991-09-23 33 [20,35)
10 PS011P2 2 2021-03-03 1991-02-08 34 [20,35)
# ℹ abbreviated name: ¹date_first_pcr_positive_testVous pouvez rechercher les valeurs maximales des variables à travers
le résumé obtenu en utilisant la fonction skimr::skim(). Au
lieu de base::cut() vous pouvez également utiliser
Hmisc::cut2(x = age_in_years,cuts = c(20,35,60)) qui
calcule la valeur maximale et ne nécessite pas plus d’arguments.
Plusieurs opérations à la fois
L’exécution individuelle des opérations de nettoyage des données peut
prendre beaucoup de temps et être source d’erreurs. La fonction
clean_data() de la librairie cleanepi permet
de simplifier ce processus en permettant d’exécuter plusieurs opérations
à la fois.
Lorsque l’utilisateur ne spécifie aucune opération de nettoyage de
données, la fonction applique automatiquement une série d’opérations de
nettoyage de données. Voici un exemple de code illustrant l’utilisation
de la fonction clean_data() sur le jeu de données simulé
d’Ebola:
R
cleaned_data <- cleanepi::clean_data(raw_ebola_data)
SORTIE
ℹ Cleaning column namesSORTIE
ℹ Removing constant columns and empty rowsSORTIE
ℹ Removing duplicated rowsSORTIE
! Found 5 duplicated rows in the dataset.
ℹ Use `attr(dat, "report")[["duplicated_rows"]]` to access them, where "dat" is
the object used to store the output from this operation.En outre, vous pouvez combiner plusieurs opérations de nettoyage de
données via l’opérateur pipe (|>) de la librairie de
base de R ou celui de la librairie {magrittr} (%>%),
comme illustré à travers le code ci-dessous.
R
# Exécuter les opérations de nettoyage de données en utilisant l'opérateur %>%
cleaned_data <- raw_ebola_data %>%
cleanepi::standardize_column_names() %>%
cleanepi::remove_constants() %>%
cleanepi::remove_duplicates() %>%
cleanepi::replace_missing_values(na_strings = "") %>%
cleanepi::check_subject_ids(
target_columns = "case_id",
range = c(1, 15000)
) %>%
cleanepi::standardize_dates(
target_columns = c("date_onset", "date_sample")
) %>%
cleanepi::convert_to_numeric(target_columns = "age") %>%
cleanepi::check_date_sequence(
target_columns = c("date_onset", "date_sample")
) %>%
cleanepi::clean_using_dictionary(dictionary = test_dict) %>%
cleanepi::timespan(
target_column = "date_sample",
end_date = as.Date("2025-01-03"),
span_unit = "years",
span_column_name = "years_since_collection",
span_remainder_unit = "months"
)
Défi
Avez-vous remarqué que cleanepi contient un ensemble de fonctions pour diagnostiquer l’état de propreté des données et un autre ensemble pour effectuer le nettoyage ?
Identifier les deux groupes :
- Sur une feuille de papier, écrivez le nom de chaque fonction dans la colonne correspondante :
| Diagnostiquer la propreté des données | Effectuer le nettoyage des données |
|---|---|
| … | … |
Notez que cleanepi contient un ensemble de fonctions
pour diagnostiquer l’état de propreté des données (par
exemple check_subject_ids() et
check_date_sequence() dans l’extrait de code ci-dessus) et
un autre pour effecture les opérations de nettoyage (le
reste des fonctions dans l’exemple ci-dessus).
Rapport du nettoyage des données
cleanepi génère un rapport complet détaillant les résultats et les actions de tous les fonctions ayant servi au nettoyages de données. Ce rapport se présente sous la forme d’un fichier HTML qui s’ouvrira automatiquement dans votre navigateur par défaut. Il comporte plusieurs sections. Chaque section correspond à une opération spécifique de nettoyage des données, et un clic sur chaque section vous permet d’accéder aux résultats de l’opération de nettoyage des données en question. Cette approche interactive permet aux utilisateurs d’examiner et d’analyser efficacement les effets des opérations de nettoyage des données.
Vous pouvez consulter le rapport de nettage de données à l’aide de la
fonction cleanepi::print_report(cleaned_data).
Exemple de rapport de nettoyage de données généré par
cleanepi
Les rapports de nettoyage de données sont générés par
cleanepi.

Comment cela est-il possible ?
Nous vous invitons à trouver la principale librairie qui rend cette normalisation possible au sein de cleanepi en lisant la section “Détails” du Manuel de référence sur la normalisation des variables de type date!