Content from Lecture des données épidémiologiques
Dernière mise à jour le 2025-11-11 | Modifier cette page
Durée estimée : 30 minutes
Vue d'ensemble
Questions
- Où stockez-vous habituellement vos données épidémiologiques ?
- Quels sont les formats des données que vous utilisez pour vos analyses ?
- Pouvez-vous importer des données à partir de bases de données et de system d’information de santé (SIS) à travers leur API ?
Objectifs
- Expliquez comment importer des données épidémiologiques dans
Rà partir de différentes sources.
Conditions préalables à l’utilisation du logiciel
Cet épisode nécessite que vous soyez familier avec Science des données: Tâches de base avec R.
Introduction
L’étape initiale de l’analyse de données épidémiologiques consiste
généralement à importer le jeu de données d’intérêt dans le logiciel
R à partir d’une source locale (comme un fichier sur votre
ordinateur) ou d’une source externe (comme une base de données). Les
données relatives aux épidémies peuvent être stockées dans des fichiers
sous multiples formats, dans des systèmes de gestion de bases de données
relationnelles (SGBDR) ou dans des systèmes d’information sanitaires
(SIS), tels que SORMAS et DHIS2 qui fournissent une interface de
programme d’application (API) à la base de données du système afin que
les utilisateurs vérifiés puissent facilement y ajouter et en extraire
des données. Cette dernière option est particulièrement adaptée à la
collecte et au stockage de large données de santé par les institutions.
Cet épisode élucidera le processus de lecture de données à partir de ces
sources.
Commençons par charger la librairie rio pour importer
les données stockées dans des fichiers et la librairie
here pour trouver facilement un chemin d’accès à ces
fichiers dans votre projet RStudio. Nous utiliserons l’opérateur pipe
(%>%) de la librairie magrittr pour
relier facilement certaines de leurs fonctions, y compris les fonctions
de la librairie de formatage de données dplyr. Nous
chargerons donc la librairie tidyverse, qui comprend à la
fois les librairies magrittr et dplyr.
R
# charger les librairies
library(tidyverse)
library(rio)
library(here)
library(readepi)
library(dbplyr)
L’opérateur double deux-points
(::)
L’opérateur :: de R permet d’accéder aux fonctions ou
aux objets d’un package spécifique sans attacher l’intégralité du
package (sans faire appel à la functionlibray()). Il offre
plusieurs avantages, notamment :
- Indiquer explicitement le package d’origine d’une fonction, réduisant ainsi les ambiguïtés et les conflits potentiels lorsque plusieurs packages possèdent des fonctions portant le même nom.
- Permettre d’appeler une fonction depuis un package sans charger
l’intégralité du package avec
library().
Par exemple, la commande dplyr::filter(data, condition)
signifie que nous appelons la fonction filter() depuis la
librairie dplyr.
Créez un projet et un dossier
- Créez un projet RStudio. Si nécessaire, suivez la procédure dans guide pratique sur “Hello RStudio Projects” pour en créer un.
- Dans le projet RStudio, créez un dossier
data/dossier. - Dans le dossier
data/enregistrez les fichiers au format CSV ebola_cases_2.csv et marburg.zip.
Lecture de fichiers
Ils existent plusieurs librairies pour importer des données
épidémiologiques dans R à partir de fichiers individuels.
Il s’agit notamment de {rio}, {readr} de la
tidyverse, {io}, {ImportExport}
et {data.table},
et des fonctions similaires de la librairie de base de R. Ensemble, ces
librairies offrent des méthodes pour lire un ou plusieurs fichiers de
différents formats.
L’exemple ci-dessous montre comment importer un fichier
csv dans R à l’aide de la librairie
rio. Nous avons utilisé la librairie here
pour indiquer à R de rechercher le fichier dans le dossier
data/ de votre projet, et la fonction
dplyr::as_tibble() pour le convertir dans un format plus
ordonné en vue d’une analyse ultérieure dans R.
R
# lire les données
# exemple:
# si le chemin d'accès au fichier est "data/raw-data/ebola_cases_2.csv",
# alors:
ebola_confirmed <- rio::import(
here::here("data", "ebola_cases_2.csv")
) %>%
dplyr::as_tibble()
# obtenir un aperçu des données
ebola_confirmed
SORTIE
# A tibble: 120 × 4
year month day confirm
<int> <int> <int> <int>
1 2014 5 18 1
2 2014 5 20 2
3 2014 5 21 4
4 2014 5 22 6
5 2014 5 23 1
6 2014 5 24 2
7 2014 5 26 10
8 2014 5 27 8
9 2014 5 28 2
10 2014 5 29 12
# ℹ 110 more rowsVous pouvez aussi importer des fichiers d’autres formats tels que
tsv, xlsx, etc en utilisant la même
fonction.
Pourquoi devrions-nous utiliser la librairie {here}
La librairie here est conçue pour simplifier le référencement des fichiers dans un projet R en fournissant un moyen fiable de construire les chemins d’accès aux fichiers par rapport à la racine du projet. La principale raison de l’utiliser est de s’assurer la Compatibilité entre environnements.
Elle fonctionne à travers différents systèmes d’exploitation (Windows, Mac, Linux) sans qu’il soit nécessaire d’ajuster les chemins d’accès aux fichiers.
- Sous Windows, les chemins d’accès sont écrits en utilisant des
barres obliques inverses (
\) comme séparateur entre les noms de dossiers :"data\raw-data\file.csv" - Sur les systèmes d’exploitation Unix tels que macOS ou Linux, on
utilise la barre oblique (
/) pour séparer les noms de dossiers :"data/raw-data/file.csv"
La librairie here permet de renforcer la reproductibilité de votre travail à travers différents systèmes d’exploitation. Si vous êtes intéressé par la reproductibilité, nous vous invitons à lire ce tutoriel pour accroître l’accès, la durabilité et la reproductibilité de vos analyses épidémiques avec R.
Lecture de données compressées
Pouvez-vous importer les données d’un fichier compressé dans
R?
Téléchargez le fichier compressé contenant les données relatives à l’épidémie de Marburg et importez-le dans votre environnement de travail.
Vous pouvez consulter la liste complète des formats de fichiers pris en charge sur le site web de la librairie rio. Pour étendre {rio} à d’autres formats, vous pouvez installer les librairies correspondantes à l’aide de la fonction ci-dessous :
R
rio::install_formats()
R
rio::import(here::here("data", "Marburg.zip"))
Lecture de données à partir d’une bases de données
La librarie readepi contient des fonctionalités qui
permettent d’importer des données directement des SGBDR ou de SIS (à
travers leurs API). La fonction readepi::read_rdbms()
permet d’importer des données à partir de serveurs tels que Microsoft
SQL, MySQL, PostgreSQL, et SQLite. Elle repose essentiellement sur la
librairie {DBI} qui sert
d’interface polyvalente pour interagir avec les systèmes de gestion de
bases de données relationnelles (SGBDR).
Avantages liées à l’importation des données à partir d’une base de données ?
La lecture de données directement à partir d’une base de données permet d’optimiser la quantité de mémoire utilisée par la session R. Si nous envoyons une requête à la base de données avec des instructions de filtrage des données (par exemple, select, filter, summarise) avant d’extraire les données, nous pouvons réduire la charge de mémoire dans notre session RStudio. À l’inverse, en effectuant toutes les manipulations de données en dehors du système de gestion de base de données après lecture de toute la base de données dans R peut entrainer l’utilisation de beaucoup plus de mémoire (RAM) que possible sur un ordinateur, pouvant entraîner un ralentissement, voire un blocage de RStudio.
Les systèmes de gestion de bases de données relationnelles (SGBDR) présentent également l’avantage de permettre à plusieurs utilisateurs d’accéder, de stocker et d’analyser simultanément différentes parties de l’ensemble de données, sans devoir transférer des fichiers individuels, ce qui rendrait très difficile le suivi de la version la plus à jour.
1. Etablir la connection à une base de données
Vous pouvez utiliser la fonction readepi::login() pour
établir une connection avec la base de données comme illuster
ci-dessous.
R
# etablir la connection avec la base de donnees
rdbms_login <- readepi::login(
from = "mysql-rfam-public.ebi.ac.uk",
type = "MySQL",
user_name = "rfamro",
password = "",
driver_name = "",
db_name = "Rfam",
port = 4497
)
SORTIE
✔ Logged in successfully!R
rdbms_login
SORTIE
<Pool> of MySQLConnection objects
Objects checked out: 0
Available in pool: 1
Max size: Inf
Valid: TRUEÀ noter
Dans cet exemple, l’accès peut être limité par des restrictions réseau organisationnelles, mais il devrait fonctionner normalement sur les réseaux domestiques
2. Accéder á la liste des tables de la base de données
Vous pouvez accéder à la liste des noms de tables qui existent dans
une base de données en utilisant la function
readepi::show_tables().
R
# obtenir la liste des tables dans la base de donnees
tables <- readepi::show_tables(login = rdbms_login)
tables
Dans un cadre de base de données, vous pouvez avoir plusieurs
tables.
Chaque table peut correspondre à une entité spécifique (par
ex. patients, unités de soins, emplois).
Toutes les tables sont liées par un identifiant commun ou une
clé primaire.
3. Lire les données d’une table dans une base de données
La fonction readepi::read_rdbms() permet d’importer les
données à travers une requête SQL ou une liste de paramètres.
R
# lire les donnees de la table 'author' en utilisant une requete SQL
dat <- readepi::read_rdbms(
login = rdbms_login,
query = "select * from author"
)
# lire les donnees de la table 'author' en utilisant une liste de parametres
dat <- readepi::read_rdbms(
login = rdbms_login,
query = list(table = "author", fields = NULL, filter = NULL)
)
Alternativement, nous pouvons lire les données de la table
author en utilisant dplyr::tbl().
R
# lire les donnees de la table 'author' en utilisant une requete SQL
dat <- rdbms_login %>%
dplyr::tbl(from = "author") %>%
dplyr::filter(initials == "A") %>%
dplyr::arrange(desc(author_id))
dat
SORTIE
# Source: SQL [?? x 6]
# Database: mysql 8.0.32-24 [@mysql-rfam-public.ebi.ac.uk:/Rfam]
# Ordered by: desc(author_id)
author_id name last_name initials orcid synonyms
<int> <chr> <chr> <chr> <chr> <chr>
1 46 Roth A Roth A "" ""
2 42 Nahvi A Nahvi A "" ""
3 32 Machado Lima A Machado Lima A "" ""
4 31 Levy A Levy A "" ""
5 27 Gruber A Gruber A "0000-0003-1219-4239" ""
6 13 Chen A Chen A "" ""
7 6 Bateman A Bateman A "0000-0002-6982-4660" "" Si nous appliquons dplyr à cette base de données SQLite, ces verbes seront traduits en requêtes SQL.
R
dat %>%
dplyr::show_query()
SORTIE
<SQL>
SELECT `author`.*
FROM `author`
WHERE (`initials` = 'A')
ORDER BY `author_id` DESC4. Extraire des données de la base de données
Utiliser dplyr::collect() pour forcer le calcul d’une
requête de base de données et extraire la sortie sur votre ordinateur
local.
R
dat %>%
dplyr::collect()
SORTIE
# A tibble: 7 × 6
author_id name last_name initials orcid synonyms
<int> <chr> <chr> <chr> <chr> <chr>
1 46 Roth A Roth A "" ""
2 42 Nahvi A Nahvi A "" ""
3 32 Machado Lima A Machado Lima A "" ""
4 31 Levy A Levy A "" ""
5 27 Gruber A Gruber A "0000-0003-1219-4239" ""
6 13 Chen A Chen A "" ""
7 6 Bateman A Bateman A "0000-0002-6982-4660" "" Idéalement, après avoir spécifié un ensemble de requêtes, nous pouvons réduire la taille du jeu de données d’entrée à utiliser dans l’environnement de notre session R.
Exécutez des requêtes SQL dans R à l’aide de dbplyr
Entraînez-vous à faire des requêtes SQL sur des bases de données
relationnelles en utilisant plusieurs logiciels. Les verbes
dplyr comme dplyr::left_join() peuvent être
appliqués entre les tables avant d’extraire les données vers votre
session locale avec dplyr::collect() !
Vous pouvez également consulter le package dbplyr en R. Pour un tutoriel pas à pas sur SQL, nous recommandons le tutoriel sur la gestion des données avec SQL pour écologistes, qui montre l’utilisation de dplyr avec SQL.
R
# SELECT FEW COLUMNS FROM ONE TABLE AND LEFT JOIN WITH ANOTHER TABLE
author <- rdbms_login %>%
dplyr::tbl(from = "author") %>%
dplyr::select(author_id, name)
family_author <- rdbms_login %>%
dplyr::tbl(from = "family_author") %>%
dplyr::select(author_id, rfam_acc)
dplyr::left_join(author, family_author, keep = TRUE) %>%
dplyr::show_query()
SORTIE
Joining with `by = join_by(author_id)`SORTIE
<SQL>
SELECT
`author`.`author_id` AS `author_id.x`,
`name`,
`family_author`.`author_id` AS `author_id.y`,
`rfam_acc`
FROM `author`
LEFT JOIN `family_author`
ON (`author`.`author_id` = `family_author`.`author_id`)R
dplyr::left_join(author, family_author, keep = TRUE) %>%
dplyr::collect()
SORTIE
Joining with `by = join_by(author_id)`SORTIE
# A tibble: 4,874 × 4
author_id.x name author_id.y rfam_acc
<int> <chr> <int> <chr>
1 1 Ames T 1 RF01831
2 2 Argasinska J 2 RF02554
3 2 Argasinska J 2 RF02555
4 2 Argasinska J 2 RF02722
5 2 Argasinska J 2 RF02720
6 2 Argasinska J 2 RF02719
7 2 Argasinska J 2 RF02721
8 2 Argasinska J 2 RF02670
9 2 Argasinska J 2 RF02718
10 2 Argasinska J 2 RF02668
# ℹ 4,864 more rowsLecture de données à partir de SIS
Les données relatives à la santé sont de plus en plus souvent stockées dans des SIS spécialisées telles que Fingertips, GoData, REDCap, DHIS2, SORMAS, etc. La version actuelle de la librairie readepi permet d’importer des données à partir de DHIS2 et SORMAS.
Lecture des données à partir de DHIS2
Le système d’information sanitaire de district DHIS2 est un logiciel open source
qui a révolutionné la gestion des informations sanitaires mondiales. La
fonction readepi::read_dhis2() permet d’importer des
données depuis le system Tracker de DHIS2 via
leurs API.
Pour cela, il faudra établir la connection au système en utilisant la
fonction readepi::login(), puis fornir le nom ou
l’identifiant du programme et de l’unité organisationnelle (structure
sanitaire, localité, pays, etc) cible.
Pour un système donné, vous pouvez accéder aux identifiants et noms
des programmes et structure sanitaires en utilisant les fonctions
get_programs() et get_organisation_units()
respectivement.
R
# etablir la connection au systeme
dhis2_login <- readepi::login(
from = "https://smc.moh.gm/dhis",
user_name = "test",
password = "Gambia@123"
)
SORTIE
✔ Logged in successfully!R
# obtenir les noms et identifiants des programmes
programs <- readepi::get_programs(login = dhis2_login)
# obtenir les noms et identifiants des unites organisationnelles
org_units <- get_organisation_units(login = dhis2_login)
R
# importer les donnees a partir de DHIS2 en utilisant les identifiants
data <- readepi::read_dhis2(
login = dhis2_login,
org_unit = "GcLhRNAFppR",
program = "E5IUQuHg3Mg"
)
# importer les donnees a partir de DHIS2 en utilisant les noms
data <- readepi::read_dhis2(
login = dhis2_login,
org_unit = "Keneba",
program = "Child Registration & Treatment "
)
tibble::as_tibble(data)
SORTIE
# A tibble: 1,116 × 69
event tracked_entity org_unit ` SMC-CR Scan QR Code` SMC-CR Did the child…¹
<chr> <chr> <chr> <chr> <chr>
1 bgSDQb… yv7MOkGD23q Keneba SMC23-0510989 1
2 y4MKmP… nibnZ8h0Nse Keneba SMC2021-018089 1
3 yK7VG3… nibnZ8h0Nse Keneba SMC2021-018089 1
4 EmNflz… nibnZ8h0Nse Keneba SMC2021-018089 1
5 UF96ms… nibnZ8h0Nse Keneba SMC2021-018089 1
6 guQTwc… FomREQ2it4n Keneba SMC23-0510012 1
7 jbkRkL… FomREQ2it4n Keneba SMC23-0510012 1
8 AEeype… FomREQ2it4n Keneba SMC23-0510012 1
9 R30SPs… E5oAWGcdFT4 Keneba koika-smc-22897 1
10 nr03Qy… E5oAWGcdFT4 Keneba koika-smc-22897 1
# ℹ 1,106 more rows
# ℹ abbreviated name: ¹`SMC-CR Did the child previously received a card?`
# ℹ 64 more variables: `SMC-CR Child First Name1` <chr>,
# `SMC-CR Child Last Name` <chr>, `SMC-CR Date of Birth` <chr>,
# `SMC-CR Select Age Category ` <chr>, `SMC-CR Child gender1` <chr>,
# `SMC-CR Mother/Person responsible full name` <chr>,
# `SMC-CR Mother/Person responsible phone number1` <chr>, …Il est important de savoir que toutes les unités organisationnelles
(structures sanitaires) ne sont pas enregistrées pour un programme
spécifique. Pour connaître les unités organisationnelles qui exécutent
un programme particulier, utilisez la fonction
get_program_org_units() comme illustré dans l’exemple
ci-dessous.
R
# obtenir les unités organisationnelles qui exécutent un programme "E5IUQuHg3Mg"
target_org_units <- readepi::get_program_org_units(
login = dhis2_login,
program = "E5IUQuHg3Mg",
org_units = org_units
)
tibble::as_tibble(target_org_units)
SORTIE
# A tibble: 26 × 3
org_unit_ids levels org_unit_names
<chr> <chr> <chr>
1 UrLrbEiWk3J Town/Village_name Sare Sibo
2 wlVsFVeHSTx Town/Village_name Jawo Kunda
3 kp0ZYUEqJE8 Town/Village_name Chewal
4 Wr3htgGxhBv Town/Village_name Madinayel
5 psyHoqeN2Tw Town/Village_name Bolibanna
6 MGBYonFM4y3 Town/Village_name Sare Mala
7 GcLhRNAFppR Town/Village_name Keneba
8 y1Z3KuvQyhI Town/Village_name Brikama
9 W3vH9yBUSei Town/Village_name Gidda
10 ISbNWYieHY8 Town/Village_name Song Kunda
# ℹ 16 more rowsLecture des données à partir de SORMAS
Le Système de surveillance, de gestion et d’analyse des réponses aux
épidémies SORMAS est un système de
santé digital open source qui optimise les processus de surveillance de
la propagation des maladies infectieuses et de réponse aux épidémies. La
fonction readepi::read_sormas() permet d’importer des
données depuis SORMAS via son API.
Dans la version actuelle de la librairie readepi, la
fonction read_sormas() renvoie des données pour les
colonnes suivantes: case_id, person_id, sex, date_of_birth,
case_origin, country, city, lat, long, case_status, date_onset,
date_admission, date_last_contact, date_first_contact, outcome,
date_outcome, Ct_values.
Un des arguments fondamentals est le nom de la maladie pour laquelle
l’utilisateur souhaite obtenir des données. Pour vous assurez de la
vraie synthaxe à utiliser lors de l’appel à la fonction, vous pouvez
obtenir la liste des noms de maladies à travers la fonction
sormas_get_diseases().
R
# obtenir la liste des noms de maladies
disease_names <- readepi::sormas_get_diseases(
base_url = "https://demo.sormas.org/sormas-rest",
user_name = "SurvSup",
password = "Lk5R7JXeZSEc"
)
tibble::as_tibble(disease_names)
SORTIE
# A tibble: 65 × 2
disease active
<chr> <chr>
1 AFP TRUE
2 CHOLERA TRUE
3 CONGENITAL_RUBELLA TRUE
4 CSM TRUE
5 DENGUE TRUE
6 EVD TRUE
7 GUINEA_WORM TRUE
8 LASSA TRUE
9 MEASLES TRUE
10 MONKEYPOX TRUE
# ℹ 55 more rowsR
# obtenir les donnees de tous les cas de COVID-19
covid_cases <- readepi::read_sormas(
base_url = "https://demo.sormas.org/sormas-rest",
user_name = "SurvSup",
password = "Lk5R7JXeZSEc",
disease = "coronavirus"
)
tibble::as_tibble(covid_cases)
SORTIE
# A tibble: 5 × 16
case_id person_id date_onset date_admission case_origin case_status outcome
<chr> <chr> <date> <date> <chr> <chr> <chr>
1 SZ3GHH-RJ… V2XMXK-K… NA NA IN_COUNTRY NOT_CLASSI… NO_OUT…
2 W5C6VE-OH… SBWO4N-3… NA NA IN_COUNTRY NOT_CLASSI… NO_OUT…
3 XBXV3A-TI… QXQ5VA-2… 2025-09-14 2025-09-14 IN_COUNTRY CONFIRMED NO_OUT…
4 SSTIVB-VS… ROTW6C-D… 2025-10-14 NA IN_COUNTRY NO_CASE NO_OUT…
5 T6ZLGJ-MU… WON54L-6… NA NA IN_COUNTRY NOT_CLASSI… NO_OUT…
# ℹ 9 more variables: sex <chr>, date_of_birth <chr>, country <chr>,
# city <chr>, latitude <chr>, longitude <chr>, contact_id <chr>,
# date_last_contact <date>, Ct_values <chr>Content from Nettoyage des données épidémiologiques
Dernière mise à jour le 2025-11-11 | Modifier cette page
Durée estimée : 30 minutes
Vue d'ensemble
Questions
- Comment nettoyer et standardiser les données épidémiologiques ?
Objectifs
- Expliquez comment nettoyer, organiser et standardiser les données épidémioloques en utilisant la librairie cleanepi
- Appliquer les opérations de nettoyage de données sur des données épidémiologiques simulées
Pré-requis
Pour cet épisode, nous utiliserons les données simulées portant sur des individus atteints de la fièvre hémorargique ébola que vous devez:
- d’abord télécharger à partir d’ici,
- puis l’enregistrez-le dans le répertoire
data/.
Introduction
Lors de l’analyse de données épidémiologiques, il est essentiel de s’assurer que les des données sont nettoyées, organisées, standardisées et validées pour garantir la précision (vous analysez ce que vous pensez analyser) et la reproductibilité (si quelqu’un veut revenir en arrière et répéter vos étapes d’analyse avec votre code, vous pouvez être sûr qu’il obtiendra les mêmes résultats) de l’analyse. Cet épisode est consacré au nettoyage de données relatives aux épidémies à l’aide du package R {cleanepi}. Pour illustrer l’utilisation des fonctionalités de ce package, nous les appliquerons sur des données simulées de cas d’Ebola.
En plus du package {cleanepi}, nous utiliserons d’autres fonctions qui proviennent des packages R suivants durant cette procédure de nettoyage des données :
- here pour faciliter le référencement des fichiers,
- rio pour importer les données dans R,
- dplyr pour effectuer certaines opérations de traitement de données,
-
magrittr pour utiliser son opérateur pipe
(
%>%).
R
# charger les librairies
library(tidyverse)
library(rio)
library(here)
library(cleanepi)
L’opérateur double deux-points
(::)
L’opérateur :: de R permet d’accéder aux fonctions ou
aux objets d’un package spécifique sans attacher l’intégralité du
package (sans faire appel à la functionlibray()). Il offre
plusieurs avantages, notamment :
- Indiquer explicitement le package d’origine d’une fonction, réduisant ainsi les ambiguïtés et les conflits potentiels lorsque plusieurs packages possèdent des fonctions portant le même nom.
- Permettre d’appeler une fonction depuis un package sans charger
l’intégralité du package avec
library().
Par exemple, la commande dplyr::filter(data, condition)
signifie que nous appelons la fonction filter() depuis la
librairie dplyr.
La première étape consiste à importer le jeu de données dans
l’environnement de travail. Pour ce faire, nous suivrons les
instructions décrites dans l’épisode Importer
les données de cas épidémiologiques pour importer les données dans
R et visualiser sa structure et son contenu.
R
# Importer les données
# supposons que le chemin d'accès au ficher est: data/simulated_ebola_2.csv
raw_ebola_data <- rio::import(
here::here("data", "simulated_ebola_2.csv")
) %>%
dplyr::as_tibble() # convertir en tibble
R
# Afficher les dix premières lignes du data frame
raw_ebola_data
SORTIE
# A tibble: 15,003 × 9
V1 `case id` age gender status `date onset` `date sample` lab region
<int> <int> <chr> <chr> <chr> <chr> <chr> <lgl> <chr>
1 1 14905 90 1 "conf… 03/15/2015 06/04/2015 NA valdr…
2 2 13043 twenty… 2 "" Sep /11/13 03/01/2014 NA valdr…
3 3 14364 54 f <NA> 09/02/2014 03/03/2015 NA valdr…
4 4 14675 ninety <NA> "" 10/19/2014 31/ 12 /14 NA valdr…
5 5 12648 74 F "" 08/06/2014 10/10/2016 NA valdr…
6 5 12648 74 F "" 08/06/2014 10/10/2016 NA valdr…
7 6 14274 sevent… female "" Apr /05/15 01/23/2016 NA valdr…
8 7 14132 sixteen male "conf… Dec /29/Y 05/10/2015 NA valdr…
9 8 14715 44 f "conf… Apr /06/Y 04/24/2016 NA valdr…
10 9 13435 26 1 "" 09/07/2014 20/ 09 /14 NA valdr…
# ℹ 14,993 more rowsDiscussion
Jettons un coup d’oeil sur ce jeu de données pour dressez la liste de toutes les anormalies qui pourraient poser problème durant l’analyse des données.
Certaines de ces anormalies vous sont-elles familières dans le cadre d’une analyse de données que vous avez déjà effectuée ?
Animez une brève discussion pour établir un lien entre les anormalies détectées et les opérations de nettoyage requises.
Vous pouvez utiliser les termes suivants pour désigner les anormalies qui ont été identifiées:
- Codification: codification des valeurs dans certaines colonnes, telles que ‘gender’, ‘age’, à l’aide de chiffres, de lettres et de mots. Également l’existence de plusieur formats de dates ((“jj/mm/aaaa”, “aaaa/mm/jj”, etc)) dans une même colonne telle que ‘date_onset’. Moins visibles, les noms des colonnes.
- Manquant: comment interpréter une entrée telle que “” dans la colonne ‘statut’ ou “-99” dans d’autres circonstances ? Disposons-nous d’un dictionaire de données provenant du processus de collecte des données ?
- Incohérences: avoir des incohérences comme lorsque la date d’échantillonnage est antérieure à la date d’apparition des symptomes.
- Valeurs non plausibles Les valeurs non plausibles, comme les observations aberrantes avec des dates en dehors de la période concernée.
- Les doublons Toutes les observations sont-elles uniques ?
Vous pouvez utiliser ces termes pour vous référer aux opérations de nettoyage:
- Normaliser le nom des colonnes
- Normaliser les variables catégorielles comme ‘gender’
- Normaliser les colonnes de type date
- Convertir des caractères en valeurs numériques
- Vérifier la séquence d’événements datés
Une inspection rapide
Une exploration et une inspection rapides des données sont
essentielles pour identifier les problèmes potentiels avant d’entamer le
processus d’analyse des données. Le package {cleanepi} simplifie ce
processus grâce à la fonction scan_data(). Voyons comment
vous pouvez l’utiliser.
R
cleanepi::scan_data(raw_ebola_data)
SORTIE
Field_names missing numeric date character logical
1 age 0.0646 0.8348 0.0000 0.1006 0
2 gender 0.1578 0.0472 0.0000 0.7950 0
3 status 0.0535 0.0000 0.0000 0.9465 0
4 date onset 0.0001 0.0000 0.9159 0.0840 0
5 date sample 0.0001 0.0000 0.9999 0.0000 0
6 region 0.0000 0.0000 0.0000 1.0000 0Le résultat donne un aperçu du contenu de toutes les colonnes de type charactère, y compris la structure de ces noms de colonnes et le pourcentage de certains types de données qu’on y retrouve. Vous pouvez constater que les noms des colonnes sont descriptifs mais manquent de cohérence. Certains sont composés de plusieurs mots séparés par des espaces. En outre, certaines colonnes contiennent plus d’un type de données, et il y a des valeurs manquantes dans d’autres.
Opérations courantes
Cette section montre comment effectuer quelques opérations courantes de nettoyage de données à l’aide de la librairie cleanepi.
Normalisation des noms de colonnes
Pour cet exemple de jeu de données, la normalisation des noms de
colonnes consiste généralement à supprimer les espaces et à relier les
mots avec "_". Cette pratique permet de maintenir la
cohérence et la lisibilité du jeu de données. Cependant, la fonction
utilisée pour normaliser les noms de colonnes offre plus d’options.
Entrez ?cleanepi::standardize_column_names dans la console
pour plus de détails.
R
sim_ebola_data <- cleanepi::standardize_column_names(raw_ebola_data)
names(sim_ebola_data)
SORTIE
[1] "v1" "case_id" "age" "gender" "status"
[6] "date_onset" "date_sample" "lab" "region" Si vous souhaitez conserver certains noms de colonnes sans les
soumettre au processus de normalisation, vous pouvez utiliser l’argument
keep de la fonction
cleanepi::standardize_column_names(). Cet argument accepte
un vecteur de noms de colonnes qui doivent rester inchangés.
Défi
Quelles différences pouvez-vous observer dans les noms de colonnes ?
Normalisez les noms des colonnes de l’ensemble de données d’entrée, mais conservez le nom de la première colonne tel quel.
Vous pouvez essayer
cleanepi::standardize_column_names(data = raw_ebola_data, keep = "V1")
Suppression des irrégularités
Les données brutes contiennent souvent des irrégularités telles que
des lignes vides et colonnes vides, ou des colonnes
constant (où toutes les entrées ont la même valeur).
Elles peuvent aussi contenir des doublons. Les fonctions de
cleanepi comme remove_duplicates() et
remove_constants() peuvent être utilisées pour supprimer
ces irrégularités, comme le montre le morceau de code ci-dessous.
R
# Supprimer les données invariables
sim_ebola_data <- cleanepi::remove_constants(sim_ebola_data)
Maintenant, afficher le résultat pour identifier les colonnes constantes que vous avez supprimé.
R
# Supprimer les doublons
sim_ebola_data <- cleanepi::remove_duplicates(sim_ebola_data)
SORTIE
! Found 5 duplicated rows in the dataset.
ℹ Use `attr(dat, "report")[["duplicated_rows"]]` to access them, where "dat" is
the object used to store the output from this operation.Vous pouvez obtenir le nombre et l’emplacement des lignes redondantes
qui ont été identifiées à l’aide de la function
cleanepi::print_report().
R
# les doublons identifiés
doublons <- print_report(sim_ebola_data, "found_duplicates")
doublons
Défi
Dans le jeu de données suivant :
SORTIE
# A tibble: 6 × 5
col1 col2 col3 col4 col5
<dbl> <dbl> <chr> <chr> <date>
1 1 1 a b NA
2 2 3 a b NA
3 NA NA a <NA> NA
4 NA NA a <NA> NA
5 NA NA a <NA> NA
6 NA NA <NA> <NA> NA Quelles sont les colonnes ou les lignes:
- contenant des doublons ?
- vides ?
- contenant des données constantes ?
Les données constantes se réfèrent principalement aux lignes et colonnes vides, mais aussi aux colonnes invariables.
R
cleaned_df <- df %>%
cleanepi::remove_constants() %>%
cleanepi::remove_duplicates()
SORTIE
! Constant data was removed after 2 iterations.
ℹ Enter `attr(dat, "report")[["constant_data"]]` for more information, where
"dat" represents the object used to store the output from
`remove_constants()`.
ℹ No duplicates were found.R
print_report(cleaned_df, "constant_data")
- indices des doublon : aucun
- colonnes vides :
- premiere itération: col5
- seconde itération: aucune
- lignes vides :
- premiere itération: 6
- seconde itération: 3, 4, 5
- colonnes constantes : col3, col4
Faites remarquer aux apprenants qu’ils peuvent obtenir des résultats
différents en variant la valeur de l’argument cutoff.
R
cleaned_df <- df %>%
cleanepi::remove_constants(cutoff = 0.5)
R
print_report(cleaned_df, "constant_data")
Remplacer les valeurs manquantes
Outre les irrégularités, les données brutes peuvent contenir des
valeurs manquantes, qui peuvent être representées par différentes
chaînes de caractères (par ex. "NA", "",
character(0)). Pour garantir une analyse solide, il est
conseillé de remplacer toutes les valeurs manquantes par NA
dans l’ensemble du jeu de données. Vous trouverez ci-dessous un extrait
de code démontrant comment vous pouvez remplacer les valeures
manquantes, représentées par une chaîne vide "", en
utilisant la librairie cleanepi:
R
sim_ebola_data <- cleanepi::replace_missing_values(
data = sim_ebola_data,
na_strings = ""
)
sim_ebola_data
SORTIE
# A tibble: 15,000 × 8
v1 case_id age gender status date_onset date_sample row_id
<int> <int> <chr> <chr> <chr> <chr> <chr> <int>
1 1 14905 90 1 confirmed 03/15/2015 06/04/2015 1
2 2 13043 twenty-five 2 <NA> Sep /11/13 03/01/2014 2
3 3 14364 54 f <NA> 09/02/2014 03/03/2015 3
4 4 14675 ninety <NA> <NA> 10/19/2014 31/ 12 /14 4
5 5 12648 74 F <NA> 08/06/2014 10/10/2016 5
6 6 14274 seventy-six female <NA> Apr /05/15 01/23/2016 7
7 7 14132 sixteen male confirmed Dec /29/Y 05/10/2015 8
8 8 14715 44 f confirmed Apr /06/Y 04/24/2016 9
9 9 13435 26 1 <NA> 09/07/2014 20/ 09 /14 10
10 10 14816 thirty f <NA> 06/29/2015 06/02/2015 11
# ℹ 14,990 more rowsValidation des identifiants des individus
Chaque entrée du jeu de données représente un individu (par exemple,
un patient ou un participant à une étude) et doit pouvoir être
distinguée par un identifiant unique formatée d’une certaine façon. Ces
identifiants peuvent contenir des nombres se situant dans un intervalle
bien déterminé, a préfixe et/ou suffixe, être formatés de façon à
contenir un nombre spécifique de caractères. La librairie
cleanepi propose la fonction
check_subject_ids() permettant de vérifier si les
identifiants des individus sont uniques et s’ils satisfont aux critères
établis par l’utilisateur.
R
# vérifier si les identifiants des individus comportent des nombres
# variant entre 0 et 15000
sim_ebola_data <- cleanepi::check_subject_ids(
data = sim_ebola_data,
target_columns = "case_id",
range = c(0, 15000)
)
SORTIE
! Found 1957 duplicated values in the subject Ids.
ℹ Enter `attr(dat, "report")[["duplicated_rows"]]` to access them, where "dat"
is the object used to store the output from this operation.
ℹ No incorrect subject id was detected.Notez que notre jeu de données simulé contient des doublons dans la colonne ayant les identifiants uniques des individus.
Visualisons un rapport préliminaire avec
cleanepi::print_report(sim_ebola_data). Concentrez-vous sur
l’onglet “Unexpected subject ids” pour identifier les identifiants qui
nécessitent une correction.
Après avoir terminé ce tutoriel, nous vous invitons à explorer le
guide de référence de la function cleanepi::check_subject_ids()
pour identifier la function qui peut remédier à cette situation.
Normalisation des dates
Un jeu de données épidémiques contient généralement des colonnes de type date pour différents événements, tels que la date d’infection, la date d’apparition des symptômes, etc. Ces dates peuvent apparaître sous différents formats, et il est bon de les normaliser afin de bénéficier des puissantes fonctionalités de R conçues pour traiter les valeurs de type date durant l’analyse des données. La librairies cleanepi fournit une fonctionnalité permettant de convertir les colonnes de type date dans les données épidémiologiques au format ISO8601, garantissant la cohérence des différentes colonnes de type dates. Voici comment vous pouvez l’utiliser sur notre jeu de données simulé :
R
sim_ebola_data <- cleanepi::standardize_dates(
sim_ebola_data,
target_columns = c("date_onset", "date_sample")
)
sim_ebola_data
SORTIE
# A tibble: 15,000 × 8
v1 case_id age gender status date_onset date_sample row_id
<int> <chr> <chr> <chr> <chr> <date> <date> <int>
1 1 14905 90 1 confirmed 2015-03-15 2015-06-04 1
2 2 13043 twenty-five 2 <NA> 2013-09-11 2014-03-01 2
3 3 14364 54 f <NA> 2014-09-02 2015-03-03 3
4 4 14675 ninety <NA> <NA> 2014-10-19 2031-12-14 4
5 5 12648 74 F <NA> 2014-08-06 2016-10-10 5
6 6 14274 seventy-six female <NA> 2015-04-05 2016-01-23 7
7 7 14132 sixteen male confirmed NA 2015-05-10 8
8 8 14715 44 f confirmed NA 2016-04-24 9
9 9 13435 26 1 <NA> 2014-09-07 2020-09-14 10
10 10 14816 thirty f <NA> 2015-06-29 2015-06-02 11
# ℹ 14,990 more rowsCette fonction convertit les valeurs dans les colonnes cibles au le format YYYY-mm-dd.
Conversion en valeurs numériques
Dans les données brutes, certaines colonnes peuvent contenir un
mélange de valeurs numériques et de caractères, et vous voudrez souvent
convertir les caractères en numériques. Par exemple, certaines valeurs
de la colonne de ‘age’ de notre jeu de données simulées sont écrites en
mots. Dans cleanepi la fonction
convert_to_numeric() permet d’effectuer la conversion de
ces mots en chiffres, comme illustré ci-dessous.
R
sim_ebola_data <- cleanepi::convert_to_numeric(
data = sim_ebola_data,
target_columns = "age"
)
sim_ebola_data
SORTIE
# A tibble: 15,000 × 8
v1 case_id age gender status date_onset date_sample row_id
<int> <chr> <dbl> <chr> <chr> <date> <date> <int>
1 1 14905 90 1 confirmed 2015-03-15 2015-06-04 1
2 2 13043 25 2 <NA> 2013-09-11 2014-03-01 2
3 3 14364 54 f <NA> 2014-09-02 2015-03-03 3
4 4 14675 90 <NA> <NA> 2014-10-19 2031-12-14 4
5 5 12648 74 F <NA> 2014-08-06 2016-10-10 5
6 6 14274 76 female <NA> 2015-04-05 2016-01-23 7
7 7 14132 16 male confirmed NA 2015-05-10 8
8 8 14715 44 f confirmed NA 2016-04-24 9
9 9 13435 26 1 <NA> 2014-09-07 2020-09-14 10
10 10 14816 30 f <NA> 2015-06-29 2015-06-02 11
# ℹ 14,990 more rowsLangues prise en charge
Il faut savoir que la fonction
cleanepi::convert_to_numeric() fait appel à la fonction
principale de la librairie numberize, qui permet de
convertir, en valeurs entières positives, les mots écrits en anglais,
français et espagnol.
Autres opérations liées à l’épidémiologie
Outre les tâches courantes de nettoyage des données, telles que celles évoquées dans les sections précédentes, la librairie cleanepi offre des fonctionnalités supplémentaires spécialement conçues pour le traitement et l’analyse des données relatives aux épidémies. Cette section couvre certaines de ces tâches spécialisées.
Vérification de la séquence des événements datés
Garantir l’ordre et la séquence correcte des événements datés est
crucial dans l’analyse des données épidémiologiques, en particulier lors
de l’analyse des maladies infectieuses, où la chronologie d’événements
tels que la date d’apparition des symptômes et la date de collecte
d’échantillons est essentielle. La librairie cleanepi
fournit une fonction utile appelée check_date_sequence()
précisément conçu dans ce but.
Voici un exemple de morceau de code démontrant l’utilisation de la
fonction check_date_sequence() dans les 100 premières
lignes de notre ensemble de données Ebola simulé.
R
cleanepi::check_date_sequence(
data = sim_ebola_data[1:100, ],
target_columns = c("date_onset", "date_sample")
)
SORTIE
! Detected 16 incorrect date sequences at lines: "10, 20, 22, 26, 29, 44, 46,
54, 60, 63, 70, 71, 73, 80, 81, 90".
ℹ Enter `attr(dat, "report")[["incorrect_date_sequence"]]` to access them,
where "dat" is the object used to store the output from this operation.Cette fonctionnalité est essentielle pour garantir l’intégrité des données et la précision dans les analyses épidémiologiques, car elle permet d’identifier toute incohérence ou erreur dans l’ordre chronologique des événements, ce qui vous permet d’y remédier de manière appropriée.
Substitution basée sur un dictionnaire
Dans le cadre du prétraitement des données, il est fréquent de rencontrer des scénarios dans lesquels certaines colonnes d’un jeu de données, comme la colonne “gender” dans notre jeu de données Ebola simulé, sont censées avoir des valeurs ou des facteurs spécifiques. Cependant, il est également fréquent que des valeurs inattendues ou erronées apparaissent dans ces colonnes, et doivent être remplacées par des valeurs de valeurs appropriées. La librairie cleanepi offre une fonction qui prend en charge la substitution de valeurs basée sur un dictionnaire données. Ainsi, vous permet de remplacer des valeurs dans des colonnes spécifiques sur la base de correspondances définies dans un dictionnaire de données.
Par ailleurs, la librairie cleanepi fournit un exemple de dictionnaire intégré spécialement conçu pour les données épidémiologiques. Le code ci-dessous montre les correspondances qui y sont établies pour la colonne “gender”.
R
test_dict <- base::readRDS(
system.file("extdata", "test_dict.RDS", package = "cleanepi")
) %>%
dplyr::as_tibble()
test_dict
SORTIE
# A tibble: 6 × 4
options values grp orders
<chr> <chr> <chr> <int>
1 1 male gender 1
2 2 female gender 2
3 M male gender 3
4 F female gender 4
5 m male gender 5
6 f female gender 6Nous pouvons maintenant utiliser ce dictionnaire pour substituer les
valeurs de la colonne “gender” selon des catégories prédéfinies. Vous
trouverez ci-dessous un exemple de code démontrant comment utiliser la
fonction clean_using_dictionary() de la librairie
{cleanepi} allant dans ce sens.
R
sim_ebola_data <- cleanepi::clean_using_dictionary(
data = sim_ebola_data,
dictionary = test_dict
)
sim_ebola_data
SORTIE
# A tibble: 15,000 × 8
v1 case_id age gender status date_onset date_sample row_id
<int> <chr> <dbl> <chr> <chr> <date> <date> <int>
1 1 14905 90 male confirmed 2015-03-15 2015-06-04 1
2 2 13043 25 female <NA> 2013-09-11 2014-03-01 2
3 3 14364 54 female <NA> 2014-09-02 2015-03-03 3
4 4 14675 90 <NA> <NA> 2014-10-19 2031-12-14 4
5 5 12648 74 female <NA> 2014-08-06 2016-10-10 5
6 6 14274 76 female <NA> 2015-04-05 2016-01-23 7
7 7 14132 16 male confirmed NA 2015-05-10 8
8 8 14715 44 female confirmed NA 2016-04-24 9
9 9 13435 26 male <NA> 2014-09-07 2020-09-14 10
10 10 14816 30 female <NA> 2015-06-29 2015-06-02 11
# ℹ 14,990 more rowsCette approche simplifie le processus de nettoyage des données, en garantissant que les variables catégorielles dans les jeux de données épidémiologiques sont catégorisées avec précision et prêtes pour une analyse plus ultérieures.
Calcul de l’intervalle de temps entre différentes dates
Durant l’analyse de données épidémiologiques, il est utile de suivre et d’analyser les événements se produisant au cours du temps, tels que la progression d’une épidémie (c’est-à-dire le temps écoulé entre la date de signalement du premier cas et aujourd’hui) ou la durée entre la durée entre la date de collection et d’analyse des échantillons (c’est-à-dire le temps écoulé entre la date de collecte des échantillons et aujourd’hui). L’exemple le plus courant est le calcul de l’âge des individus à partir de leur date de naissance (c’est-à-dire la différence de temps entre aujourd’hui et leurs dates de naissance).
La librairie cleanepi dispose d’une fonction
permettant de calculer le temps écoulé entre deux événements datés à
différentes échelles de temps. Par exemple, l’extrait de code ci-dessous
utilise la fonction cleanepi::timespan() pour calculer le
temps écoulé depuis la date à laquelle les échantillons ont été
prélevées jusqu’au 3 janvier 2025 ("2025-01-03").
R
sim_ebola_data <- cleanepi::timespan(
data = sim_ebola_data,
target_column = "date_sample",
end_date = as.Date("2025-01-03"),
span_unit = "years",
span_column_name = "years_since_collection",
span_remainder_unit = "months"
)
sim_ebola_data %>%
dplyr::select(case_id, date_sample, years_since_collection, remainder_months)
SORTIE
# A tibble: 15,000 × 4
case_id date_sample years_since_collection remainder_months
<chr> <date> <dbl> <dbl>
1 14905 2015-06-04 9 7
2 13043 2014-03-01 10 10
3 14364 2015-03-03 9 10
4 14675 2031-12-14 -6 -11
5 12648 2016-10-10 8 2
6 14274 2016-01-23 8 11
7 14132 2015-05-10 9 7
8 14715 2016-04-24 8 8
9 13435 2020-09-14 4 3
10 14816 2015-06-02 9 7
# ℹ 14,990 more rowsAprès avoir exécuté la fonction cleanepi::timespan(),
deux nouvelles colonnes nommées years_since_collection et
remainder_months ont été ajoutées à la base de données
sim_ebola_data. Ces colonnes contiennent respectivement
le temps écoulé depuis la date de prélèvement de l’échantillon de chaque
individu, mesuré en années, et le temps restant mesuré en mois.
Défi
Les données relatives à l’âge sont utiles dans beaucoup d’analyses qui seront menées après le nettoyage des données. Vous pouvez classer les âges en catégories pour générer des estimations stratifiées.
Utilisez le jeu de données test_df.RDS qui se trouve
dans la librairie cleanepi pour calculez l’âge en années
jusqu’au 1er mars à partir de la date de naissance des
sujets, et le temps restant en mois. Nettoyez et standardisez les
éléments nécessaires pour y parvenir.
R
# lire les donnees dans test_df.RDS
dat <- readRDS(
file = system.file("extdata", "test_df.RDS", package = "cleanepi")
) %>%
dplyr::as_tibble()
Avant de calculer l’âge, vous aurez besoin de:
- normaliser les noms des colonnes
- standardiser les colonnes de type date
- remplacer les valeurs manquantes par ‘NA’
Dans la solution, nous avons ajouter
date_first_pcr_positive_test parmi les colonnes de type
date à standardiser étant donné que c’est une variable qui est souvent
utilisée durant les analyse épidémiologiques.
R
dat_clean <- dat %>%
# normaliser les noms de colonnes et les colonnes de type date
cleanepi::standardize_column_names() %>%
cleanepi::standardize_dates(
target_columns = c("date_of_birth", "date_first_pcr_positive_test")
) %>%
# replacer les chaînes de caractères représentant les données
# manquantes avec NA
cleanepi::replace_missing_values(
target_columns = c("sex", "date_of_birth"),
na_strings = "-99"
) %>%
# calculer l'age en 'annees' and retourner le reste en 'mois'
cleanepi::timespan(
target_column = "date_of_birth",
end_date = as.Date("2025-03-01"),
span_unit = "years",
span_column_name = "age_in_years",
span_remainder_unit = "months"
)
SORTIE
! Found <numeric> values that could also be of type <Date> in column:
date_of_birth.
ℹ It is possible to convert them into <Date> using: `lubridate::as_date(x,
origin = as.Date("1900-01-01"))`
• where "x" represents here the vector of values from these columns
(`data$target_column`).Maintenant, comment définiriez-vous les classes d’une variable numérique ?
La solution la plus simple consiste à utiliser
Hmisc::cut2(). Vous pouvez également utiliser
dplyr::case_when(). Cependant cette solution nécessite plus
de lignes de code et est plus appropriée pour les catégorisations
personnalisées. Nous vous proposons ici une solution basée sur
base::cut():
R
dat_clean %>%
# selectionner les colonnes d'intéret
dplyr::select(
study_id,
sex,
date_first_pcr_positive_test,
date_of_birth,
age_in_years
) %>%
# categoriser la variable age [ajouter comme indice dans le challenge]
# replacer avec la valeur maximale si elle est connue
dplyr::mutate(
age_category = base::cut(
x = age_in_years,
breaks = c(0, 20, 35, 60, Inf),
include.lowest = TRUE,
right = FALSE
)
)
SORTIE
# A tibble: 10 × 6
study_id sex date_first_pcr_posit…¹ date_of_birth age_in_years age_category
<chr> <int> <date> <date> <dbl> <fct>
1 PS001P2 1 2020-12-01 1972-06-01 52 [35,60)
2 PS002P2 1 2021-01-01 1952-02-20 73 [60,Inf]
3 PS004P2… NA 2021-02-11 1961-06-15 63 [60,Inf]
4 PS003P2 1 2021-02-01 1947-11-11 77 [60,Inf]
5 P0005P2 2 2021-02-16 2000-09-26 24 [20,35)
6 PS006P2 2 2021-05-02 NA NA <NA>
7 PB500P2 1 2021-02-19 1989-11-03 35 [35,60)
8 PS008P2 2 2021-09-20 1976-10-05 48 [35,60)
9 PS010P2 1 2021-02-26 1991-09-23 33 [20,35)
10 PS011P2 2 2021-03-03 1991-02-08 34 [20,35)
# ℹ abbreviated name: ¹date_first_pcr_positive_testVous pouvez rechercher les valeurs maximales des variables à travers
le résumé obtenu en utilisant la fonction skimr::skim(). Au
lieu de base::cut() vous pouvez également utiliser
Hmisc::cut2(x = age_in_years,cuts = c(20,35,60)) qui
calcule la valeur maximale et ne nécessite pas plus d’arguments.
Plusieurs opérations à la fois
L’exécution individuelle des opérations de nettoyage des données peut
prendre beaucoup de temps et être source d’erreurs. La fonction
clean_data() de la librairie cleanepi permet
de simplifier ce processus en permettant d’exécuter plusieurs opérations
à la fois.
Lorsque l’utilisateur ne spécifie aucune opération de nettoyage de
données, la fonction applique automatiquement une série d’opérations de
nettoyage de données. Voici un exemple de code illustrant l’utilisation
de la fonction clean_data() sur le jeu de données simulé
d’Ebola:
R
cleaned_data <- cleanepi::clean_data(raw_ebola_data)
SORTIE
ℹ Cleaning column namesSORTIE
ℹ Removing constant columns and empty rowsSORTIE
ℹ Removing duplicated rowsSORTIE
! Found 5 duplicated rows in the dataset.
ℹ Use `attr(dat, "report")[["duplicated_rows"]]` to access them, where "dat" is
the object used to store the output from this operation.En outre, vous pouvez combiner plusieurs opérations de nettoyage de
données via l’opérateur pipe (|>) de la librairie de
base de R ou celui de la librairie {magrittr} (%>%),
comme illustré à travers le code ci-dessous.
R
# Exécuter les opérations de nettoyage de données en utilisant l'opérateur %>%
cleaned_data <- raw_ebola_data %>%
cleanepi::standardize_column_names() %>%
cleanepi::remove_constants() %>%
cleanepi::remove_duplicates() %>%
cleanepi::replace_missing_values(na_strings = "") %>%
cleanepi::check_subject_ids(
target_columns = "case_id",
range = c(1, 15000)
) %>%
cleanepi::standardize_dates(
target_columns = c("date_onset", "date_sample")
) %>%
cleanepi::convert_to_numeric(target_columns = "age") %>%
cleanepi::check_date_sequence(
target_columns = c("date_onset", "date_sample")
) %>%
cleanepi::clean_using_dictionary(dictionary = test_dict) %>%
cleanepi::timespan(
target_column = "date_sample",
end_date = as.Date("2025-01-03"),
span_unit = "years",
span_column_name = "years_since_collection",
span_remainder_unit = "months"
)
Défi
Avez-vous remarqué que cleanepi contient un ensemble de fonctions pour diagnostiquer l’état de propreté des données et un autre ensemble pour effectuer le nettoyage ?
Identifier les deux groupes :
- Sur une feuille de papier, écrivez le nom de chaque fonction dans la colonne correspondante :
| Diagnostiquer la propreté des données | Effectuer le nettoyage des données |
|---|---|
| … | … |
Notez que cleanepi contient un ensemble de fonctions
pour diagnostiquer l’état de propreté des données (par
exemple check_subject_ids() et
check_date_sequence() dans l’extrait de code ci-dessus) et
un autre pour effecture les opérations de nettoyage (le
reste des fonctions dans l’exemple ci-dessus).
Rapport du nettoyage des données
cleanepi génère un rapport complet détaillant les résultats et les actions de tous les fonctions ayant servi au nettoyages de données. Ce rapport se présente sous la forme d’un fichier HTML qui s’ouvrira automatiquement dans votre navigateur par défaut. Il comporte plusieurs sections. Chaque section correspond à une opération spécifique de nettoyage des données, et un clic sur chaque section vous permet d’accéder aux résultats de l’opération de nettoyage des données en question. Cette approche interactive permet aux utilisateurs d’examiner et d’analyser efficacement les effets des opérations de nettoyage des données.
Vous pouvez consulter le rapport de nettage de données à l’aide de la
fonction cleanepi::print_report(cleaned_data).
Exemple de rapport de nettoyage de données généré par
cleanepi
Les rapports de nettoyage de données sont générés par
cleanepi.
Points clés
Content from Valider les données épidémiologiques
Dernière mise à jour le 2025-11-11 | Modifier cette page
Durée estimée : 12 minutes
Vue d'ensemble
Questions
- Comment convertir les données brutes en un objet de la classe
linelist?
Objectifs
- Démontrez comment convertir les données portant sur les cas
épidémiques en objet de type
linelist - Démontrer comment étiqueter et valider les données pour rendre l’analyse plus fiable
Pré-requis
Pour cet épisode vous devez :
- Télécharger le fichier cleaned_data.csv
- Et le sauvegarder dans le dossier
data/.
Introduction
Après la lecture et le nettoyage des données, il est essentiel
d’ajouter une étape supplémentaire fondamentale pour garantir
l’intégrité et la fiabilité durant le processus d’analyse des données
épidémiologiques. Dans le cas contraire, vous risquez de rencontrer des
problèmes lors de l’analyse des données à cause de la création ou
suppression de certaines variables ou de la modification de leur type
(tel que <date> ou <chr>), etc.
Plus précisément, cette étape supplémentaire implique :
- La vérification de la présence de certaines colonnes et de leur type de données. Ce processus est communément appelé étiquetage ;
- La mise en œuvre de mesures pour s’assurer que ces colonnes étiquettées ne soient pas supprimées par inadvertance lors du traitement des données. On parle de validation des données.
Cet épisode est consacré à l’étiquettage et la validation des données
épidémiologiques à l’aide de la librairie {linelist}. Nous
aurons besoin de la librairie rio pour importer les
données, de linelist pour créer un objet de la classe
linelist. Nous utiliserons l’opérateur pipe (%>%) pour
connecter certaines de leurs fonctions, y compris celles de la librairie
dplyr. Pour cette raison, nous allons donc charger la
librairie {tidyverse}.
R
# charger packages
library(tidyverse)
library(rio)
library(here)
library(linelist) # pour etiquetter et valider les donnees
L’opérateur double deux-points
(::)
L’opérateur :: de R permet d’accéder aux fonctions ou
aux objets d’un package spécifique sans attacher l’intégralité du
package (sans faire appel à la functionlibray()). Il offre
plusieurs avantages, notamment :
- Indiquer explicitement le package d’origine d’une fonction, réduisant ainsi les ambiguïtés et les conflits potentiels lorsque plusieurs packages possèdent des fonctions portant le même nom.
- Permettre d’appeler une fonction depuis un package sans charger
l’intégralité du package avec
library().
Par exemple, la commande dplyr::filter(data, condition)
signifie que nous appelons la fonction filter() depuis la
librairie dplyr.
Importez le jeu de données en suivant les directives énoncées dans l’épisode document Lecture des données épidémiologiques. Il s’agit d’importer les données dans l’environnement de travail et de visualiser sa structure et son contenu.
R
# lecture des données
# supposons que le chemin d'acces au fichier
# est: data/simulated_ebola_2.csv, alors:
cleaned_data <- rio::import(
here::here("data", "cleaned_data.csv")
) %>%
dplyr::as_tibble()
# voir un aperçu des 10 premieres ligne du tableau de donnees
cleaned_data
SORTIE
# A tibble: 15,000 × 10
v1 case_id age gender status date_onset date_sample row_id
<int> <int> <dbl> <chr> <chr> <IDate> <IDate> <int>
1 1 14905 90 male confirmed 2015-03-15 2015-06-04 1
2 2 13043 25 female <NA> 2013-09-11 2014-03-01 2
3 3 14364 54 female <NA> 2014-09-02 2015-03-03 3
4 4 14675 90 <NA> <NA> 2014-10-19 2031-12-14 4
5 5 12648 74 female <NA> 2014-08-06 2016-10-10 5
6 6 14274 76 female <NA> 2015-04-05 2016-01-23 7
7 7 14132 16 male confirmed NA 2015-05-10 8
8 8 14715 44 female confirmed NA 2016-04-24 9
9 9 13435 26 male <NA> 2014-09-07 2020-09-14 10
10 10 14816 30 female <NA> 2015-06-29 2015-06-02 11
# ℹ 14,990 more rows
# ℹ 2 more variables: years_since_collection <int>, remainder_months <int>Discussion
Avez-vous déjà été confronté à une modification inattendue de certaines observations et/ou colonnes de vos données d’entrée lors d’une analyse épidémiologique ? Comment protégez-vous votre analyse de ce désagrément ?
Un changement inattendu
Vous êtes dans une situation d’urgence. Vous devez produire des rapports de situation quotidiennes. Vous avez automatisé votre analyse pour lire les données directement à partir du serveur en ligne :grin :. Cependant, les personnes chargées de la collecte et de l’administration des données devaient supprimer/renommer/reformater une variable qui s’est afférée utile 😞 !
Comment pouvez-vous détecter si les données saisies sont toujours valides pour reproduire l’analyse en utilisant le même code que la veille ?
Si les apprenants n’ont pas d’expérience à partager à ce sujet, nous, en tant qu’instructeurs, pouvons en partager une.
Un tel scénario se produit généralement lorsque l’institution qui effectue l’analyse des données n’est pas la même que celle qui collecte les données. Cette dernière peut prendre des décisions sur la structure et le format des données qui peuvent affecter les processus en aval et avoir un impact sur la durée l’analyse et la précision des résultats.
Création d’un objet de type linelist et étiquetage des colonnes
Après avoir importé et nettoyé les données, nous pouvons les
convertir en un objet de la classe linelist à l’aide de la
fonction make_linelist() de la librairie
linelist comme dans l’exemple suivant ci-dessous.
R
# creer un objet de type linelist a partir des donnees nettoyees
linelist_data <- linelist::make_linelist(
x = cleaned_data, # les donnees d'entree
id = "case_id", # colonne avec les identifiants uniques des individus
date_onset = "date_onset", # colonne avec la date d'apparition des symptomes
gender = "gender" # colonne ayant le genre des individus
)
# voir un aperçu de l'objet cree
linelist_data
SORTIE
// linelist object
# A tibble: 15,000 × 10
v1 case_id age gender status date_onset date_sample row_id
<int> <int> <dbl> <chr> <chr> <IDate> <IDate> <int>
1 1 14905 90 male confirmed 2015-03-15 2015-06-04 1
2 2 13043 25 female <NA> 2013-09-11 2014-03-01 2
3 3 14364 54 female <NA> 2014-09-02 2015-03-03 3
4 4 14675 90 <NA> <NA> 2014-10-19 2031-12-14 4
5 5 12648 74 female <NA> 2014-08-06 2016-10-10 5
6 6 14274 76 female <NA> 2015-04-05 2016-01-23 7
7 7 14132 16 male confirmed NA 2015-05-10 8
8 8 14715 44 female confirmed NA 2016-04-24 9
9 9 13435 26 male <NA> 2014-09-07 2020-09-14 10
10 10 14816 30 female <NA> 2015-06-29 2015-06-02 11
# ℹ 14,990 more rows
# ℹ 2 more variables: years_since_collection <int>, remainder_months <int>
// tags: id:case_id, date_onset:date_onset, gender:gender La librairie linelist fournit des étiquettes aux
variables épidémiologiques courantes et leurs associe un ensemble de
types de données appropriés. Vous pouvez consulter la liste des
étiquettes disponibles à partir du nom de la variable et les types de
données acceptables pour chacune d’entre elles en utilisant la fonction
linelist::tags_types().
Défi
Etiquettons d’autres variables. Dans certains jeu de données, il est possible de trouver des noms de variables qui sont différents des noms des étiquettes disponibles. Dans ces cas là, nous pouvons les associer en fonction de la façon dont les variables ont été définies durant la collecte des données.
Maintenant:
- Explorer les noms de étiquettes disponibles dans {linelist}.
- Trouver quelles autres variables de ce jeu de données peuvent être associées à l’une des étiquettes disponibles.
-
Étiquetter ces variables comme on l’a montré
ci-dessus en utilisant la fonction
linelist::make_linelist().
Vous pouvez accéder à la liste des noms des étiquettes disponibles dans la librairie {linelist} en utilisant :
R
# obtenir la liste des noms et types des etiquettes
linelist::tags_types()
# obtenir la liste des noms des etiquettes uniquement
linelist::tags_names()
R
linelist::make_linelist(
x = cleaned_data,
id = "case_id",
date_onset = "date_onset",
gender = "gender",
age = "age", # meme nom que celui utilise dans la liste des etiquettes
date_reporting = "date_sample" # noms differents mais lies
)
Comment apparaissent ces étiquettes supplémentaires dans le nouveau objet ?
Validation
Pour s’assurer que toutes les variables étiquettées sont aux normes
et que leurs types de données sont correctes, utilisez la fonction
linelist::validate_linelist() comme le montre l’exemple
ci-dessous :
R
linelist::validate_linelist(linelist_data)
SORTIE
'linelist_data' is a valid linelist objectDéfi
Imaginons le scénario suivant lors d’une épidémie en cours. Vous remarquez à un moment donné que la source de données sur laquelle vous vous appuyiez comporte un ensemble de nouvelles entrées (c’est-à-dire des lignes ou des observations) et que le type de données d’une variable a changé.
Prenons l’exemple où le type de la variable age est
passé de double (<dbl>) à caractère
(<chr>).
Pour simuler cette situation :
- Modifiez le type de données de la variable,
- Étiquetez la variable dans un object linelist, puis
- Validez la variable.
Décrivez comment linelist::validate_linelist() réagit
lorsque le type de données d’une variable a été modifié.
Nous pouvons utiliser dplyr::mutate() pour modifier le
type de la variable avant de l’étiqueter pour la validation.
R
cleaned_data %>%
# modifier le type de donnees d'une variable
dplyr::mutate(age = as.character(age)) %>%
# etiqueter variable
linelist::... %>%
# valider l'objet linelist
linelist::...
Veuillez exécuter le code ligne par ligne, en vous concentrant uniquement sur les parties situées avant l’opérateur pipe (
%>%). Après chaque étape, observez la sortie avant de passer à la ligne suivante.
Si le résultat age passe de double
(<dbl>) en caractère (<chr>), nous
obtenons ce qui suit :
R
cleaned_data %>%
# convertir la variable 'age' en chaine de caracteres
dplyr::mutate(age = as.character(age)) %>%
# etiqueter la variable age
linelist::make_linelist(age = "age") %>%
# valider l'objet linelist
linelist::validate_linelist()
ERREUR
Error: Some tags have the wrong class:
- age: Must inherit from class 'numeric'/'integer', but has class 'character'Pourquoi recevons-nous un message d'Erreur ?
Devrions-nous avoir un message d’alerte à la place ? Expliquez pourquoi. Explorez d’autres situations pour comprendre ce comportement en convertissant:
-
date_onsetde<date>en caractère (<chr>), -
genderde caractère (<chr>) en nombre entier (<int>).
Ensuite, étiquetez-les dans dans l’objet linelist pour validation.
Est-ce que le message d'Erreur nous propose-t-il une
solution ?
R
# deuxieme changement
# executer ce code ligne par ligne pour voir les changements
cleaned_data %>%
# modifier le type de la variable 'date_onset'
dplyr::mutate(date_onset = as.character(date_onset)) %>%
# tiqueter le variable 'date_onset'
linelist::make_linelist(
date_onset = "date_onset"
) %>%
# valider l'objet linelist
linelist::validate_linelist()
R
# troisieme changement
# executer ce code ligne par ligne pour voir les changements
cleaned_data %>%
# modifier le type de la variable 'gender'
dplyr::mutate(gender = as.factor(gender)) %>%
dplyr::mutate(gender = as.integer(gender)) %>%
# etiqueter la variable 'gender'
linelist::make_linelist(
gender = "gender"
) %>%
# valider l'objet linelist
linelist::validate_linelist()
Nous obtenons des messages d'Erreur car le type par
défaut de ces variables dans linelist::tags_types() est
différent de celui dont on les a associé.
Le message d'Erreur nous informe que pour
valider notre objet linelist, nous devons changer le
type de variable d’entrée de sorte qu’il corresponde au type d’étiquette
attendu. Dans un script d’analyse de données, nous pouvons le faire en
ajoutant une étape de nettoyage dans le pipeline.
Défi
En dehors de l’étiquetage et de la validation de l’objet linelist, de quelle autre étape avons-nous besoin lors de la construction de l’objet ?
Voici un scénario où nous allons essayer d’étiqueter une variable qui n’existe pas dans notre jeu de données.
R
cleaned_data %>%
# supprimer la variable 'age'
select(-age) %>%
# etiqueter la variable 'age' qui n'existe plus
linelist::make_linelist(age = "age")
ERREUR
Error in base::tryCatch(base::withCallingHandlers({: 1 assertions failed:
* Variable 'tag': Must be element of set
* {'v1','case_id','gender','status','date_onset','date_sample','row_id','years_since_collection','remainder_months'},
* but is 'age'.On a donc besoin de mettre en place un mécanisme pour protéger les données de toutes actions inopportunes.
Protéger les données
La protection des données est implicitement intégrée dans les objets de la classe linelist. Si vous essayez de supprimer l’une des variables étiquetées, vous recevrez un message d’erreur ou d’avertissement, comme le montre l’exemple ci-dessous.
R
new_df <- linelist_data %>%
dplyr::select(case_id, gender)
AVERTISSEMENT
Warning: The following tags have lost their variable:
date_onset:date_onsetCe message d’avertissement qui s’affiche est l’option par défaut
lorsque nous perdons des étiquetes dans un objet de la classe linelist.
Cependant, il peut être remplacé par un message d'Erreur en
utilisant la fonction linelist::lost_tags_action().
Défi
Testons les implications de la variation de la configuration de
protection d’un objet linelist de
Avertissement à Erreur.
- Tout d’abord, exécutez ce code pour compter la fréquence des catégorie au sein d’une variable catégorielle :
R
linelist_data %>%
dplyr::select(case_id, gender) %>%
dplyr::count(gender)
- Spéficier qu’on souhaite obtenir un message d’Erreur au lieu d’un Avertissement à la suite de la perte d’une variable étiquetée de l’objet linelist :
R
# retourner une erreur
linelist::lost_tags_action(action = "error")
- Maintenant, réexécutez le bloc de code ci-dessus avec
dplyr::count(). - Quelle est la différence entre le message
d'Avertissementet le messaged'Erreur? - Quelles pourraient être les implications de ce changement pour votre pipeline d’analyse quotidienne de données lors d’une réponse à une épidémie ?
Décider entre Avertir et continuer l'exécution du code
ou Arréter l'exécution du code avec un message d'erreur
dépendra du niveau d’importance et de flexibilité dont vous avez besoin
lorsque vous perdez des étiquettes. L’un vous alertera d’un changement
mais continuera à exécuter le code en aval. L’autre arrêtera votre
pipeline d’analyse et le reste ne sera pas exécuté.
Un script de lecture, nettoyage et validation de données peut nécessiter un pipeline plus stable ou fixe. Une analyse exploratoire des données peut nécessiter une approche plus souple. Ces deux processus peuvent être isolés dans des scripts ou des dossiers différents afin d’adapter le mécanisme de protection à vos besoins.
Avant de continuer, rétablissez la configuration à l’option par
défaut de Avertissement:
R
# retablir l'option par defaut: "warning"
linelist::lost_tags_action()
SORTIE
Lost tags will now issue a warning.A objet linelist ressemble à un data frame mais offre
des caractéristiques et fonctionnalités plus riches. Les librairies qui
prennent en compte les objets linelist peuvent exploiter ces
fonctionnalités. Par exemple, vous pouvez extraire un data frame
contenant uniquement les colonnes étiquetées à l’aide de la fonction
linelist::tags_df() comme indiqué ci-dessous :
R
# extraire uniquement les colonnes étiquetées
linelist::tags_df(linelist_data)
SORTIE
# A tibble: 15,000 × 3
id date_onset gender
<int> <IDate> <chr>
1 14905 2015-03-15 male
2 13043 2013-09-11 female
3 14364 2014-09-02 female
4 14675 2014-10-19 <NA>
5 12648 2014-08-06 female
6 14274 2015-04-05 female
7 14132 NA male
8 14715 NA female
9 13435 2014-09-07 male
10 14816 2015-06-29 female
# ℹ 14,990 more rowsAvec la librairie cleanepi, vous pouvez également
choisir d’appliquer certaines opérations de nettoyage de données
uniquement sur les les colonnes étiquetées. Il suffira de donner la
valeur "linelist_tags" à l’argument
target_columns de la finction correspondante.
R
# identifier les doublons uniquement à travers les colonnes etiquetees
linelist_data %>%
cleanepi::find_duplicates(target_columns = "linelist_tags")
SORTIE
! Found 177 duplicated rows in the dataset.
ℹ Use `attr(dat, "report")[["duplicated_rows"]]` to access them, where "dat" is
the object used to store the output from this operation.SORTIE
// linelist object
# A tibble: 15,000 × 10
v1 case_id age gender status date_onset date_sample row_id
<int> <int> <dbl> <chr> <chr> <IDate> <IDate> <int>
1 1 14905 90 male confirmed 2015-03-15 2015-06-04 1
2 2 13043 25 female <NA> 2013-09-11 2014-03-01 2
3 3 14364 54 female <NA> 2014-09-02 2015-03-03 3
4 4 14675 90 <NA> <NA> 2014-10-19 2031-12-14 4
5 5 12648 74 female <NA> 2014-08-06 2016-10-10 5
6 6 14274 76 female <NA> 2015-04-05 2016-01-23 7
7 7 14132 16 male confirmed NA 2015-05-10 8
8 8 14715 44 female confirmed NA 2016-04-24 9
9 9 13435 26 male <NA> 2014-09-07 2020-09-14 10
10 10 14816 30 female <NA> 2015-06-29 2015-06-02 11
# ℹ 14,990 more rows
# ℹ 2 more variables: years_since_collection <int>, remainder_months <int>
// tags: id:case_id, date_onset:date_onset, gender:gender Quand dois-je utiliser
{linelist}?
L’analyse des données au cours d’une réponse à une épidémie ou d’une surveillance de masse exige un ensemble de différentes façons de “protéger les données” par rapport aux situations de recherche habituelles. Par exemple, vos données changeront ou seront mises à jour au fil du temps avec de nouvelles entrées, nouvelles variables, des variables seront renommées, etc.
La librairie linelist est plus appropriée pour ce type de surveillance continue, mais aussi aux analyses de données générée sur une longue période. Consultez la section de la vignette “Get started” à propos de When I should consider using {linelist}? pour plus d’informations.
Points clés
- Utiliser la librairie linelist pour étiqueter, valider et protéger les données épidémiologiques pour l’analyse en aval.
Content from Agréger et visualiser les données épidémiologiques
Dernière mise à jour le 2025-11-11 | Modifier cette page
Durée estimée : 30 minutes
Vue d'ensemble
Questions
- Comment agréger et résumer les données épidémiologiques ?
- Comment visualiser les données agrégées ?
- Quelle est la répartition des cas dans le temps, l’espace, en fonction du sexe, l’âge, etc ?
Objectifs
- Simuler des données synthétiques sur les épidémies
- Convertir un tableau de données des cas individuels (linelist) en tableau d’incidence des cas au cours du temps
- Créer des courbes épidémiques à partir de données d’incidence
Introduction
Dans un pipeline d’analyse de données, l’analyse exploratoire des données (AED) est une étape importante avant la modélisation mathématique. L’AED aide à de déterminer les relations entre les variables et de résumer leurs principales caractéristiques, souvent en visualisant les données.
Dans cet épisode, nous allons explorer les techniques d’analyse
exploratoire des données épidémiologiques en utilisant des librairies de
R.
L’un des aspects clés de l’analyse des données épidémiologiques est la notion de “personne, lieu et temps”. Il est important d’identifier comment les événements observés - tels que les nombres de cas confirmés, d’hospitalisations, de décès et de guérisons - évoluent au cours du temps et comment ils varient en fonction des différents localités et facteurs démographiques, notamment le sexe, l’âge, etc.
Commençons par charger la librairie simulist pour
simuler des données épidémiologiques à analyser. Nous utiliserons
ensuite la librairie incidence2 permettant d’agréger un
tableau des données individuelles en fonction de caractéristiques
spécifiques et de visualiser les courbes épidémiques (épicourbes) qui en
résultent. Le tableau de données agrégées obtenu représente le nombre de
nouveaux événements (c’est-à-dire l’incidence des cas au fil du temps).
Pour le formatage des figures, nous utiliserons la librairie
{tracetheme}. Nous utiliserons l’opérateur pipe
(%>%) pour connecter certaines de leurs fonctions, y
compris celles des packages dplyr et
ggplot2, ferons donc faire appel à la librairie
{tidyverse}.
R
# Charger les librairies
library(incidence2) # Pour aggréger et visualiser les données
library(simulist)
library(tracetheme)
library(tidyverse)
L’opérateur double deux-points
(::)
L’opérateur :: de R permet d’accéder aux fonctions ou
aux objets d’un package spécifique sans attacher l’intégralité du
package (sans faire appel à la functionlibray()). Il offre
plusieurs avantages, notamment :
- Indiquer explicitement le package d’origine d’une fonction, réduisant ainsi les ambiguïtés et les conflits potentiels lorsque plusieurs packages possèdent des fonctions portant le même nom.
- Permettre d’appeler une fonction depuis un package sans charger
l’intégralité du package avec
library().
Par exemple, la commande dplyr::filter(data, condition)
signifie que nous appelons la fonction filter() depuis la
librairie dplyr.
Données synthétiques sur les épidémies
Pour illustrer le processus d’analyse exploratoire des données épidémiologiques, nous allons générer un tableau de données d’une épidémie hypothétique à l’aide de la librairie simulist. simulist génère des données simulées pour une épidémie selon la configuration définie. Sa configuration par défaut permet de générer un tableau de données individuelles comme le montre le morceau de code ci-dessous.
Pour rappelle, vous pouvez désormais utiliser la librairie {readepi} pour importer vos données portant sur les cas individuels á partir des systèmes de gestion de base de données relationnelles, de DHIS2 et SORMAS.
R
# initialiser le générateur de nombres aléatoires pour la reproducibilité
set.seed(1)
# simuler un tableau de données
# pour une épidémie de la taille de 1000 à 1500 cas
sim_data <- simulist::sim_linelist(outbreak_size = c(1000, 1500)) %>%
dplyr::as_tibble()
AVERTISSEMENT
Warning: Number of cases exceeds maximum outbreak size.
Returning data early with 1546 cases and 3059 total contacts (including cases).R
# Voir un aperçu des donnees simulees
sim_data
SORTIE
# A tibble: 1,546 × 13
id case_name case_type sex age date_onset date_reporting
<int> <chr> <chr> <chr> <int> <date> <date>
1 1 Zahra al-Masri probable f 37 2023-01-01 2023-01-01
2 3 Waleeda al-Muhammad probable f 12 2023-01-11 2023-01-11
3 6 Rhett Jackson confirmed m 53 2023-01-18 2023-01-18
4 8 Sunnique Sims confirmed f 36 2023-01-23 2023-01-23
5 11 Danielle Griggs probable f 77 2023-01-30 2023-01-30
6 14 Mohamed Parker probable m 37 2023-01-24 2023-01-24
7 15 Melissa Eriacho probable f 67 2023-01-31 2023-01-31
8 16 Maria Laughlin probable f 80 2023-01-30 2023-01-30
9 20 Phillip Park confirmed m 70 2023-01-27 2023-01-27
10 21 Dewarren Newton probable m 87 2023-02-09 2023-02-09
# ℹ 1,536 more rows
# ℹ 6 more variables: date_admission <date>, outcome <chr>,
# date_outcome <date>, date_first_contact <date>, date_last_contact <date>,
# ct_value <dbl>Ce tableau de données contient des enregistements simulés concernant des individus répertoriés lors d’une épidémie.
Ce qui précède est la configuration par défaut de
simulist. Elle suppose un certain nombre d’hypothèses sur
la transmissibilité et la gravité de l’agent pathogène. Si vous
souhaitez en savoir plus sur la fonction
simulist::sim_linelist() et les autres fonctions de cette
librairie, consultez le site avec la
documentation.
Vous pouvez également télécharger des jeux de données
épidémiologiques réelles concernant des épidémies antérieures dans la
librairie {outbreaks}
de R.
Agrégation des données
Nous voudrons généralement analyser et visualiser le nombre de cas
enregistrés en un jour ou une semaine, plutôt que de nous concentrer sur
chaque cas individuel. Pour ce faire, il est nécessaire de regrouper le
tableau des données individuelles en tableau de données d’incidence. La
fonction incidence2::incidence() de la librairie
[{incidence2}] (([https://www.reconverse.org/incidence2/articles/incidence2.html)
permet de regrouper les données individuelles, généralement en fonction
d’événements datés et/ou d’autres caractéristiques. Le morceau de code
fourni ci-dessous montre la procédure de création d’un objet de la
classe incidence2 qui contiendra le nombre agrégé de cas à
partir de la date d’apparition de leurs symptômes en utilisant le
tableau de données d’Ebola simulé.
R
# creer un objet de classe incidence
# en agregeant le nombre de cas
# en fonction de la date d'apparition des symptomes
daily_incidence <- incidence2::incidence(
sim_data,
date_index = "date_onset",
interval = "day" # Aggreger les données de façon journalière
)
# Voir un aperçu des données d'incidence
daily_incidence
SORTIE
# incidence: 232 x 3
# count vars: date_onset
date_index count_variable count
<date> <chr> <int>
1 2023-01-01 date_onset 1
2 2023-01-11 date_onset 1
3 2023-01-18 date_onset 1
4 2023-01-23 date_onset 1
5 2023-01-24 date_onset 1
6 2023-01-27 date_onset 2
7 2023-01-29 date_onset 1
8 2023-01-30 date_onset 2
9 2023-01-31 date_onset 2
10 2023-02-01 date_onset 1
# ℹ 222 more rowsLa librairie incidence2 vous permet de varier l’intervalle de temps à utiliser lorsque vous agrégez vos données (par exemple, les données peuvent être agrégées de façon journalière, hebdomadaire, mensuelle, etc). Elle permet également de classer les cas selon un ou plusieurs facteurs. L’exemple ci-dessous est un extrait de code calculant le nombre hebdomadaire de cas regroupés selon la date d’apparition des symptômes, le sexe et le type de cas.
R
# calculer l'incidence hebdomadaire
# des cas regroupés selon le sexe et le type de cas
weekly_incidence <- incidence2::incidence(
sim_data,
date_index = "date_onset",
interval = "week", # obtenir le nombre hebdomadaire de cas
groups = c("sex", "case_type") # regrouper par sexe et type de cas
)
# voir un aperçu de l'incidence hebdomadaire des cas
weekly_incidence
SORTIE
# incidence: 201 x 5
# count vars: date_onset
# groups: sex, case_type
date_index sex case_type count_variable count
<isowk> <chr> <chr> <chr> <int>
1 2022-W52 f probable date_onset 1
2 2023-W02 f probable date_onset 1
3 2023-W03 m confirmed date_onset 1
4 2023-W04 f confirmed date_onset 2
5 2023-W04 f probable date_onset 1
6 2023-W04 m confirmed date_onset 1
7 2023-W04 m probable date_onset 1
8 2023-W05 f confirmed date_onset 3
9 2023-W05 f probable date_onset 4
10 2023-W05 f suspected date_onset 1
# ℹ 191 more rowsCompléter les dates manquantes
Lorsque les cas sont regroupés en fonction de différents facteurs, il
est possible que les événements impliquant ces groupes se produisent à
des dates différentes dans l’objet de type incidence2 qui
en résulte. La fonction incidence2::complete_dates() permet
de insérer les dates manquantes pour chaque groupe de l’objet de la
classe incidence. Par défaut, le nombre de cas de ces groupes sera
représenté par 0 pour cette date (où aucun membre de ces groupe n’avait
été enregistré). Cette fonctionnalité peut également être activées
durant l’appel à la fonction incidence2::incidence() en
donnant la valeur TRUE à l’argument
complete_dates.
R
# creer un objet de la classe incidence
# contenant l'incidence journalière du
# nombre de cas, groupé par sexe
daily_incidence_2 <- incidence2::incidence(
sim_data,
date_index = "date_onset",
groups = "sex",
interval = "day", # Agreger par interval journalier
complete_dates = TRUE # Compléter les dates manquantes
)
# voir un aperçu de l'incidence journaliere des cas
daily_incidence_2
SORTIE
# incidence: 534 x 4
# count vars: date_onset
# groups: sex
date_index sex count_variable count
<date> <chr> <chr> <int>
1 2023-01-01 f date_onset 1
2 2023-01-01 m date_onset 0
3 2023-01-02 f date_onset 0
4 2023-01-02 m date_onset 0
5 2023-01-03 f date_onset 0
6 2023-01-03 m date_onset 0
7 2023-01-04 f date_onset 0
8 2023-01-04 m date_onset 0
9 2023-01-05 f date_onset 0
10 2023-01-05 m date_onset 0
# ℹ 524 more rowsDéfi 1 : Pouvez-vous le faire ?
-
Tâche : calculer l’incidence
bihebdomadaire des cas dans le tableau de donnèes
sim_dataen fonction de leur date d’admission et de leur revenu. Sauvegarder le résultat dans un objet appelébiweekly_incidence.
Visualisation
Les object de la classe incidence2 peuvent être
visualisés à l’aide de la fonction plot() de la librairie
de base de R. Le graphe qui en résulte est appelé courbe épidémique, ou
épi-courbe. Le code ci-dessous génère des épi-courbes pour lee objets
daily_incidence et weekly_incidence créés
ci-dessus.
R
# visualiser l'incidence journaliere
base::plot(daily_incidence) +
ggplot2::labs(
x = "Time (in days)",
y = "Dialy cases"
) +
tracetheme::theme_trace() # appliquer le theme personnalise de epiverse
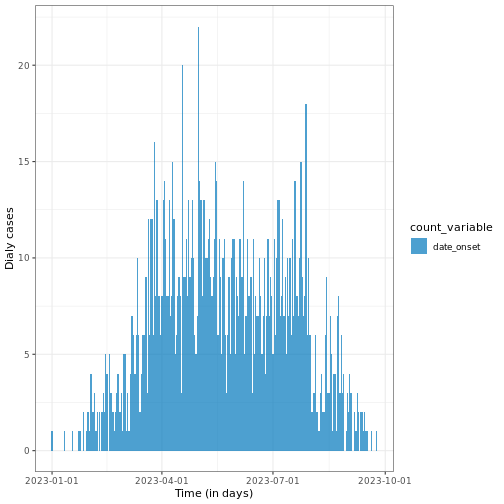
R
# visualiser l'incidence hebdomadaire
base::plot(weekly_incidence) +
ggplot2::labs(
x = "Time (in weeks)",
y = "weekly cases" #
) +
tracetheme::theme_trace()
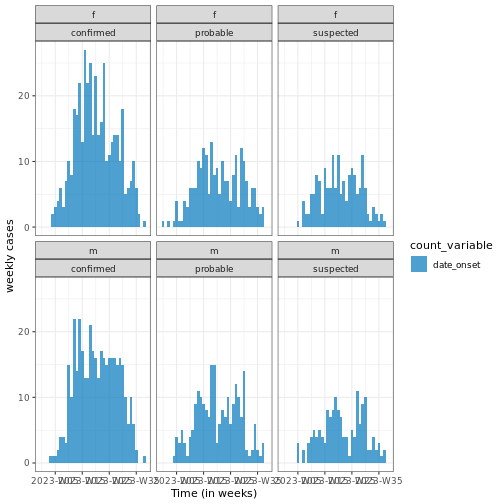
Esthétique simple
Nous vous invitons à parcourir la Vignette
de la librairie incidence2. Découvrez comment vous pouvez
utiliser les arguments de la fonction plot() pour donner de
l’esthétique lors de la visualisation de vos objets de la classe
incidence2.
R
base::plot(weekly_incidence, fill = "sex")
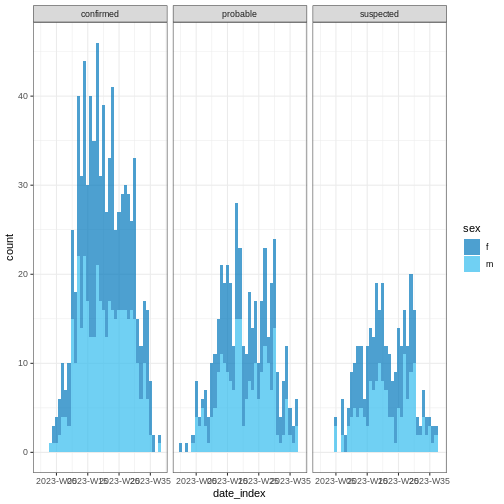
Vous y trouverez entre autre argument:
show_cases = TRUE, angle = 45 et
n_breaks = 5. Essayer de les utiliser pour voir comment ils
changent l’apparence du graphe qui en découle.
Défi 2 : Pouvez-vous le faire ?
-
Tâche visualiser l’objet
biweekly_incidence.
Courbe du nombre cumulé de cas
Le nombre cumulé de cas peut être calculé à l’aide de la fonction
incidence2::cumulate() à partir d’un objet de la classe
incidence2 et peut être visualisé de la même façon que dans
les exemples précédents.
R
# calculer l'incidence cumulée
cum_df <- incidence2::cumulate(daily_incidence)
# visualiser l'incidence cumulée avec {ggplot2}
base::plot(cum_df) +
ggplot2::labs(
x = "Time (in days)",
y = "weekly cases" #
) +
tracetheme::theme_trace()
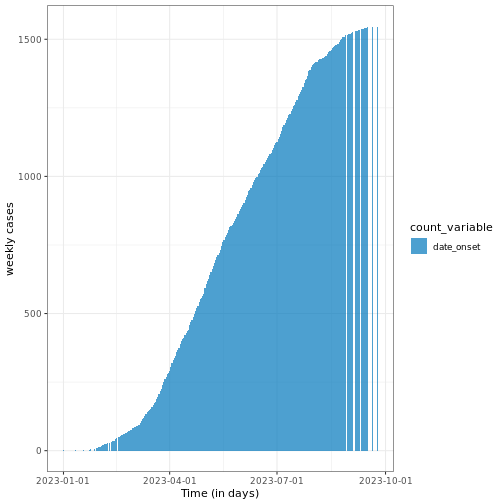
Notez que cette fonction préserve le regroupement, c’est-à-dire que
si l’objet de la classeincidence2 contient des groupes,
elle accumulera les cas en conséquence.
Défi 3 : Pouvez-vous le faire ?
-
Tâche visuliser le nombre cumulé de cas de l’objet
biweekly_incidence.
Estimation la date du pic de l’épidémie
Vous pouvez estimer le pic - le moment où le nombre de cas
enregistrés est le plus élevé - à l’aide de la fonction
incidence2::estimate_peak() de la librairie {incidence2}.
Cette fonction utilise une méthode de bootstrap pour déterminer la
période de pic du nombre de cas (c’est-à-dire en rééchantillonnant les
dates avec remplacement, ce qui donne une distribution des périodes de
pic estimés).
R
# estimer le pic de l'incidence journaliere
peak <- incidence2::estimate_peak(
daily_incidence,
n = 100, # nombre de simulations à utiliser pour trouver le pic
alpha = 0.05, # niveau de significativite pour l'interval de confiance
first_only = TRUE, # retourner la valeur du premier pic trouvé
progress = FALSE # Désactiver les messages de progression
)
# voir un aperçu du pic estime
print(peak)
SORTIE
# A tibble: 1 × 7
count_variable observed_peak observed_count bootstrap_peaks lower_ci
<chr> <date> <int> <list> <date>
1 date_onset 2023-05-01 22 <df [100 × 1]> 2023-03-26
# ℹ 2 more variables: median <date>, upper_ci <date>Cet exemple montre comment estimer la date du pic de l’épidémie (date
à laquelle le nombre de cas le plus élevé a été répertorié) à l’aide de
la fonction incidence2::estimate_peak() avec un interval de
confiance de \(95%\) et en
ré-échantillonnant 100 fois.
Défi 4 : Pouvez-vous le faire ?
-
Tâche Estimation la période du pic de l’épidémie à
partir de l’objet
biweekly_incidence.
Visualisation avec ggplot2
incidence2 permet d’obtenir des graphes basiques pour les épi-courbes, mais un travail supplémentaire est nécessaire pour créer des graphiques bien annotés. Cependant, en urilisant la librairie ggplot2 vous pouvez générer des graphes plus sophistiqués et des épi-courbes avec plus de flexibilité dans leur annotation. ggplot2 est un package qui offre de nombreuses fonctionnalités. Cependant, nous nous focaliserons sur trois éléments clés pour la production d’épi-courbes : les histogrammes, la mise à l’échelle des axes des variables de type Date et de leurs étiquettes, et l’annotation générale du thème du graphe. L’exemple ci-dessous montre comment configurer ces trois éléments pour un simple objet de la classe incidence2.
R
# definir comment les dates seront reparties sur l'axe des x
breaks <- seq.Date(
from = min(as.Date(daily_incidence$date_index, na.rm = TRUE)),
to = max(as.Date(daily_incidence$date_index, na.rm = TRUE)),
by = 20 # chaque 20 jours
)
# creation de l'histogramme
ggplot2::ggplot(data = daily_incidence) +
geom_histogram(
mapping = aes(
x = as.Date(date_index),
y = count
),
stat = "identity",
color = "blue", # couleur de bordure de l'histogramme
fill = "lightblue", # couleur de remplissage des bars de l'histogramme
width = 1 # l'epaisseur des bars de l'histogramme
) +
theme_minimal() + # utiliser un theme minimal
theme(
plot.title = element_text(face = "bold",
hjust = 0.5), # centrer le titre du gaphe en gras
plot.subtitle = element_text(hjust = 0.5), # centrer le sous-titre
plot.caption = element_text(face = "italic",
hjust = 0), # mettre la legende en italique
axis.title = element_text(face = "bold"), # mettre les noms des axes en gras
axis.text.x = element_text(angle = 45, vjust = 0.5) # tourner des 45 degrés
) +
labs(
x = "Date", # definir le nom de l'axe des x
y = "Number of cases", # definir le nom de l'axe des y
title = "Daily Outbreak Cases", # definir le titre du graphe
subtitle = "Epidemiological Data for the Outbreak",
caption = "Data Source: Simulated Data" # definir la legende du graphe
) +
scale_x_date(
breaks = breaks, # definir les limites personnalisee pour l'axe des x
labels = scales::label_date_short() # Raccourcir les noms sur l'axe x
)
AVERTISSEMENT
Warning in geom_histogram(mapping = aes(x = as.Date(date_index), y = count), :
Ignoring unknown parameters: `binwidth`, `bins`, and `pad`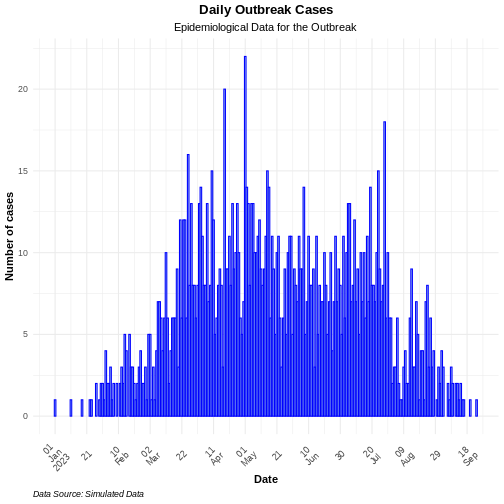
Utilisez l’option group dans la fonction mapping pour
visualiser une courbe épidémique avec différents groupes. Si plusieurs
variables ont été utilisées pour regrouper les cas, utilisez l’option
facet_wrap() comme le montre l’exemple ci-dessous :
R
# visualiser l'incidence journaliere avec des facettes liées au sexe
ggplot2::ggplot(data = daily_incidence_2) +
geom_histogram(
mapping = aes(
x = as.Date(date_index),
y = count,
group = sex, # grouper les bars par sexe
fill = sex
),
stat = "identity"
) +
theme_minimal() +
theme(
plot.title = element_text(face = "bold", hjust = 0.5),
plot.subtitle = element_text(hjust = 0.5),
plot.caption = element_text(face = "italic", hjust = 0),
axis.title = element_text(face = "bold"),
axis.text.x = element_text(angle = 45, vjust = 0.5)
) +
labs(
x = "Date",
y = "Number of cases",
title = "Daily Outbreak Cases by Sex",
subtitle = "Incidence of Cases Grouped by Sex",
caption = "Data Source: Simulated Data"
) +
facet_wrap(~sex) + # creer un panel separant les groupes de la variable sexe
scale_x_date(
breaks = breaks,
labels = scales::label_date_short()
) +
scale_fill_manual(values = c("lightblue",
"lightpink")) # couleur personnalisees
AVERTISSEMENT
Warning in geom_histogram(mapping = aes(x = as.Date(date_index), y = count, :
Ignoring unknown parameters: `binwidth`, `bins`, and `pad`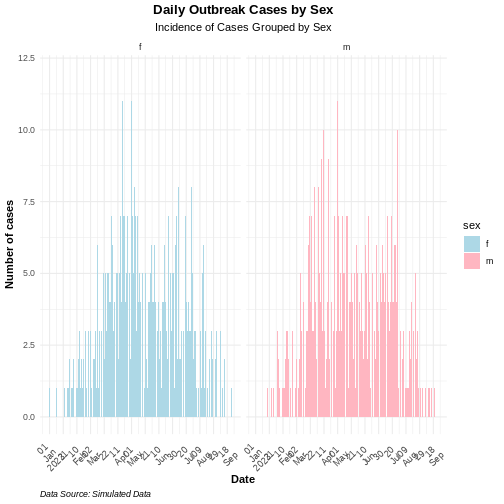
Défi 5 : Pouvez-vous le faire ?
-
Tâche: produire une figure annotée pour l’objet
contenant les incidences bihebdomadiares
biweekly_incidenceà l’aide de la librairie ggplot2.
Points clés
- Utiliser simulist pour générer des données épidémiologiques synthétiques
- Utilisez incidence2 pour agréger les données épidémiologiques en fonction d’événements datés et d’autres variables pour produire des épi-courbes.
- Utilisez ggplot2 pour produire des courbes épidémiques mieux annotées.

Comment cela est-il possible ?
Nous vous invitons à trouver la principale librairie qui rend cette normalisation possible au sein de cleanepi en lisant la section “Détails” du Manuel de référence sur la normalisation des variables de type date!