Acceder a las distribuciones de retrasos epidemiológicos
Última actualización: 2024-11-19 | Mejora esta página
Tiempo estimado: 30 minutos
Hoja de ruta
Preguntas
- ¿Cómo acceder a las distribuciones de retraso de la enfermedad desde una base de datos preestablecida para su uso en el análisis?
Objetivos
- Obtener retrasos de una base de datos de búsqueda bibliográfica con
{epiparameter}. - Obtén parámetros de distribución y estadísticas resumidas de distribuciones de retrasos.
Requisitos previos
Este episodio requiere que estés familiarizado con
Ciencia de datos Programación básica con R
Teoría epidémica : Pparámetros epidemiológicos, periodos de tiempo de la enfermedad, como el periodo de incubación, el tiempo de generación y el intervalo serial.
Introducción
Las enfermedades infecciosas siguen un ciclo de infección, que generalmente incluye las siguientes fases: periodo presintomático, periodo sintomático y periodo de recuperación, tal y como se describe en su historia natural. Estos periodos de tiempo pueden utilizarse para comprender la dinámica de transmisión e informar sobre las intervenciones de prevención y control de enfermedades.
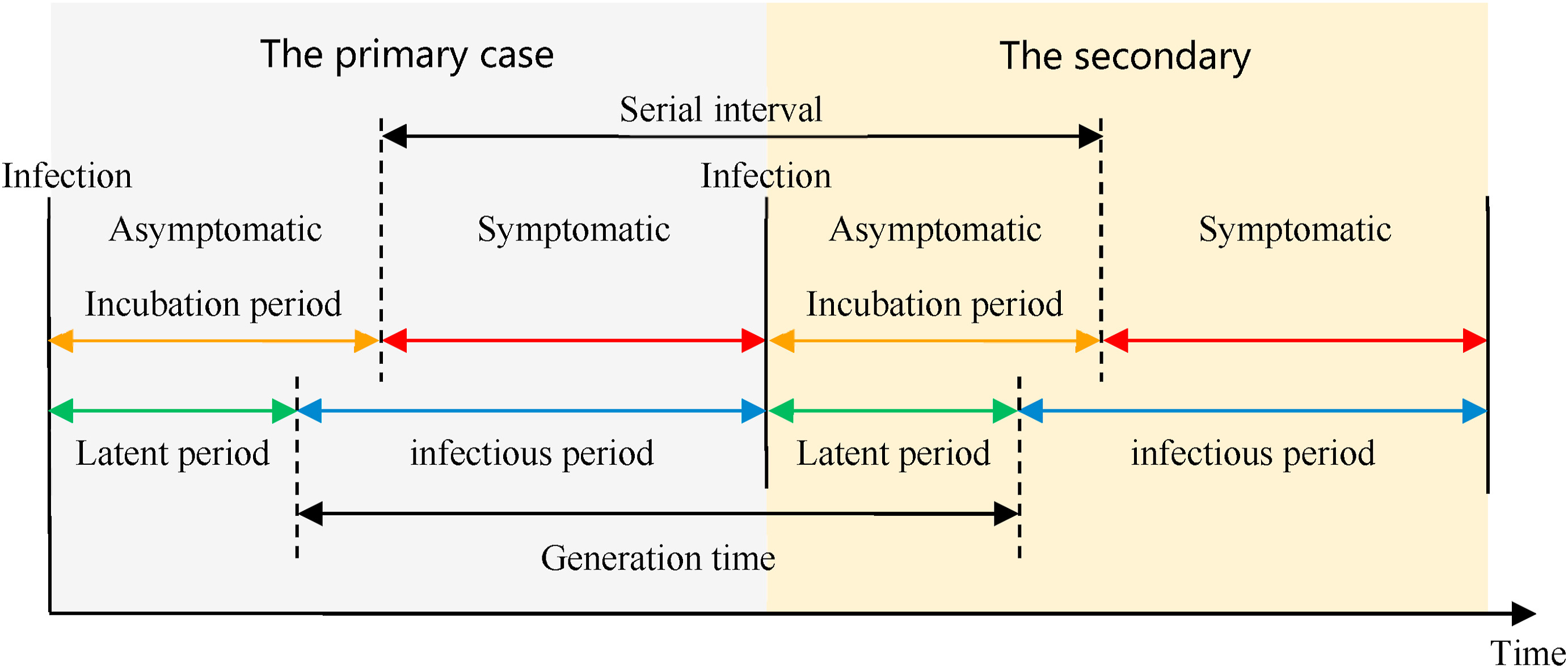
Definiciones
Mira el glosario ¡para ver las definiciones de todos los periodos de tiempo de la figura anterior!
Sin embargo, al inicio de una epidemia, los esfuerzos de modelamiento
pueden verse retrasados por la falta de un recurso centralizado que
resuma los parámetros de entrada para la enfermedad de interés (Nash et al., 2023).
Proyectos como {epiparameter} y {epireview}
están construyendo catálogos en línea siguiendo protocolos de síntesis
de literatura que pueden ayudar a parametrizar modelos accediendo
fácilmente a una extensa biblioteca de parámetros epidemiológicos
previamente estimados de brotes pasados.
Para ejemplificar cómo utilizar el {epiparameter} R en
tu canal de análisis, nuestro objetivo en este episodio será acceder a
un conjunto específico de parámetros epidemiológicos de la literatura,
en lugar de copiarlos y pegarlos manualmente, para integrarlos en un
flujo de trabajo de análisis con EpiNow2
<En este episodio, aprenderemos a acceder a un conjunto concreto
de parámetros epidemiológicos de la bibliografía y a obtener sus
estadísticas resumidas mediante
{epiparameter}. –>
Empecemos cargando el paquete {epiparameter}.
Utilizaremos la tubería %>% para conectar algunas de sus
funciones, algunas funciones detibble y
dplyr, así que llamaremos también al
paquetetidyverse:
R
library(epiparameter)
library(tidyverse)
El doble punto
El doble punto :: en R te permite llamar a una función
específica de un paquete sin cargar todo el paquete en el entorno
actual.
Por ejemplo dplyr::filter(data, condition) utiliza
filter() del paquetedplyr.
Esto nos ayuda a recordar las funciones del paquete y a evitar conflictos de espacio de nombres.
El problema
Si queremos estimar la transmisibilidad de una infección, es común
utilizar un paquete como EpiEstim o
EpiNow2. Sin embargo, ambos requieren cierta información
epidemiológica como entrada. Por ejemplo, en EpiNow2
utilizamos EpiNow2::Gamma() para especificar un tiempo de generación como una
distribución de probabilidad añadiendo su media mean
desviación estándar (sd) y el valor máximo
(max).
Para especificar un tiempo de generación generation_time
que sigue a un Gamma con media \(\mu
= 4\) y desviación estándar \(\sigma =
2\) y un valor máximo de 20, escribimos
R
generation_time <-
EpiNow2::Gamma(
mean = 4,
sd = 2,
max = 20
)
Es una práctica común para analistas, buscar manualmente en la
literatura disponible y copiar y pegar el resumen
estadístico o los parámetros de distribución
de las publicaciones científicas. Un reto frecuente al que nos
enfrentamos a menudo es que la información sobre las distintas
distribuciones estadísticas no es coherente en toda la literatura. El
objetivo de {epiparameter} es facilitar el acceso a
estimaciones confiables de los parámetros de distribución para una serie
de enfermedades infecciosas, de modo que puedan implementarse fácilmente
en las líneas de análisis de brotes.
En este episodio acceder a a las estadísticas resumidas del
tiempo de generación de COVID-19 desde la biblioteca de parámetros
epidemiológicos proporcionada por {epiparameter}. Estos
parámetros pueden utilizarse para estimar la transmisibilidad de esta
enfermedad utilizando EpiNow2 en episodios
posteriores.
Empecemos por ver cuántas entradas hay disponibles en el base
de datos de distribuciones epidemiológicas en
{epiparameter} utilizando epidist_db() para la
distribución epidemiológica epi_dist llamada tiempo de
generación con la cadena "generation":
R
epiparameter::epidist_db(
epi_dist = "generation"
)
SALIDA
Returning 1 results that match the criteria (1 are parameterised).
Use subset to filter by entry variables or single_epidist to return a single entry.
To retrieve the citation for each use the 'get_citation' functionSALIDA
Disease: Influenza
Pathogen: Influenza-A-H1N1
Epi Distribution: generation time
Study: Lessler J, Reich N, Cummings D, New York City Department of Health and
Mental Hygiene Swine Influenza Investigation Team (2009). "Outbreak of
2009 Pandemic Influenza A (H1N1) at a New York City School." _The New
England Journal of Medicine_. doi:10.1056/NEJMoa0906089
<https://doi.org/10.1056/NEJMoa0906089>.
Distribution: weibull
Parameters:
shape: 2.360
scale: 3.180Actualmente, en la biblioteca de parámetros epidemiológicos, tenemos
una entrada de tiempo generación "generation" para
Influenza. En su lugar, podemos consultar intervalos seriales
"serial" para COVID-19. ¡Veamos qué debemos
tener en cuenta para ello!
Tiempo de generación vs intervalo serial
El tiempo de generación, junto con el número reproductivo (\(R\)), proporcionan información valiosa sobre la fuerza de transmisión e informan la implementación de medidas de control. Dado un \(R>1\), cuanto más corto sea el tiempo de generación, más rápidamente aumentará la incidencia de casos de enfermedad.
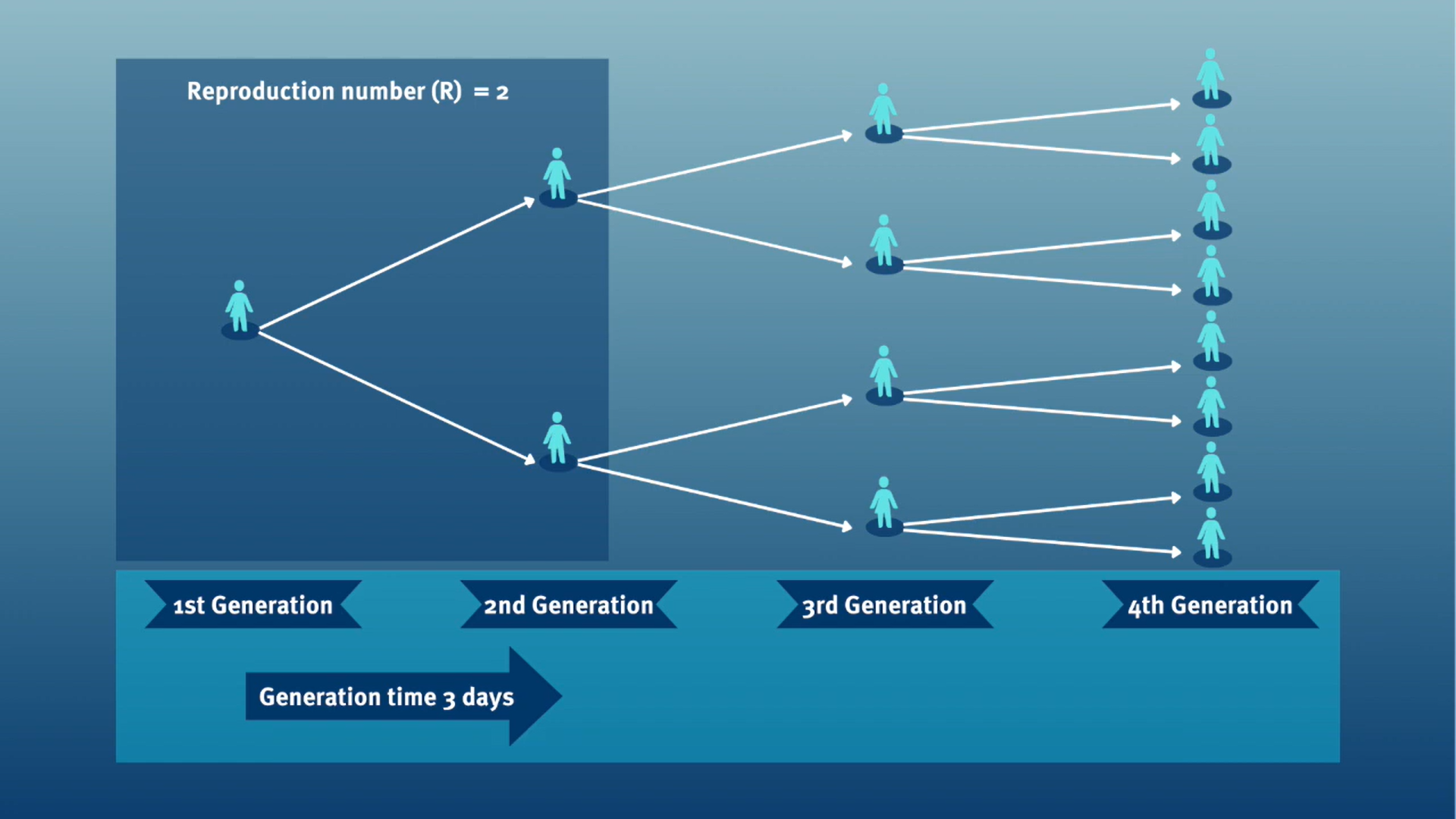
Al calcular el número de reproducción efectivo (\(R_{t}\)), el tiempo de generación suele aproximarse mediante el intervalo serial serial. Esta aproximación frecuente se debe a que es más fácil observar y medir el inicio de los síntomas que el inicio de la infección.
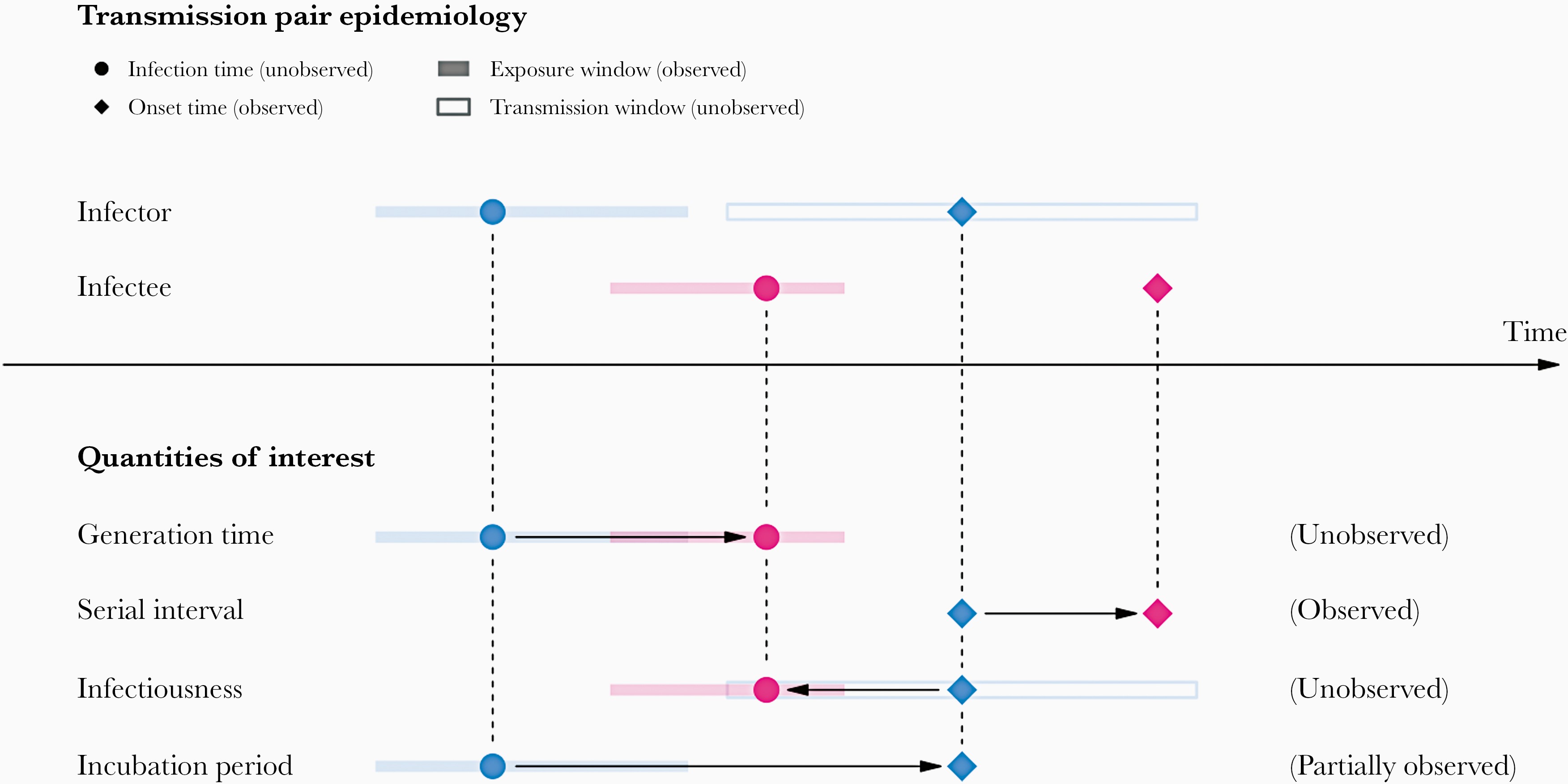
Sin embargo, usar elintervalo serial como una aproximación del tiempo de generación es válido principalmente para las enfermedades en las que la infecciosidad comienza después de la aparición de los síntomas (Chung Lau et al., 2021). En los casos en que la infecciosidad comienza antes de la aparición de los síntomas, los intervalos seriales pueden tener valores negativos, como ocurre en las enfermedades con transmisión presintomática (Nishiura et al., 2020).
De los periodos de tiempo a las distribuciones de probabilidad.
Cuando calculamos el intervalo serial vemos que no todos los pares de casos tienen la misma duración temporal. Observaremos esta variabilidad para cualquier par de casos y periodo de tiempo individual, incluido el periodo de incubación y periodo infeccioso.
Para resumir estos datos de periodos de tiempo individuales y de pares, podemos encontrar las distribuciones estadísticas que mejor se ajusten a los datos (McFarland et al., 2023).
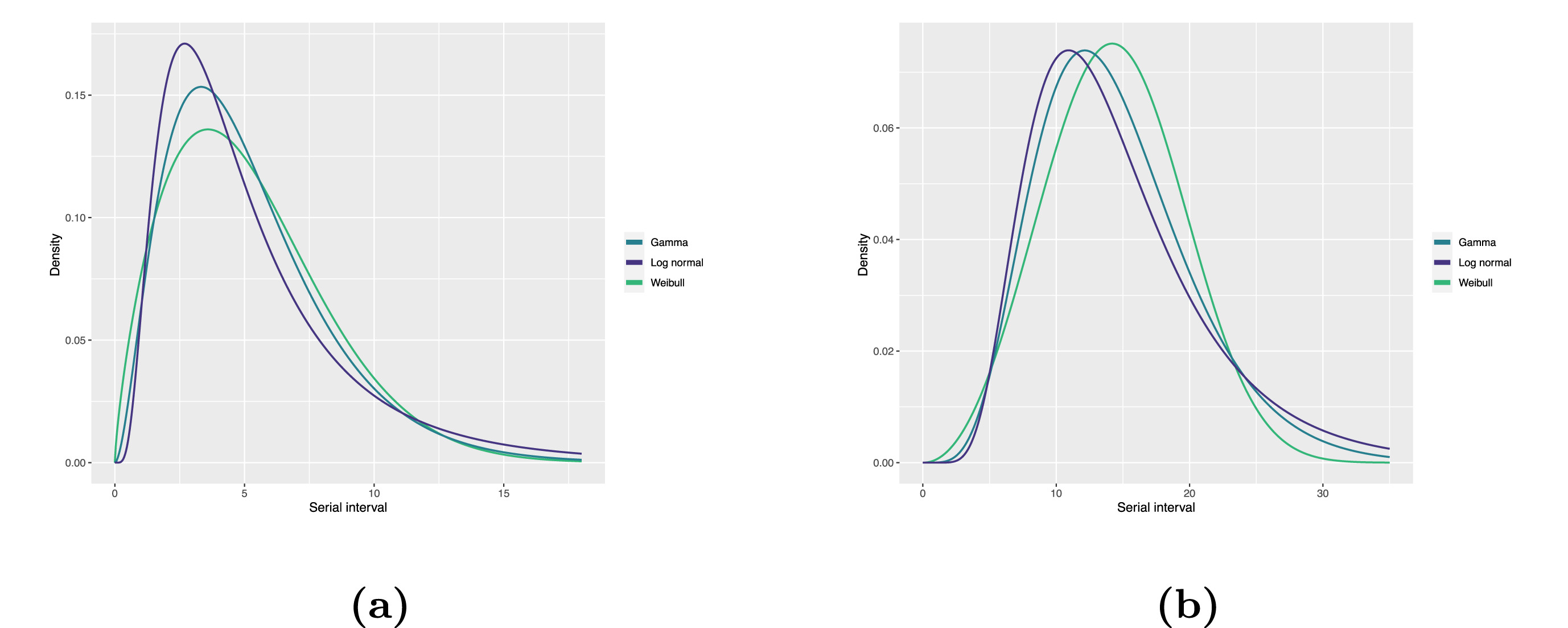
Las distribuciones estadísticas se resumen en función de sus estadísticas de resumen como la ubicación (media y percentiles) y dispersión (varianza o desviación estándar) de la distribución, o con su parámetros de distribución que informan sobre la forma (shape y rate/scale) de la distribución. Estos valores estimados pueden reportarse con su incertidumbre (intervalos de confianza del 95%).
| Gamma | media | forma | velocidad/escala |
|---|---|---|---|
| MERS-CoV | 14.13(13.9-14.7) | 6.31(4.88-8.52) | 0.43(0.33-0.60) |
| COVID-19 | 5.1(5.0-5.5) | 2.77(2.09-3.88) | 0.53(0.38-0.76) |
| Weibull | media | forma | velocidad/escala |
|---|---|---|---|
| MERS-CoV | 14.2(13.3-15.2) | 3.07(2.64-3.63) | 16.1(15.0-17.1) |
| COVID-19 | 5.2(4.6-5.9) | 1.74(1.46-2.11) | 5.83(5.08-6.67) |
| Log normal | media | mean-log | sd-log |
|---|---|---|---|
| MERS-CoV | 14.08(13.1-15.2) | 2.58(2.50-2.68) | 0.44(0.39-0.5) |
| COVID-19 | 5.2(4.2-6.5) | 1.45(1.31-1.61) | 0.63(0.54-0.74) |
Tabla: Estimaciones del intervalo serial utilizando las distribuciones Gamma, Weibull y Log Normal. Los intervalos de confianza del 95% para los parámetros de forma y escala (shape y rate, en inglés) para Gamma (meanlog y sdlog para Log Normal) se muestran entre paréntesis (Althobaity et al., 2022).
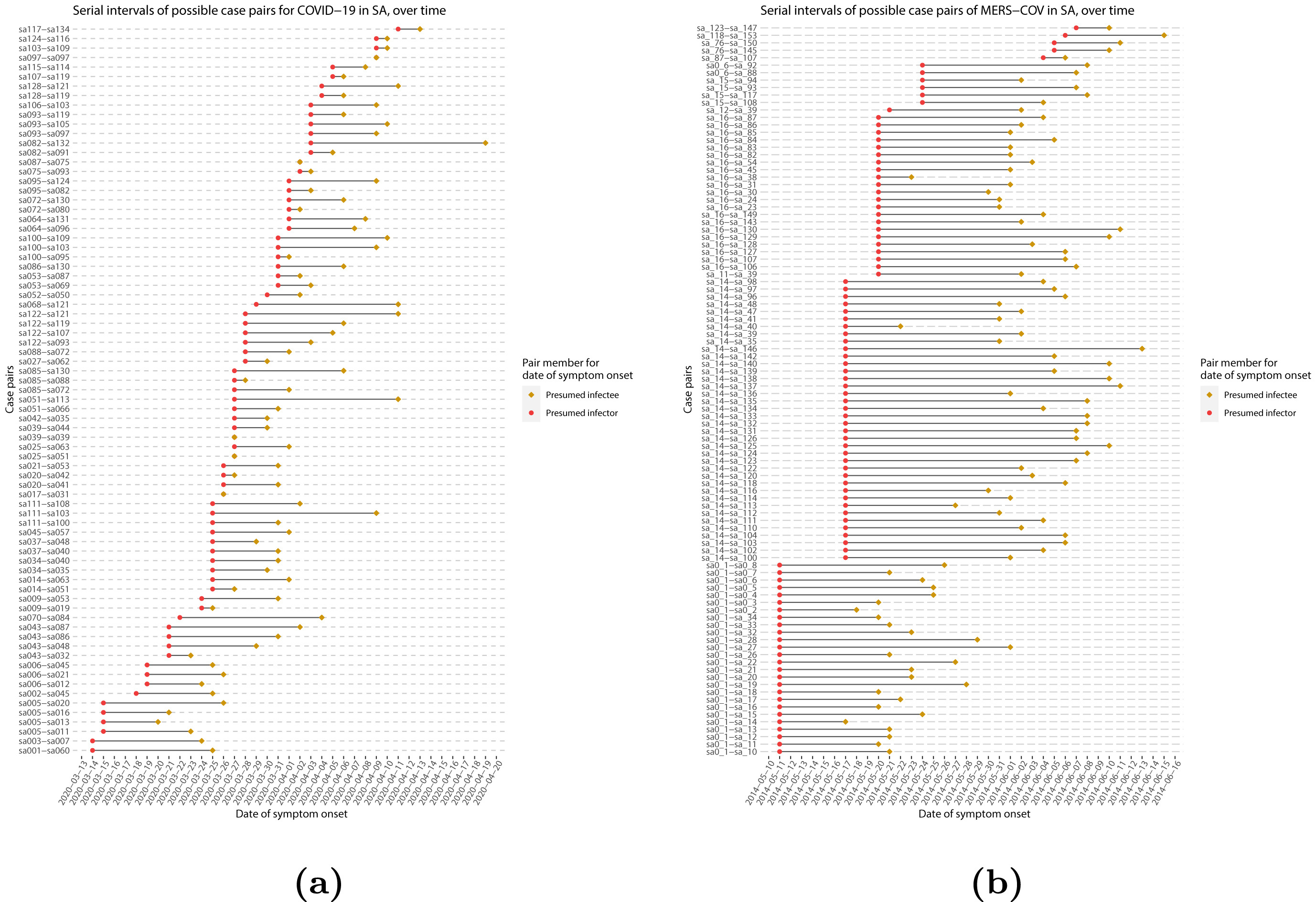
Intervalo serial
Supongamos que COVID-19 y SARS tienen valores similares de número de reproducción y que el intervalo serial se aproxima al tiempo de generación.
Dado el intervalo serial de ambas infecciones en la siguiente gráfica
- ¿Cuál sería más difícil de controlar?
- ¿Por qué llegas a esa conclusión?
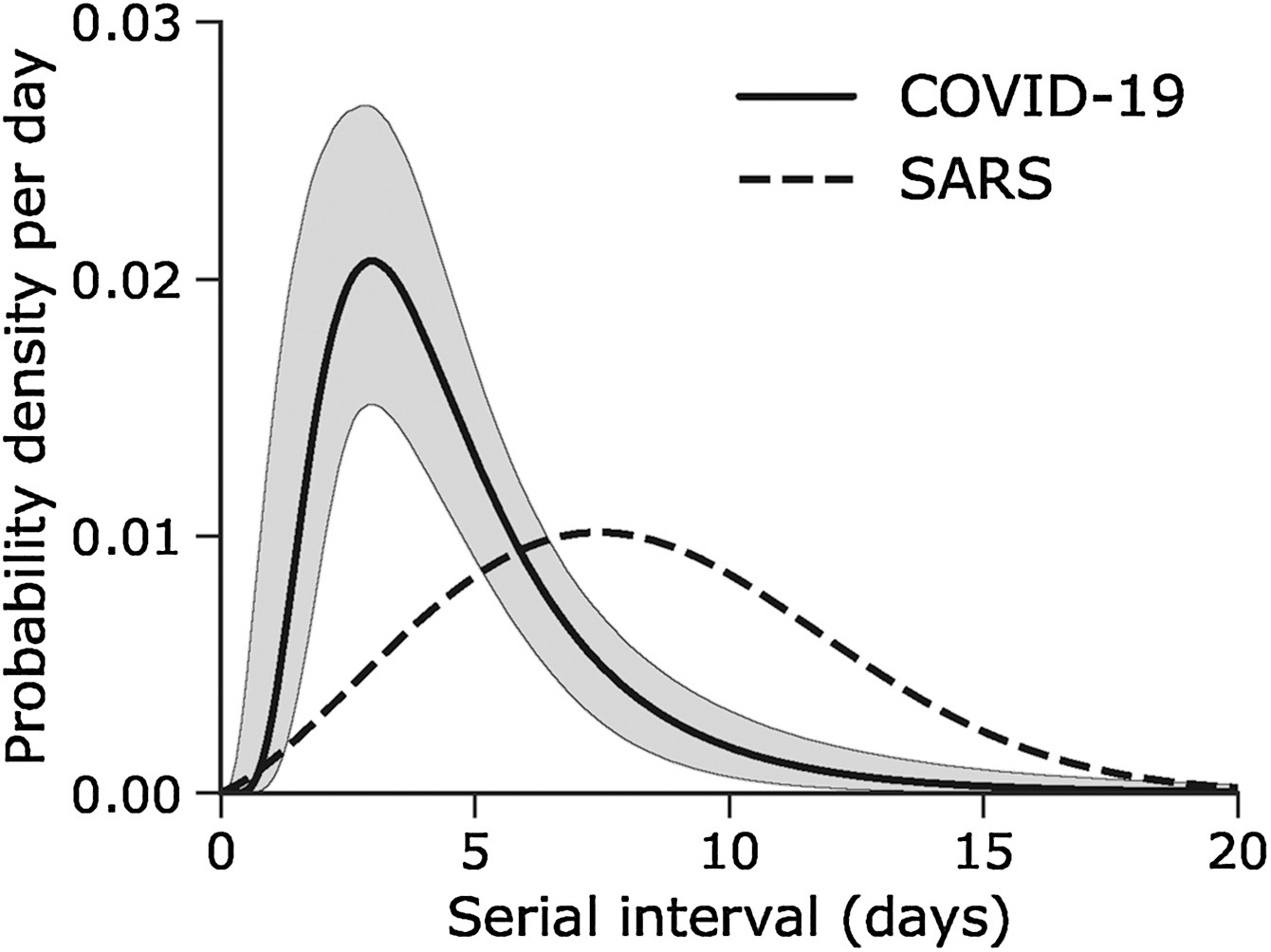
El pico de cada curva puede informarte sobre la ubicación de la media de cada distribución. Cuanto mayor sea la media, mayor será el intervalo serial.
¿Cuál sería más difícil de controlar?
COVID-19
¿Por qué concluyes eso?
COVID-19 tiene el intervalo serial promedioo más bajo. El valor promedio aproximado del intervalo serial de COVID-19 es de unos cuatro días, mientras que del SARS es de aproximadamentesiete días. Por lo tanto, es probable que COVID-19 tenga nuevas generaciones en menos tiempo que el SARS, asumiendo valores de número de reproducción similares.
El objetivo de la evaluación anterior es valorar la interpretación de un tiempo de generación mayor o menor.
Elección de parámetros epidemiológicos
En esta sección, utilizaremos {epiparameter} para
obtener el intervalo serial de COVID-19, como una alternativa al tiempo
de generación.
Preguntémonos ahora cuántos parámetros tenemos en la base de datos de
distribuciones epidemiológicas (epidist_db()) con la
enfermedaddisease denominada covid-19.
¡Ejecútalo localmente!
R
epiparameter::epidist_db(
disease = "covid"
)
Desde el paquete {epiparameter} podemos utilizar la
función epidist_db() para consultar cualquier enfermedad
disease y también para una distribución epidemiológica
concreta (epi_dist). Ejecútalo en tu consola:
R
epiparameter::epidist_db(
disease = "COVID",
epi_dist = "serial"
)
Con esta combinación de consultas, obtenemos más de una distribución
de retraso. Esta salida es un <epidist> objeto de
clase.
INSENSIBLE A MAYÚSCULAS Y MINÚSCULAS
epidist_db es insensible
a mayúsculas y minúsculas. Esto significa que puedes utilizar
cadenas con letras en mayúsculas o minúsculas indistintamente. Cadenas
como "serial", "serial interval" o
"serial_interval" también son válidos.
Como se sugiere en los resultados, para resumir una
<epidist> y obtener los nombres de las columnas de la
base de datos de parámetros subyacente, podemos añadir la función
epiparameter::parameter_tbl() al código anterior utilizando
la tubería %>%:
R
epiparameter::epidist_db(
disease = "covid",
epi_dist = "serial"
) %>%
epiparameter::parameter_tbl()
SALIDA
Returning 4 results that match the criteria (3 are parameterised).
Use subset to filter by entry variables or single_epidist to return a single entry.
To retrieve the citation for each use the 'get_citation' functionSALIDA
# Parameter table:
# A data frame: 4 × 7
disease pathogen epi_distribution prob_distribution author year sample_size
<chr> <chr> <chr> <chr> <chr> <dbl> <dbl>
1 COVID-19 SARS-CoV… serial interval <NA> Alene… 2021 3924
2 COVID-19 SARS-CoV… serial interval lnorm Nishi… 2020 28
3 COVID-19 SARS-CoV… serial interval weibull Nishi… 2020 18
4 COVID-19 SARS-CoV… serial interval norm Yang … 2020 131En el epiparameter::parameter_tbl() salida, también
podemos encontrar distintos tipos de distribuciones de probabilidad (por
ejemplo, Log-normal, Weibull, Normal).
{epiparameter} utiliza la base R para las
distribuciones. Por eso Normal logarítmica se llama
lnorm.
Las entradas con un valor faltante (<NA>) en la
columna prob_distribution son entradas no
parametrizada. Tienen estadísticas de resumen, pero no una
distribución de probabilidad. Compara estos dos resultados:
R
# get an <epidist> object
distribution <-
epiparameter::epidist_db(
disease = "covid",
epi_dist = "serial"
)
distribution %>%
# pluck the first entry in the object class <list>
pluck(1) %>%
# check if <epidist> object have distribution parameters
is_parameterised()
# check if the second <epidist> object
# have distribution parameters
distribution %>%
pluck(2) %>%
is_parameterised()
Las entradas parametrizadas tienen un método de Inferencia
Como se detalla en ?is_parameterised una distribución
parametrizada es la entrada que tiene una distribución de probabilidad
asociada proporcionada por un método inference_method como
se muestra en los metadatosmetadata:
R
distribution[[1]]$metadata$inference_method
distribution[[2]]$metadata$inference_method
distribution[[4]]$metadata$inference_method
¡Encuentra tus distribuciones de retraso!
Tómate 2 minutos para explorar el paquete
{epiparameter}.
Elige una enfermedad de interés (por ejemplo, Influenza estacional, sarampión, etc.) y una distribución de retrasos (por ejemplo, el periodo de incubación, desde el inicio hasta la muerte, etc.).
Encuéntra:
¿Cuántas distribuciones de retraso hay para esa enfermedad?
¿Cuántos tipos de distribución de probabilidad (por ejemplo, gamma, log normal) hay para un retraso determinado en esa enfermedad?
Pregunta:
¿Reconoces los artículos?
¿Debería la revisión de literatura de
{epiparameter}considerar otro artículo?
La función epidist_db() con disease sólo
con la enfermedad cuenta el número de entradas como
- estudios, y
- distribuciones de retrasos.
La función epidist_db() función con la enfermedad
disease y epi_dist obtiene una lista de todas
las entradas con:
- la cita completa,
- en tipo de distribución de probabilidad, y
- valores de los parámetros de la distribución.
La combinación de epidist_db() y
parameter_tbl() obtiene un marco de datos de todas las
entradas con columnas como
- el tipo de la distribución de probabilidad por cada fila, y
- autor y año del estudio.
Elegimos explorar las distribuciones de retraso del Ébola:
R
# we expect 16 delays distributions for ebola
epiparameter::epidist_db(
disease = "ebola"
)
SALIDA
Returning 17 results that match the criteria (17 are parameterised).
Use subset to filter by entry variables or single_epidist to return a single entry.
To retrieve the citation for each use the 'get_citation' functionSALIDA
# List of 17 <epidist> objects
Number of diseases: 1
❯ Ebola Virus Disease
Number of epi distributions: 9
❯ hospitalisation to death ❯ hospitalisation to discharge ❯ incubation period ❯ notification to death ❯ notification to discharge ❯ offspring distribution ❯ onset to death ❯ onset to discharge ❯ serial interval
[[1]]
Disease: Ebola Virus Disease
Pathogen: Ebola Virus
Epi Distribution: offspring distribution
Study: Lloyd-Smith J, Schreiber S, Kopp P, Getz W (2005). "Superspreading and
the effect of individual variation on disease emergence." _Nature_.
doi:10.1038/nature04153 <https://doi.org/10.1038/nature04153>.
Distribution: nbinom
Parameters:
mean: 1.500
dispersion: 5.100
[[2]]
Disease: Ebola Virus Disease
Pathogen: Ebola Virus-Zaire Subtype
Epi Distribution: incubation period
Study: Eichner M, Dowell S, Firese N (2011). "Incubation period of ebola
hemorrhagic virus subtype zaire." _Osong Public Health and Research
Perspectives_. doi:10.1016/j.phrp.2011.04.001
<https://doi.org/10.1016/j.phrp.2011.04.001>.
Distribution: lnorm
Parameters:
meanlog: 2.487
sdlog: 0.330
[[3]]
Disease: Ebola Virus Disease
Pathogen: Ebola Virus-Zaire Subtype
Epi Distribution: onset to death
Study: The Ebola Outbreak Epidemiology Team, Barry A, Ahuka-Mundeke S, Ali
Ahmed Y, Allarangar Y, Anoko J, Archer B, Abedi A, Bagaria J, Belizaire
M, Bhatia S, Bokenge T, Bruni E, Cori A, Dabire E, Diallo A, Diallo B,
Donnelly C, Dorigatti I, Dorji T, Waeber A, Fall I, Ferguson N,
FitzJohn R, Tengomo G, Formenty P, Forna A, Fortin A, Garske T,
Gaythorpe K, Gurry C, Hamblion E, Djingarey M, Haskew C, Hugonnet S,
Imai N, Impouma B, Kabongo G, Kalenga O, Kibangou E, Lee T, Lukoya C,
Ly O, Makiala-Mandanda S, Mamba A, Mbala-Kingebeni P, Mboussou F,
Mlanda T, Makuma V, Morgan O, Mulumba A, Kakoni P, Mukadi-Bamuleka D,
Muyembe J, Bathé N, Ndumbi Ngamala P, Ngom R, Ngoy G, Nouvellet P, Nsio
J, Ousman K, Peron E, Polonsky J, Ryan M, Touré A, Towner R, Tshapenda
G, Van De Weerdt R, Van Kerkhove M, Wendland A, Yao N, Yoti Z, Yuma E,
Kalambayi Kabamba G, Mwati J, Mbuy G, Lubula L, Mutombo A, Mavila O,
Lay Y, Kitenge E (2018). "Outbreak of Ebola virus disease in the
Democratic Republic of the Congo, April–May, 2018: an epidemiological
study." _The Lancet_. doi:10.1016/S0140-6736(18)31387-4
<https://doi.org/10.1016/S0140-6736%2818%2931387-4>.
Distribution: gamma
Parameters:
shape: 2.400
scale: 3.333
# ℹ 14 more elements
# ℹ Use `print(n = ...)` to see more elements.
# ℹ Use `parameter_tbl()` to see a summary table of the parameters.
# ℹ Explore database online at: https://epiverse-trace.github.io/epiparameter/dev/articles/database.htmlAhora, a partir de la salida de
epiparameter::epidist_db() ¿Qué es un distribución de la
descendencia?
Elegimos encontrar los periodos de incubación del ébola. Esta salida lista todos los documentos y parámetros encontrados. Ejecútalo localmente si es necesario:
R
epiparameter::epidist_db(
disease = "ebola",
epi_dist = "incubation"
)
Utilizamos parameter_tbl() para obtener una
visualización resumida de todo:
R
# we expect 2 different types of delay distributions
# for ebola incubation period
epiparameter::epidist_db(
disease = "ebola",
epi_dist = "incubation"
) %>%
parameter_tbl()
SALIDA
Returning 5 results that match the criteria (5 are parameterised).
Use subset to filter by entry variables or single_epidist to return a single entry.
To retrieve the citation for each use the 'get_citation' functionSALIDA
# Parameter table:
# A data frame: 5 × 7
disease pathogen epi_distribution prob_distribution author year sample_size
<chr> <chr> <chr> <chr> <chr> <dbl> <dbl>
1 Ebola Vi… Ebola V… incubation peri… lnorm Eichn… 2011 196
2 Ebola Vi… Ebola V… incubation peri… gamma WHO E… 2015 1798
3 Ebola Vi… Ebola V… incubation peri… gamma WHO E… 2015 49
4 Ebola Vi… Ebola V… incubation peri… gamma WHO E… 2015 957
5 Ebola Vi… Ebola V… incubation peri… gamma WHO E… 2015 792Encontramos dos tipos de distribuciones de probabilidad para esta consulta: log normal y gamma.
¿Cómo realiza {epiparameter} la recopilación y revisión
de la literatura revisada por pares? ¡Te invitamos a leer la viñeta
sobre “Protocolo
de Recopilación y Síntesis de Datos” !
Selecciona una única distribución
En epiparameter::epidist_db() funciona como una función
de filtrado o subconjunto. Utilicemos el argumento author
para filtrar los parámetros Hiroshi Nishiura:
R
epiparameter::epidist_db(
disease = "covid",
epi_dist = "serial",
author = "Hiroshi"
) %>%
epiparameter::parameter_tbl()
SALIDA
Returning 2 results that match the criteria (2 are parameterised).
Use subset to filter by entry variables or single_epidist to return a single entry.
To retrieve the citation for each use the 'get_citation' functionSALIDA
# Parameter table:
# A data frame: 2 × 7
disease pathogen epi_distribution prob_distribution author year sample_size
<chr> <chr> <chr> <chr> <chr> <dbl> <dbl>
1 COVID-19 SARS-CoV… serial interval lnorm Nishi… 2020 28
2 COVID-19 SARS-CoV… serial interval weibull Nishi… 2020 18Seguimos obteniendo más de un parámetro epidemiológico. Podemos
establecer el argumento single_epidist en TRUE
para obtener sólo uno:
R
epiparameter::epidist_db(
disease = "covid",
epi_dist = "serial",
single_epidist = TRUE
)
SALIDA
Using Nishiura H, Linton N, Akhmetzhanov A (2020). "Serial interval of novel
coronavirus (COVID-19) infections." _International Journal of
Infectious Diseases_. doi:10.1016/j.ijid.2020.02.060
<https://doi.org/10.1016/j.ijid.2020.02.060>..
To retrieve the citation use the 'get_citation' functionSALIDA
Disease: COVID-19
Pathogen: SARS-CoV-2
Epi Distribution: serial interval
Study: Nishiura H, Linton N, Akhmetzhanov A (2020). "Serial interval of novel
coronavirus (COVID-19) infections." _International Journal of
Infectious Diseases_. doi:10.1016/j.ijid.2020.02.060
<https://doi.org/10.1016/j.ijid.2020.02.060>.
Distribution: lnorm
Parameters:
meanlog: 1.386
sdlog: 0.568¿Cómo funciona ‘single_epidist’?
Consultando la documentación de ayuda de
?epiparameter::epidist_db():
- Si varias entradas coinciden con los argumentos suministrados y
single_epidist = TRUEentonces devolverá el<epidist>parametrizado con el mayor tamaño de muestra - Si varias entradas son iguales después de esta clasificación, se devolverá la primera entrada.
¿Qué es un <epidist>parametrizado ? Mira
?is_parameterised.
Asignemos este objeto de clase <epidist> al
objetocovid_serialint.
R
covid_serialint <-
epiparameter::epidist_db(
disease = "covid",
epi_dist = "serial",
single_epidist = TRUE
)
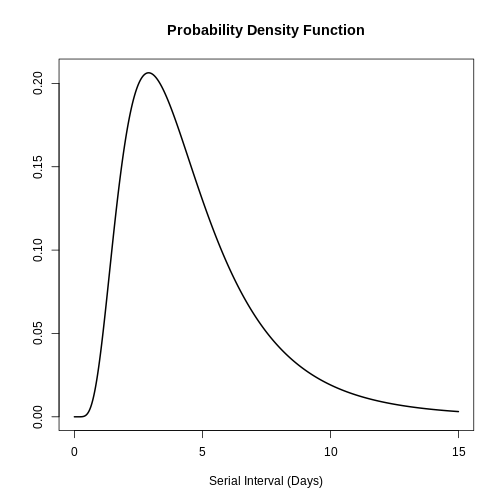
Puedes utilizar plot() para objetos
<epidist> para visualizarlos:
- la Función de densidad de probabilidad (PDF, por sus siglas en inglés) y
- la Función de distribución acumulativa (CDF, por sus siglas en inglés).
R
# plot <epidist> object
plot(covid_serialint)
Con el argumento day_range, puedes cambiar la duración o
el número de días del x eje. Explora cómo se ve esto:
R
# plot <epidist> object
plot(covid_serialint, day_range = 0:20)
Extrae las estadísticas de resumen
Podemos obtener la media o primediomean y la desviación
estándar(sd) a partir de <epidist>
accediendo al objetosummary_stats:
R
# get the mean
covid_serialint$summary_stats$mean
SALIDA
[1] 4.7¡Ahora tenemos un parámetro epidemiológico que podemos reutilizar!
Dado que el covid_serialint es una distribución log normal
lnorm o, podemos reemplazar las estadísticas de
resumen que introducimos en la función
EpiNow2::LogNormal()
R
generation_time <-
EpiNow2::LogNormal(
mean = covid_serialint$summary_stats$mean, # replaced!
sd = covid_serialint$summary_stats$sd, # replaced!
max = 20
)
En el próximo episodio aprenderemos a utilizar EpiNow2
para especificar correctamente las distribuciones y estimar la
transmisibilidad. Después, cómo utilizar funciones de
distribución para obtener un valor máximo (max)
para EpiNow2::LogNormal() y utilizar
{epiparameter} en tu análisis.
Distribuciones logarítmicas normales
Si necesitas los parámetros de la distribución log normal log
normales en lugar de las estadísticas de resumen, podemos
utilizar epiparameter::get_parameters():
R
covid_serialint_parameters <-
epiparameter::get_parameters(covid_serialint)
covid_serialint_parameters
SALIDA
meanlog sdlog
1.3862617 0.5679803 Se obtiene un vector de clase <numeric> ¡listo
para usar como entrada para cualquier otro paquete!
Desafíos
Intervalo serial del ébola
Tómate 1 minuto para
Obtener acceso al intervalo serial de ébola con el mayor tamaño de muestra.
Responde:
¿Qué es el
sdde la distribución epidemiológica?¿Cuál es el tamaño de muestra
sample_sizeutilizado en ese estudio?
R
# ebola serial interval
ebola_serial <-
epiparameter::epidist_db(
disease = "ebola",
epi_dist = "serial",
single_epidist = TRUE
)
SALIDA
Using WHO Ebola Response Team, Agua-Agum J, Ariyarajah A, Aylward B, Blake I,
Brennan R, Cori A, Donnelly C, Dorigatti I, Dye C, Eckmanns T, Ferguson
N, Formenty P, Fraser C, Garcia E, Garske T, Hinsley W, Holmes D,
Hugonnet S, Iyengar S, Jombart T, Krishnan R, Meijers S, Mills H,
Mohamed Y, Nedjati-Gilani G, Newton E, Nouvellet P, Pelletier L,
Perkins D, Riley S, Sagrado M, Schnitzler J, Schumacher D, Shah A, Van
Kerkhove M, Varsaneux O, Kannangarage N (2015). "West African Ebola
Epidemic after One Year — Slowing but Not Yet under Control." _The New
England Journal of Medicine_. doi:10.1056/NEJMc1414992
<https://doi.org/10.1056/NEJMc1414992>..
To retrieve the citation use the 'get_citation' functionR
ebola_serial
SALIDA
Disease: Ebola Virus Disease
Pathogen: Ebola Virus
Epi Distribution: serial interval
Study: WHO Ebola Response Team, Agua-Agum J, Ariyarajah A, Aylward B, Blake I,
Brennan R, Cori A, Donnelly C, Dorigatti I, Dye C, Eckmanns T, Ferguson
N, Formenty P, Fraser C, Garcia E, Garske T, Hinsley W, Holmes D,
Hugonnet S, Iyengar S, Jombart T, Krishnan R, Meijers S, Mills H,
Mohamed Y, Nedjati-Gilani G, Newton E, Nouvellet P, Pelletier L,
Perkins D, Riley S, Sagrado M, Schnitzler J, Schumacher D, Shah A, Van
Kerkhove M, Varsaneux O, Kannangarage N (2015). "West African Ebola
Epidemic after One Year — Slowing but Not Yet under Control." _The New
England Journal of Medicine_. doi:10.1056/NEJMc1414992
<https://doi.org/10.1056/NEJMc1414992>.
Distribution: gamma
Parameters:
shape: 2.188
scale: 6.490R
# get the sd
ebola_serial$summary_stats$sd
SALIDA
[1] 9.6R
# get the sample_size
ebola_serial$metadata$sample_size
SALIDA
[1] 305Intenta visualizar esta distribución utilizando
plot().
Explora también todos los demás elementos anidados dentro del objecto
<epidist> .
Comparte sobre:
- ¿Qué elementos encuentras útiles para tu análisis?
- ¿Qué otros elementos te gustaría ver en este objeto? ¿Cómo?
Un elemento interesante del contenido anidado es el
method_assess, que se refiere a los métodos utilizados por
los autores del estudio para evaluar el sesgo al estimar la distribución
del intervalo serial.
R
covid_serialint$method_assess
SALIDA
$censored
[1] TRUE
$right_truncated
[1] TRUE
$phase_bias_adjusted
[1] FALSE¡Exploraremos estos conceptos en los siguientes episodios!
Parámetro de severidad del ébola
Un parámetro de severidad como la duración de la hospitalización podría añadir información necesaria sobre la capacidad de camas en respuesta a un brote (Cori et al., 2017).
Para el ébola:
- ¿Cuál es la estimación puntual reportada de la duración media de la asistencia sanitaria y el aislamiento de casos?
Un retraso informativo debe medir el tiempo transcurrido desde el inicio de los síntomas hasta la recuperación o la muerte.
Encuentra una forma de acceder a toda la base de datos
{epiparameter} y averigua cómo se puede almacenar ese
retraso. La salida de parameter_tbl() es un dataframe o
tabla de datos.
R
# one way to get the list of all the available parameters
epidist_db(disease = "all") %>%
parameter_tbl() %>%
as_tibble() %>%
distinct(epi_distribution)
SALIDA
Returning 122 results that match the criteria (99 are parameterised).
Use subset to filter by entry variables or single_epidist to return a single entry.
To retrieve the citation for each use the 'get_citation' functionSALIDA
# A tibble: 12 × 1
epi_distribution
<chr>
1 incubation period
2 serial interval
3 generation time
4 onset to death
5 offspring distribution
6 hospitalisation to death
7 hospitalisation to discharge
8 notification to death
9 notification to discharge
10 onset to discharge
11 onset to hospitalisation
12 onset to ventilation R
ebola_severity <- epidist_db(
disease = "ebola",
epi_dist = "onset to discharge"
)
SALIDA
Returning 1 results that match the criteria (1 are parameterised).
Use subset to filter by entry variables or single_epidist to return a single entry.
To retrieve the citation for each use the 'get_citation' functionR
# point estimate
ebola_severity$summary_stats$mean
SALIDA
[1] 15.1Comprueba que para algunas entradas de {epiparameter}
también tendrás la incertidumbre en torno a las estimación
puntual de cada estadística de síntesis:
R
# 95% confidence intervals
ebola_severity$summary_stats$mean_ci
SALIDA
[1] 95R
# limits of the confidence intervals
ebola_severity$summary_stats$mean_ci_limits
SALIDA
[1] 14.6 15.6Un zoológico de distribuciones
¡Explora esta shinyapp llamada The distribution zoo !
Sigue estos pasos para reproducir la forma de la distribución de
intervalo serial COVID desde {epiparameter}
(covid_serialint objeto):
- Accede al sitio web de shiny app https://ben18785.shinyapps.io/distribution-zoo/,
- Ve al panel izquierdo,
- Mantén pulsado el botón Categoría de distribución:
Continuous Univariate, - Selecciona un nuevo Tipo de distribución:
Log-Normal, - Mueve los controles deslizantes es decir, el
elemento de control gráfico que te permite ajustar un valor moviendo una
barra horizontal hacia la posición
covid_serialintparámetros.
Reprodúcelos con el botón distribution y todos sus
elementos de lista: [[2]], [[3]] y
[[4]]. Explora cómo cambia la forma de una distribución
cuando cambian sus parámetros.
Comparte sobre:
- ¿Qué otras funciones del sitio web te parecen útiles?
En el contexto de las interfaces de usuario y de las interfaces gráficas de usuario (GUI), como el Zoo de la Distribución una aplicación deslizador es un elemento de control gráfico que permite a los usuarios ajustar un valor moviendo la barra. Conceptualmente, proporciona una forma de seleccionar un valor numérico dentro de un rango especificado deslizando o arrastrando visualmente un puntero (el tirador) a lo largo de un eje continuo.
Puntos Clave
- Utiliza
{epiparameter}para acceder al catálogo de literatura sobre distribuciones epidemiológicas de retraso. - Utiliza
epidist_db()para seleccionar distribuciones de retraso individuales. - Utiliza
parameter_tbl()para obtener una visión general de las distribuciones de retardo múltiples. - Reutiliza las estimaciones conocidas para una enfermedad desconocida en la fase inicial de un brote cuando no se disponga de datos de rastreo de contactos.
