Función que genera el mapa por departamentos o municipios con el número de casos o la incidencia de una enfermedad o evento.
Usage
plot_map(
data_agrupada,
col_distribucion = "incidencia",
col_codigos = NULL,
fuente_data = NULL,
dpto = NULL,
mpio = NULL,
ruta_dir = NULL,
cache = FALSE
)Arguments
- data_agrupada
Un `data.frame` que contiene los datos de la enfermedad agrupados por departamento y número de casos.
- col_distribucion
Un `character` (cadena de caracteres) que contiene el nombre de la columna que tiene los valores de la distribución, ya sea por número de casos o incidencia; su valor por defecto es `"incidencia"`.
- col_codigos
Un `character` (cadena de caracteres) que contiene el nombre de la columna con los códigos de los departamentos o municipios, los cuales se utilizan para obtener los poligonos de las áreas geográficas del archivo geoespacial o Shapefile; su valor por defecto `NULL`.
- fuente_data
Un `character` (cadena de caracteres) que contiene la leyenda o fuente de información de los datos de la enfermedad o evento; su valor por defecto `NULL`.
- dpto
Un `character` (cadena de caracteres) que contiene el nombre del departamento; su valor por defecto `NULL`.
- mpio
Un `character` (cadena de caracteres) que contiene el nombre del municipio; su valor por defecto `NULL`.
- ruta_dir
Un `character` (cadena de caracteres) que contiene la ruta del directorio donde se almacenará el Shapefile del mapa de Colombia. Su valor por defecto es `NULL`.
- cache
Un `logical` (`TRUE` o `FALSE`) que indica si el Shapefile del mapa de Colombia debe ser almacenado en caché. Su valor por defecto es `FALSE`.
Value
Un `plot` o mapa por departamentos o municipios con el número de casos o incidencia de un evento o enfermedad específica.
Examples
# \donttest{
data(dengue2020)
data_limpia <- limpiar_data_sivigila(dengue2020)
data_estandar <- estandarizar_geo_cods(data_limpia)
# Mapa por departamentos
geo_ocurrencia <- obtener_tip_ocurren_geo(nombre_event = "dengue")
data_espacial <- agrupar_dpto(data_event = data_estandar,
geo_ocurrencia[1:4])
if (interactive()) {
plot_map(
data_agrupada = data_espacial,
col_distribucion = "casos",
cache = TRUE
)
}
# Mapa por municipios de un departamento especifico
data_filtrada_dpto <- geo_filtro(
data_event = data_estandar,
dpto = "Cundinamarca"
)
data_espacial_dpto <- agrupar_mpio(data_event = data_filtrada_dpto)
plot_map(
data_agrupada = data_espacial_dpto,
col_codigos = "cod_mun_o",
col_distribucion = "casos",
ruta_dir = tempdir()
)
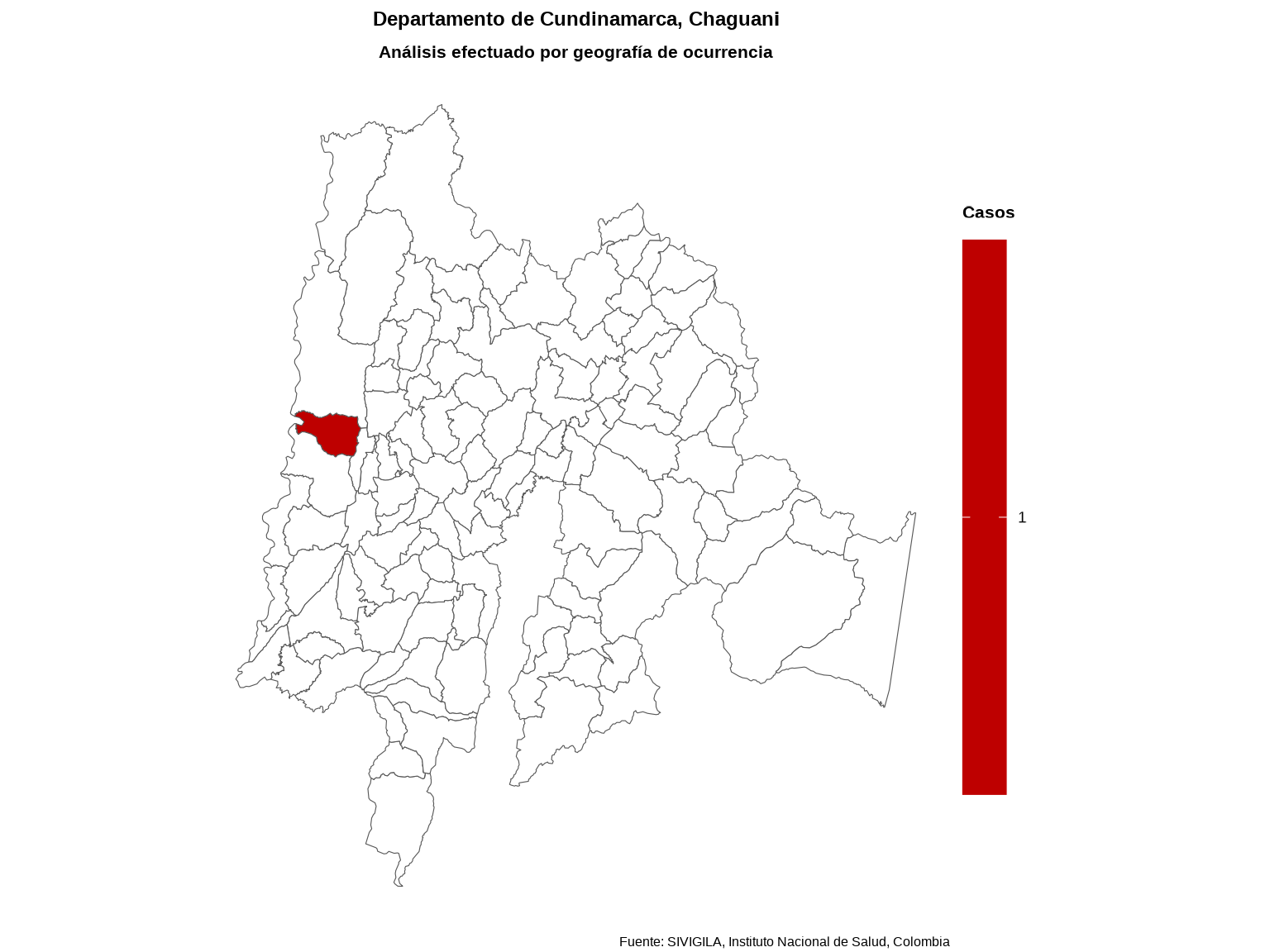 # Mapa por municipio especifico
data_filtrada_mpio <- geo_filtro(
data_event = data_estandar,
dpto = "Antioquia",
mpio = "Medellin"
)
data_espacial_mpio <- agrupar_mpio(data_event = data_filtrada_mpio)
if (interactive()) {
plot_map(
data_agrupada = data_espacial_mpio,
col_codigos = "cod_mun_o",
col_distribucion = "casos",
dpto = "Antioquia",
mpio = "Medellin",
cache = TRUE
)
}
# Mapa con la incidencia por municipios de un departamento específico
incidencia_dpto <-
calcular_incidencia_geo(data_agrupada = data_espacial_dpto,
ruta_dir = tempdir())
#> Las incidencias se calcularon con la poblacion a riesgo definida por el Ministerio de Salud para el 2020
plot_map(
data_agrupada = incidencia_dpto$data_incidencia,
col_codigos = "cod_mun_o",
col_distribucion = "incidencia",
ruta_dir = tempdir()
)
# Mapa por municipio especifico
data_filtrada_mpio <- geo_filtro(
data_event = data_estandar,
dpto = "Antioquia",
mpio = "Medellin"
)
data_espacial_mpio <- agrupar_mpio(data_event = data_filtrada_mpio)
if (interactive()) {
plot_map(
data_agrupada = data_espacial_mpio,
col_codigos = "cod_mun_o",
col_distribucion = "casos",
dpto = "Antioquia",
mpio = "Medellin",
cache = TRUE
)
}
# Mapa con la incidencia por municipios de un departamento específico
incidencia_dpto <-
calcular_incidencia_geo(data_agrupada = data_espacial_dpto,
ruta_dir = tempdir())
#> Las incidencias se calcularon con la poblacion a riesgo definida por el Ministerio de Salud para el 2020
plot_map(
data_agrupada = incidencia_dpto$data_incidencia,
col_codigos = "cod_mun_o",
col_distribucion = "incidencia",
ruta_dir = tempdir()
)
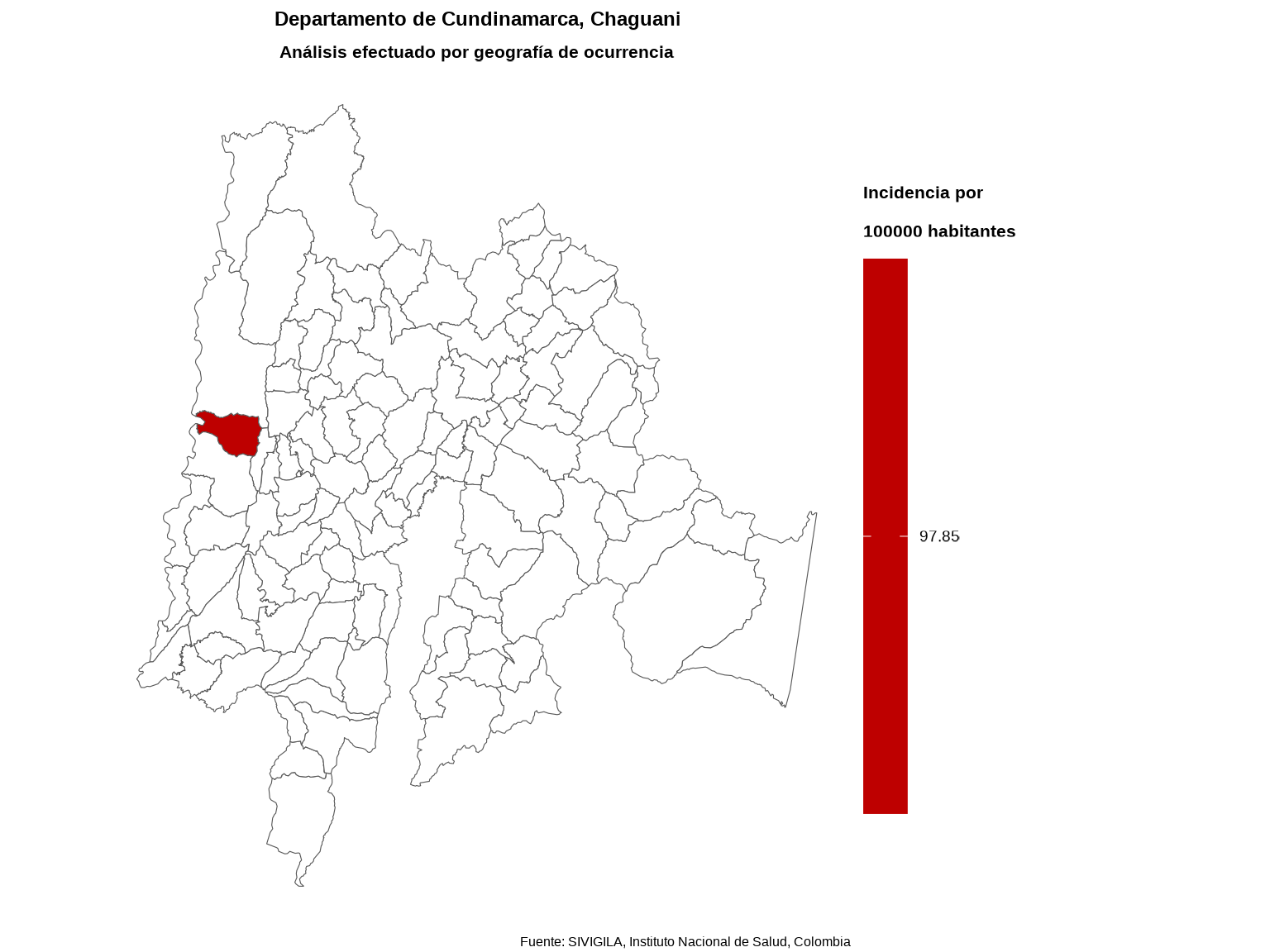 # }
# }