Accéder aux distribution des délais épidémiologiques
Dernière mise à jour le 2025-11-11 | Modifier cette page
Vue d'ensemble
Questions
- Comment accéder aux distributions des délais des maladies à partir d’une base de données préétablie pour les utiliser dans l’analyse ?
Objectifs
- Obtenez les délais à partir d’une base de données de recherche documentaire avec epiparameter.
- Obtenez les paramètres de distribution et les statistiques sommaires des distributions de délais.
Conditions préalables
Cet épisode nécessite que vous soyez familier avec :
la science des données Programmation de base avec R.
Théorie des épidémies La théorie des épidémies est basée sur les paramètres épidémiologiques, les périodes de la maladie, telles que la période d’incubation, le temps de génération et l’intervalle sériel.
Introduction
Les maladies infectieuses suivent un cycle infectieux qui comprend généralement les phases suivantes : période présymptomatique, période symptomatique et période de guérison, comme le décrivent leurs histoire naturelle. Ces périodes peuvent être utilisées pour comprendre la dynamique de la transmission et informer les interventions de prévention et de contrôle des maladies.
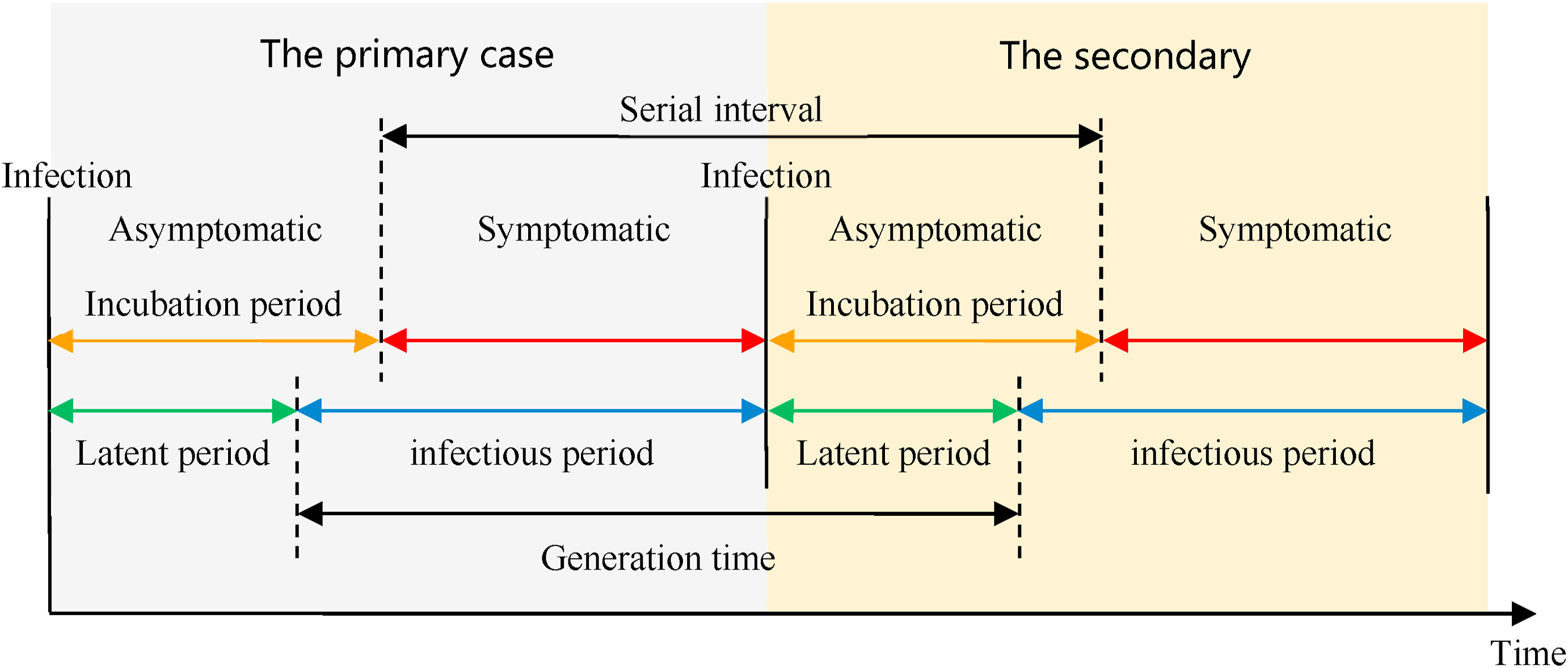
Définitions
Regardez le glossaire pour connaître les définitions de toutes les périodes de la figure ci-dessus !
Toutefois, au début d’une épidémie, les efforts visant à comprendre
l’épidémie et les implications pour la lutte peuvent être retardés par
l’absence d’un moyen facile d’accéder aux paramètres clés de la maladie
en question (Nash et al.,
2023). Des projets tels que epiparameter et
{epireview} construisent des catalogues en ligne en suivant
des protocoles de synthèse de la littérature qui peuvent aider à
informer l’analyse et à paramétrer les modèles en fournissant une
bibliothèque de paramètres épidémiologiques précédemment estimés à
partir d’épidémies passées.
Pour illustrer l’utilisation de l’outil epiparameter R dans votre pipeline d’analyse, notre objectif dans cet épisode sera d’accéder à un ensemble spécifique de paramètres épidémiologiques de la littérature, au lieu d’extraire des articles et de les copier-coller à la main. Nous les insérerons ensuite dans un EpiNow2 flux de travail d’analyse.
Commençons par charger le fichier epiparameter
paquetage. Nous utiliserons le tube %>% pour connecter
certaines de ses fonctions, certaines tibble et
dplyr donc appelons aussi à la fonction
tidyverse paquetage :
R
library(epiparameter)
library(tidyverse)
Le double point-virgule
Le double point-virgule :: dans R vous permet d’appeler
une fonction spécifique d’un paquetage sans charger l’ensemble du
paquetage dans l’environnement actuel.
Par exemple, vous pouvez appeler une fonction spécifique d’un package
sans charger le package entier dans l’environnement actuel,
dplyr::filter(data, condition) utilise
filter() à partir de l’outil dplyr
paquet.
Cela nous permet de nous souvenir des fonctions du paquet et d’éviter les conflits d’espace de noms.
Le problème
Si nous voulons estimer la transmissibilité d’une infection, il est
courant d’utiliser un package tel que EpiEstim ou
EpiNow2. Le paquet EpiEstim permet
d’estimer en temps réel le nombre de reproductions à l’aide des données
relatives aux cas dans le temps, reflétant ainsi l’évolution de la
transmission en fonction de la date d’apparition des symptômes. Pour
estimer la transmission en fonction du moment où les personnes ont été
effectivement infectées (plutôt que de l’apparition des symptômes), le
logiciel EpiNow2 étend cette idée en la combinant avec un
modèle qui tient compte des délais dans les données observées. Les deux
logiciels requièrent certaines informations épidémiologiques en entrée.
Par exemple, dans EpiNow2 nous utilisons
EpiNow2::Gamma() pour spécifier un temps de génération sous la
forme d’une distribution de probabilité ajoutant son mean
l’écart-type (sd) et sa valeur maximale
(max).
Pour spécifier une valeur generation_time qui suit un
Gamma avec une moyenne \(\mu =
4\) écart-type \(\sigma = 2\) et
une valeur maximale de 20, nous écrivons :
R
generation_time <-
EpiNow2::Gamma(
mean = 4,
sd = 2,
max = 20
)
Il est courant que les analystes recherchent manuellement la littérature disponible et copient et collent les résultats de la recherche. statistiques sommaires ou les paramètres de distribution des publications scientifiques. Une difficulté à laquelle on est souvent confronté est que la présentation des différentes distributions statistiques n’est pas cohérente dans la littérature (par exemple, un article peut ne présenter que la moyenne, plutôt que la distribution sous-jacente complète). epiparameter L’objectif du projet est de faciliter l’accès à des estimations fiables des paramètres de distribution pour une série de maladies infectieuses, afin qu’elles puissent être facilement mises en œuvre dans les pipelines d’analyse des épidémies.
Dans cet épisode, nous allons accéder les statistiques sommaires du temps de génération pour COVID-19 à partir de la bibliothèque de paramètres épidémiologiques fournie par l’Agence européenne pour la sécurité et la santé au travail (ESA). epiparameter. Ces paramètres peuvent être utilisés pour estimer la transmissibilité de cette maladie à l’aide de l’outil EpiNow2 dans les épisodes suivants.
Commençons par examiner le nombre d’entrées actuellement disponibles
dans la base de données des base de données des distributions
épidémiologiques en epiparameter en utilisant
epiparameter_db() pour la distribution épidémiologique
epi_name appelé temps de génération avec la chaîne
"generation":
R
epiparameter::epiparameter_db(
epi_name = "generation"
)
SORTIE
Returning 3 results that match the criteria (2 are parameterised).
Use subset to filter by entry variables or single_epiparameter to return a single entry.
To retrieve the citation for each use the 'get_citation' functionSORTIE
# List of 3 <epiparameter> objects
Number of diseases: 2
❯ Chikungunya ❯ Influenza
Number of epi parameters: 1
❯ generation time
[[1]]
Disease: Influenza
Pathogen: Influenza-A-H1N1
Epi Parameter: generation time
Study: Lessler J, Reich N, Cummings D, New York City Department of Health and
Mental Hygiene Swine Influenza Investigation Team (2009). "Outbreak of
2009 Pandemic Influenza A (H1N1) at a New York City School." _The New
England Journal of Medicine_. doi:10.1056/NEJMoa0906089
<https://doi.org/10.1056/NEJMoa0906089>.
Distribution: weibull (days)
Parameters:
shape: 2.360
scale: 3.180
[[2]]
Disease: Chikungunya
Pathogen: Chikungunya Virus
Epi Parameter: generation time
Study: Salje H, Cauchemez S, Alera M, Rodriguez-Barraquer I, Thaisomboonsuk B,
Srikiatkhachorn A, Lago C, Villa D, Klungthong C, Tac-An I, Fernandez
S, Velasco J, Roque Jr V, Nisalak A, Macareo L, Levy J, Cummings D,
Yoon I (2015). "Reconstruction of 60 Years of Chikungunya Epidemiology
in the Philippines Demonstrates Episodic and Focal Transmission." _The
Journal of Infectious Diseases_. doi:10.1093/infdis/jiv470
<https://doi.org/10.1093/infdis/jiv470>.
Parameters: <no parameters>
Mean: 14 (days)
[[3]]
Disease: Chikungunya
Pathogen: Chikungunya Virus
Epi Parameter: generation time
Study: Guzzetta G, Vairo F, Mammone A, Lanini S, Poletti P, Manica M, Rosa R,
Caputo B, Solimini A, della Torre A, Scognamiglio P, Zumla A, Ippolito
G, Merler S (2020). "Spatial modes for transmission of chikungunya
virus during a large chikungunya outbreak in Italy: a modeling
analysis." _BMC Medicine_. doi:10.1186/s12916-020-01674-y
<https://doi.org/10.1186/s12916-020-01674-y>.
Distribution: gamma (days)
Parameters:
shape: 8.633
scale: 1.447
# ℹ Use `parameter_tbl()` to see a summary table of the parameters.
# ℹ Explore database online at: https://epiverse-trace.github.io/epiparameter/articles/database.htmlDans la bibliothèque des paramètres épidémiologiques, on peut ne pas
disposer d’un "generation" pour la maladie qui nous
intéresse. À la place, nous pouvons consulter le serial
intervalles pour COVID-19. Voyons ce qu’il faut prendre en compte pour
cela !
Analyse systématique des données relatives aux agents pathogènes prioritaires
Les données de l’examen systématique des pathogènes prioritaires {epireview}
paquet R contient des paramètres sur Ebola, Marburg et Lassa issus
de revues systématiques récentes. D’autres agents pathogènes
prioritaires sont prévus pour les prochaines versions. Jetez un coup
d’œil à cette
vignette pour plus d’informations sur l’utilisation de ces
paramètres avec epiparameter.
Temps de génération vs intervalle série
Le temps de génération, conjointement avec le nombre de reproduction (\(R\)), peut fournir des indications précieuses sur le taux de croissance probable de l’épidémie et, partant, sur la mise en œuvre de mesuresde lutte. Plus la valeur de \(R\) est grande et/ou plus le temps de génération est court, plus le nombre de nouvelles infections attendues par jour est élevé, et donc plus l’incidence des cas de maladie augmentera rapidement.
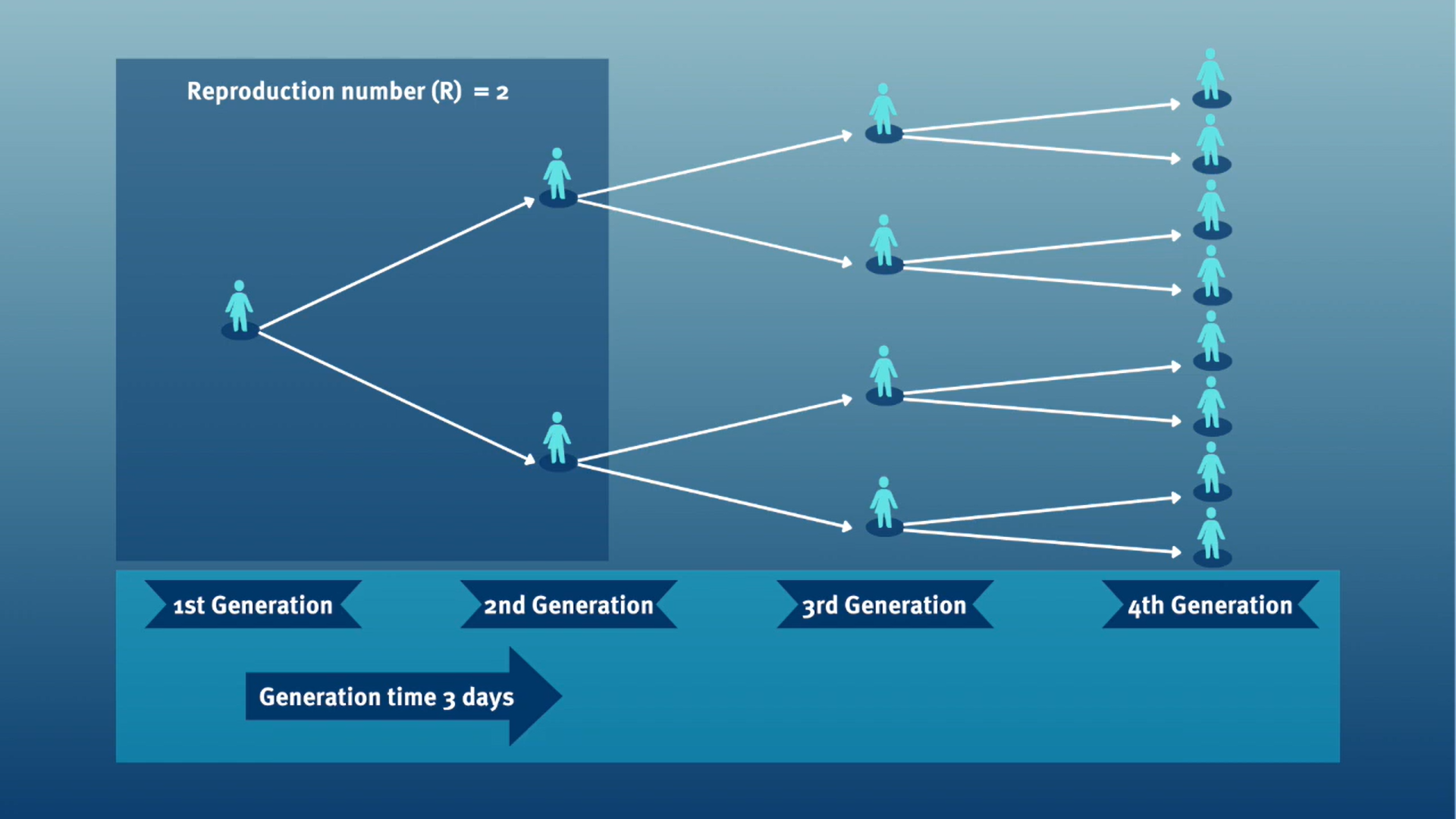
Pour calculer le nombre effectif de reproduction (\(R_{t}\)), le temps de génération (c’est-à-dire le délai entre une infection et la suivante) est souvent approximée par la distribution des temps de génération. intervalle sériel (c’est-à-dire le délai entre l’apparition des symptômes chez l’infecteur et l’apparition des symptômes chez l’infecté). Cette approximation est fréquemment utilisée car il est plus facile d’observer et d’enregistrer l’apparition des symptômes que le moment exact de l’infection.
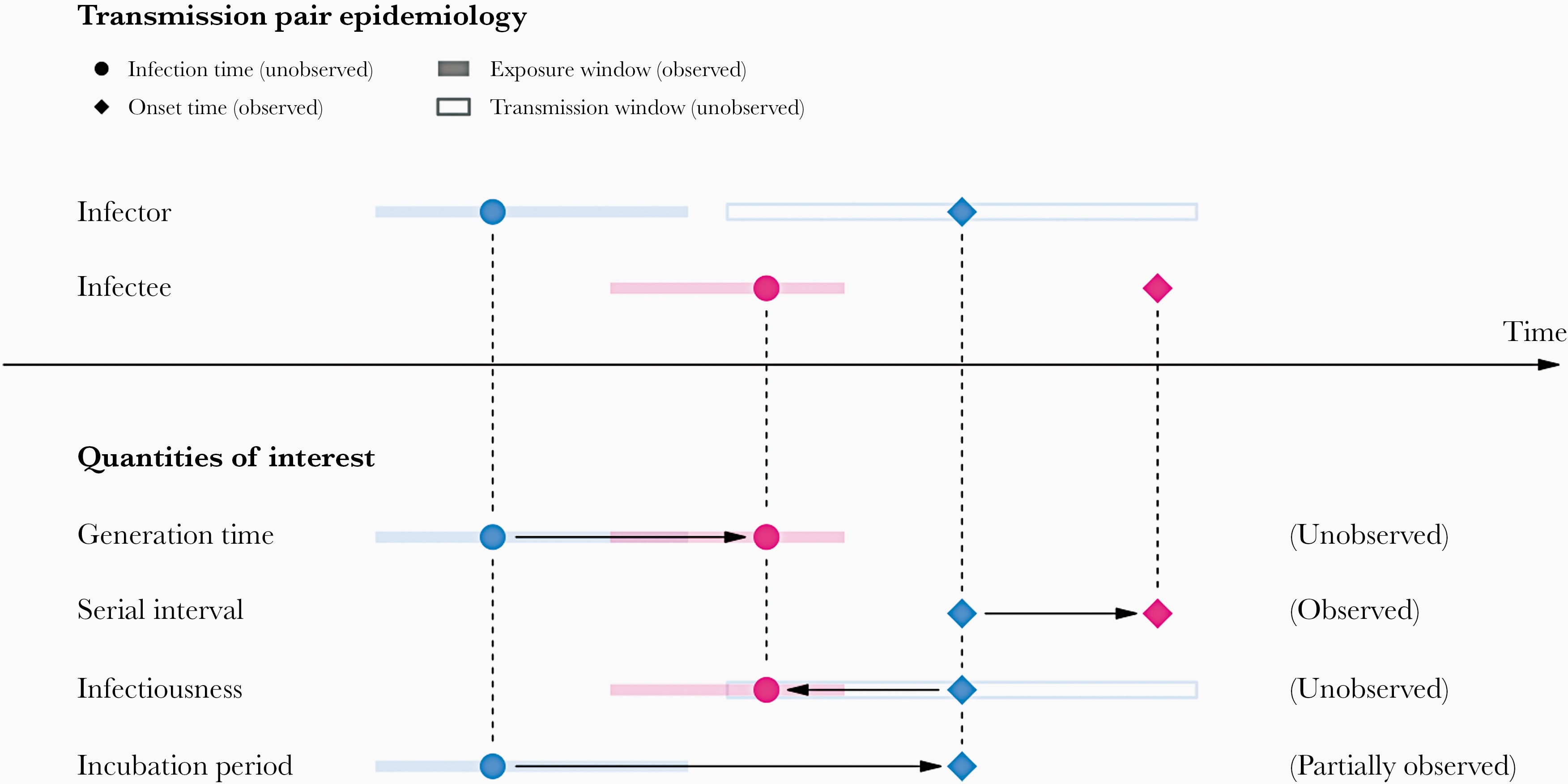
Cependant, l’utilisation de la intervalle sériel comme approximation de l’intervalle temps de génération est plus appropriée pour les maladies dans lesquelles l’infectiosité commence après l’apparition des symptômes (Chung Lau et al., 2021). Dans les cas où l’infectiosité commence avant l’apparition des symptômes, les intervalles sériels peuvent avoir des valeurs négatives, ce qui se produit lorsque l’infecté développe des symptômes avant l’infecteur dans une paire de transmission (Nishiura et al., 2020).
Des délais moyens aux distributions de probabilité
Si nous mesurons les intervalle sériel dans des données réelles, nous constatons généralement que toutes les paires de cas n’ont pas le même délai d’apparition. Nous pouvons également observer cette variabilité pour d’autres délais épidémiologiques clés, notamment le délai d’apparition de la maladie. la période d’incubation et période infectieuse.
Pour résumer ces données relatives aux périodes individuelles et aux paires, il est donc utile de quantifier les distribution statistique des délais qui correspondent le mieux aux données, plutôt que de se concentrer sur la moyenne (McFarland et al., 2023).
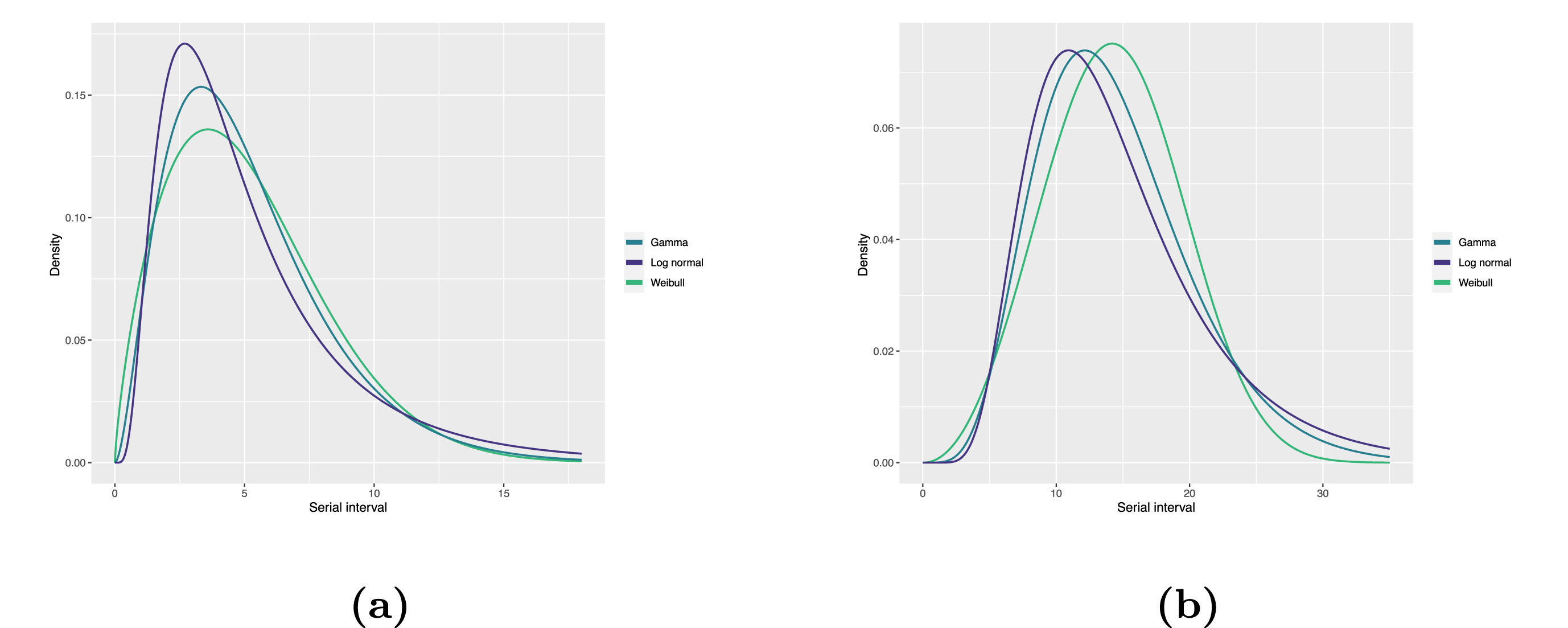
Les distributions statistiques sont résumées en fonction de leur statistiques sommaires comme l’emplacement (moyenne et percentiles) et l’étendue (variance ou écart-type) de la distribution, ou avec leur paramètres de distribution qui renseignent sur la forme (forme et taux/échelle) de la distribution. Ces valeurs estimées peuvent être rapportées avec leur incertitude (intervalles de confiance à 95 %).
| Gamma | moyenne | forme | taux/échelle |
|---|---|---|---|
| MERS-CoV | 14.13(13.9-14.7) | 6.31(4.88-8.52) | 0.43(0.33-0.60) |
| COVID-19 | 5.1(5.0-5.5) | 2.77(2.09-3.88) | 0.53(0.38-0.76) |
| Weibull | moyenne | forme | taux/échelle |
|---|---|---|---|
| MERS-CoV | 14.2(13.3-15.2) | 3.07(2.64-3.63) | 16.1(15.0-17.1) |
| COVID-19 | 5.2(4.6-5.9) | 1.74(1.46-2.11) | 5.83(5.08-6.67) |
| Log normal | moyenne | moyenne-log | sd-log |
|---|---|---|---|
| MERS-CoV | 14.08(13.1-15.2) | 2.58(2.50-2.68) | 0.44(0.39-0.5) |
| COVID-19 | 5.2(4.2-6.5) | 1.45(1.31-1.61) | 0.63(0.54-0.74) |
Tableau : Estimations des intervalles de série à l’aide des distributions Gamma, Weibull et Log Normal. Les intervalles de confiance à 95 % pour les paramètres de forme et d’échelle (logmoy et sd pour Log Normal) sont indiqués entre parenthèses (Althobaity et al, 2022).
Intervalle de série
Supposons que COVID-19 et le SRAS aient des valeurs de nombre de reproduction similaires et que l’intervalle de série soit proche du temps de génération.
Étant donné l’intervalle sériel des deux infections dans la figure ci-dessous :
- Laquelle serait la plus difficile à contrôler ?
- Pourquoi en concluez-vous ainsi ?
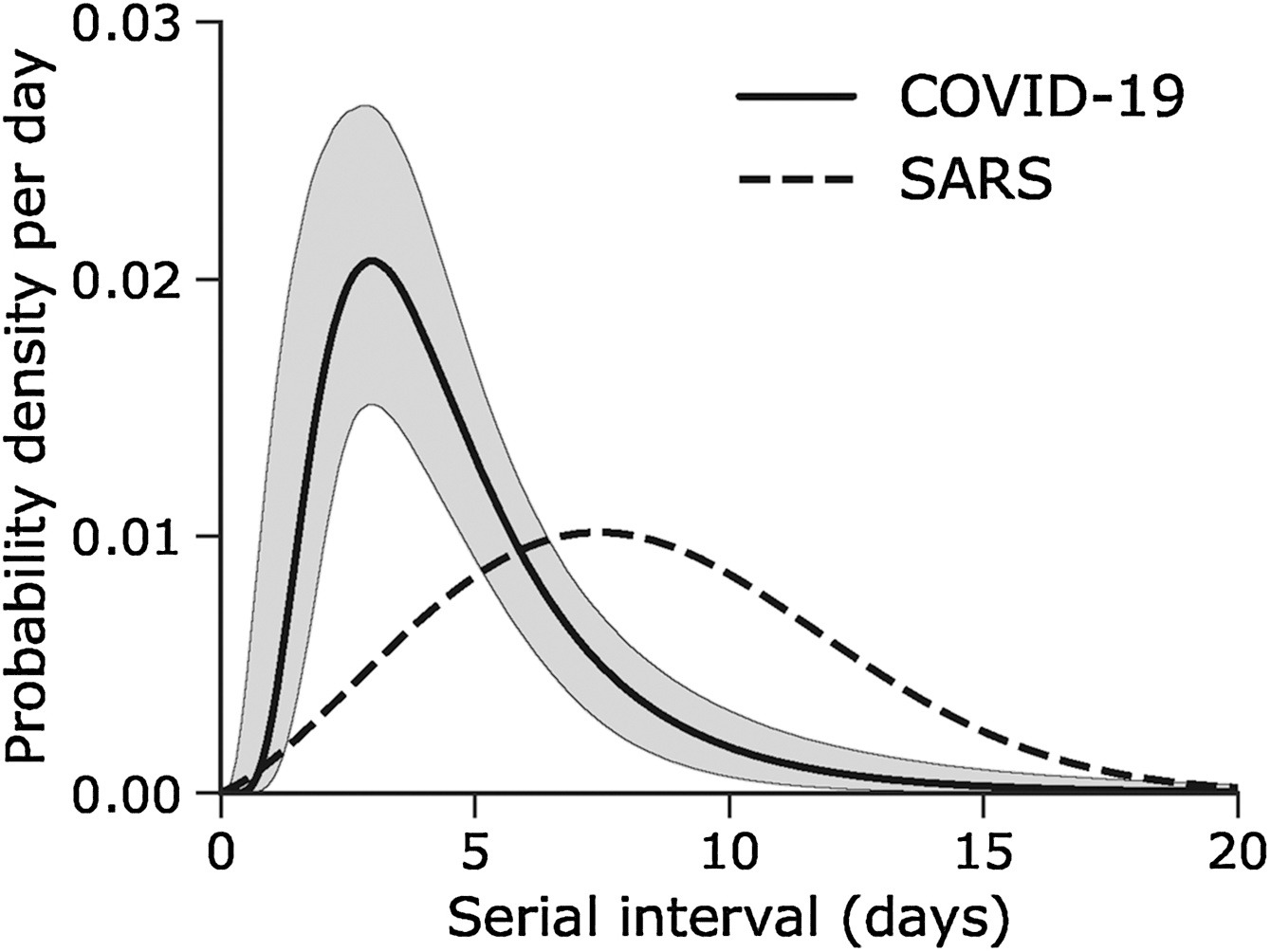
Le sommet de chaque courbe peut vous renseigner sur l’emplacement de la moyenne de chaque distribution. Une moyenne plus élevée indique un délai plus long entre l’apparition des symptômes chez l’infecteur et l’infecté.
Laquelle serait la plus difficile à contrôler ?
COVID-19
Pourquoi concluez-vous cela ?
L’intervalle sériel moyen de COVID-19 est plus faible. La valeur moyenne approximative de l’intervalle sériel de COVID-19 est d’environ quatre jours, alors que celle du SRAS est d’environ sept jours. Par conséquent, s’il y a beaucoup d’infections dans la population, COVID-19 produira en moyenne plus de nouvelles générations d’infections en moins de temps que le SRAS, en supposant des nombres de reproduction similaires. Cela signifie qu’il faudrait beaucoup plus de ressources pour lutter contre l’épidémie.
Choix des paramètres épidémiologiques
Dans cette section, nous utiliserons epiparameter pour obtenir l’intervalle sériel de COVID-19, comme alternative au temps de génération.
Tout d’abord, voyons combien de paramètres nous avons dans la base de
données des distributions épidémiologiques
(epiparameter_db()) avec l’option disease
nommé covid-19. Exécutez ce code :
R
epiparameter::epiparameter_db(
disease = "covid"
)
A partir du epiparameter nous pouvons utiliser le
paquet epiparameter_db() pour demander n’importe quel
disease ainsi qu’une distribution épidémiologique
spécifique (epi_name). Exécutez cette fonction dans votre
console :
R
epiparameter::epiparameter_db(
disease = "COVID",
epi_name = "serial"
)
Avec cette combinaison de requêtes, nous obtenons plus d’une
distribution des délais (parce que la base de données a plusieurs
entrées). Cette sortie est un <epiparameter> objet de
classe.
INSENSIBLE À LA CASSE
epiparameter_db est insensible
à la casse. Cela signifie que vous pouvez utiliser des chaînes avec
des lettres en majuscules ou en minuscules indistinctement. Des chaînes
comme "serial", "serial interval" ou
"serial_interval" sont également valables.
Comme le suggèrent les résultats, pour résumer une
<epiparameter> et obtenir les noms des colonnes de la
base de données de paramètres sous-jacente, nous pouvons ajouter
l’élément epiparameter::parameter_tbl() au code précédent à
l’aide du tuyau %>%:
R
epiparameter::epiparameter_db(
disease = "covid",
epi_name = "serial"
) %>%
epiparameter::parameter_tbl()
SORTIE
Returning 4 results that match the criteria (3 are parameterised).
Use subset to filter by entry variables or single_epiparameter to return a single entry.
To retrieve the citation for each use the 'get_citation' functionSORTIE
# Parameter table:
# A data frame: 4 × 7
disease pathogen epi_name prob_distribution author year sample_size
<chr> <chr> <chr> <chr> <chr> <dbl> <dbl>
1 COVID-19 SARS-CoV-2 serial interval <NA> Alene… 2021 3924
2 COVID-19 SARS-CoV-2 serial interval lnorm Nishi… 2020 28
3 COVID-19 SARS-CoV-2 serial interval weibull Nishi… 2020 18
4 COVID-19 SARS-CoV-2 serial interval norm Yang … 2020 131Dans le epiparameter::parameter_tbl() nous pouvons
également trouver différents types de distributions de probabilité (par
exemple, Log-normal, Weibull, Normal).
epiparameter utilise la fonction base R
pour les distributions. C’est pourquoi Log normal
s’appelle lnorm.
Les entrées avec une valeur manquante (<NA>) dans
le prob_distribution sont non paramétrés non
paramétrées. Elles présentent des statistiques sommaires (par exemple,
une moyenne et un écart type), mais aucune distribution de probabilité
n’est spécifiée. Comparez ces deux résultats :
R
# get an <epiparameter> object
distribution <-
epiparameter::epiparameter_db(
disease = "covid",
epi_name = "serial"
)
distribution %>%
# pluck the first entry in the object class <list>
pluck(1) %>%
# check if <epiparameter> object have distribution parameters
is_parameterised()
# check if the second <epiparameter> object
# have distribution parameters
distribution %>%
pluck(2) %>%
is_parameterised()
Les entrées paramétrées ont une méthode d’inférence
Comme indiqué dans ?is_parameterised une distribution
paramétrée est l’entrée à laquelle est associée une distribution de
probabilité fournie par une méthode d’inférence.
inference_method comme indiqué dans
metadata:
R
distribution[[1]]$metadata$inference_method
distribution[[2]]$metadata$inference_method
distribution[[4]]$metadata$inference_method
Trouvez vos distributions de délais !
Prenez 2 minutes pour explorer les epiparameter bibliothèque.
Choisissez une maladie d’intérêt (par exemple, la grippe, la rougeole, etc.) et une distribution des délais (par exemple, la période d’incubation, le début de la maladie jusqu’au décès, etc.)
Trouvez :
Combien y a-t-il de distributions de délais pour cette maladie ?
Combien de types de distribution de probabilité (par exemple, gamma, log normale) y a-t-il pour un délai donné dans cette maladie ?
Posez la question :
Reconnaissez-vous les journaux ?
La revue de littérature d’{epiparameter} devrait- elle prendre en compte un autre article?
L’analyse epiparameter_db() fonction avec
disease compte à elle seule le nombre d’entrées comme :
- études, et
- les répartitions des délais.
Les epiparameter_db() avec disease et
epi_name obtient une liste de toutes les entrées avec :
- la citation complète,
- le type d’une distribution de probabilité, et
- les valeurs des paramètres de la distribution.
La combinaison de epiparameter_db() plus
parameter_tbl() permet d’obtenir un cadre de données de
toutes les entrées avec des colonnes comme :
- les type de la distribution de probabilité par délai, et
- l’auteur et l’année de l’étude.
Nous avons choisi d’explorer les distributions des délais d’Ebola :
R
# we expect 16 delay distributions for Ebola
epiparameter::epiparameter_db(
disease = "ebola"
)
SORTIE
Returning 17 results that match the criteria (17 are parameterised).
Use subset to filter by entry variables or single_epiparameter to return a single entry.
To retrieve the citation for each use the 'get_citation' functionSORTIE
# List of 17 <epiparameter> objects
Number of diseases: 1
❯ Ebola Virus Disease
Number of epi parameters: 9
❯ hospitalisation to death ❯ hospitalisation to discharge ❯ incubation period ❯ notification to death ❯ notification to discharge ❯ offspring distribution ❯ onset to death ❯ onset to discharge ❯ serial interval
[[1]]
Disease: Ebola Virus Disease
Pathogen: Ebola Virus
Epi Parameter: offspring distribution
Study: Lloyd-Smith J, Schreiber S, Kopp P, Getz W (2005). "Superspreading and
the effect of individual variation on disease emergence." _Nature_.
doi:10.1038/nature04153 <https://doi.org/10.1038/nature04153>.
Distribution: nbinom (No units)
Parameters:
mean: 1.500
dispersion: 5.100
[[2]]
Disease: Ebola Virus Disease
Pathogen: Ebola Virus-Zaire Subtype
Epi Parameter: incubation period
Study: Eichner M, Dowell S, Firese N (2011). "Incubation period of ebola
hemorrhagic virus subtype zaire." _Osong Public Health and Research
Perspectives_. doi:10.1016/j.phrp.2011.04.001
<https://doi.org/10.1016/j.phrp.2011.04.001>.
Distribution: lnorm (days)
Parameters:
meanlog: 2.487
sdlog: 0.330
[[3]]
Disease: Ebola Virus Disease
Pathogen: Ebola Virus-Zaire Subtype
Epi Parameter: onset to death
Study: The Ebola Outbreak Epidemiology Team, Barry A, Ahuka-Mundeke S, Ali
Ahmed Y, Allarangar Y, Anoko J, Archer B, Abedi A, Bagaria J, Belizaire
M, Bhatia S, Bokenge T, Bruni E, Cori A, Dabire E, Diallo A, Diallo B,
Donnelly C, Dorigatti I, Dorji T, Waeber A, Fall I, Ferguson N,
FitzJohn R, Tengomo G, Formenty P, Forna A, Fortin A, Garske T,
Gaythorpe K, Gurry C, Hamblion E, Djingarey M, Haskew C, Hugonnet S,
Imai N, Impouma B, Kabongo G, Kalenga O, Kibangou E, Lee T, Lukoya C,
Ly O, Makiala-Mandanda S, Mamba A, Mbala-Kingebeni P, Mboussou F,
Mlanda T, Makuma V, Morgan O, Mulumba A, Kakoni P, Mukadi-Bamuleka D,
Muyembe J, Bathé N, Ndumbi Ngamala P, Ngom R, Ngoy G, Nouvellet P, Nsio
J, Ousman K, Peron E, Polonsky J, Ryan M, Touré A, Towner R, Tshapenda
G, Van De Weerdt R, Van Kerkhove M, Wendland A, Yao N, Yoti Z, Yuma E,
Kalambayi Kabamba G, Mwati J, Mbuy G, Lubula L, Mutombo A, Mavila O,
Lay Y, Kitenge E (2018). "Outbreak of Ebola virus disease in the
Democratic Republic of the Congo, April–May, 2018: an epidemiological
study." _The Lancet_. doi:10.1016/S0140-6736(18)31387-4
<https://doi.org/10.1016/S0140-6736%2818%2931387-4>.
Distribution: gamma (days)
Parameters:
shape: 2.400
scale: 3.333
# ℹ 14 more elements
# ℹ Use `print(n = ...)` to see more elements.
# ℹ Use `parameter_tbl()` to see a summary table of the parameters.
# ℹ Explore database online at: https://epiverse-trace.github.io/epiparameter/articles/database.htmlMaintenant, à partir de la sortie de
epiparameter::epiparameter_db() Quelle est la distribution de la
descendance?
Nous choisissons de trouver les périodes d’incubation d’Ebola. Cette sortie liste tous les articles et paramètres trouvés. Exécutez-la localement si nécessaire :
R
epiparameter::epiparameter_db(
disease = "ebola",
epi_name = "incubation"
)
Nous utilisons parameter_tbl() pour obtenir un
récapitulatif de toutes les données :
R
# we expect 2 different types of delay distributions
# for ebola incubation period
epiparameter::epiparameter_db(
disease = "ebola",
epi_name = "incubation"
) %>%
parameter_tbl()
SORTIE
Returning 5 results that match the criteria (5 are parameterised).
Use subset to filter by entry variables or single_epiparameter to return a single entry.
To retrieve the citation for each use the 'get_citation' functionSORTIE
# Parameter table:
# A data frame: 5 × 7
disease pathogen epi_name prob_distribution author year sample_size
<chr> <chr> <chr> <chr> <chr> <dbl> <dbl>
1 Ebola Virus Dise… Ebola V… incubat… lnorm Eichn… 2011 196
2 Ebola Virus Dise… Ebola V… incubat… gamma WHO E… 2015 1798
3 Ebola Virus Dise… Ebola V… incubat… gamma WHO E… 2015 49
4 Ebola Virus Dise… Ebola V… incubat… gamma WHO E… 2015 957
5 Ebola Virus Dise… Ebola V… incubat… gamma WHO E… 2015 792Nous trouvons deux types de distributions de probabilités pour cette requête : log normale et gamma.
Comment le epiparameter collecte-t-elle et examine-t-elle la littérature évaluée par les pairs ? Nous vous invitons à lire la vignette sur “Protocole de collecte et de synthèse des données!
Sélectionnez une distribution unique
Les epiparameter::epiparameter_db() fonctionne comme une
fonction de filtrage ou de sous-ensemble. Nous pouvons utiliser la
fonction author pour conserver
Hiroshi Nishiura ou l’argument subset pour
conserver les paramètres des études dont la taille de l’échantillon est
supérieure à 10 :
R
epiparameter::epiparameter_db(
disease = "covid",
epi_name = "serial",
author = "Nishiura",
subset = sample_size > 10
) %>%
epiparameter::parameter_tbl()
Nous obtenons toujours plus d’un paramètre épidémiologique. Au lieu
de cela, nous pouvons définir l’argument
single_epiparameter à TRUE pour n’en obtenir
qu’un seul :
R
epiparameter::epiparameter_db(
disease = "covid",
epi_name = "serial",
single_epiparameter = TRUE
)
SORTIE
Using Nishiura H, Linton N, Akhmetzhanov A (2020). "Serial interval of novel
coronavirus (COVID-19) infections." _International Journal of
Infectious Diseases_. doi:10.1016/j.ijid.2020.02.060
<https://doi.org/10.1016/j.ijid.2020.02.060>..
To retrieve the citation use the 'get_citation' functionSORTIE
Disease: COVID-19
Pathogen: SARS-CoV-2
Epi Parameter: serial interval
Study: Nishiura H, Linton N, Akhmetzhanov A (2020). "Serial interval of novel
coronavirus (COVID-19) infections." _International Journal of
Infectious Diseases_. doi:10.1016/j.ijid.2020.02.060
<https://doi.org/10.1016/j.ijid.2020.02.060>.
Distribution: lnorm (days)
Parameters:
meanlog: 1.386
sdlog: 0.568Attribuons cette <epiparameter> à l’objet de
classe covid_serialint objet.
R
covid_serialint <-
epiparameter::epiparameter_db(
disease = "covid",
epi_name = "serial",
single_epiparameter = TRUE
)
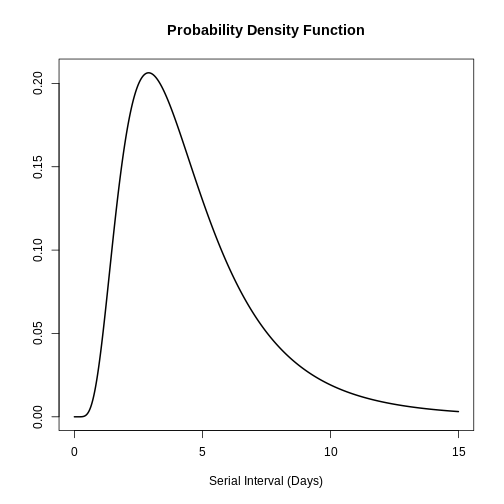
Vous pouvez utiliser plot() pour
<epiparameter> pour visualiser les objets :
- le Fonction de densité de probabilité (PDF) et
- la Fonction de distribution cumulative (FDC).
R
# plot <epiparameter> object
plot(covid_serialint)
Avec la xlim vous pouvez modifier la durée ou le nombre
de jours de la période d’essai. x de l’axe. Découvrez à
quoi cela ressemble :
R
# plot <epiparameter> object
plot(covid_serialint, xlim = c(1, 60))
Extrayez les statistiques récapitulatives
Nous pouvons obtenir les mean et l’écart-type
(sd) à partir de ce <epiparameter>
plonger dans le summary_stats l’objet :
R
# get the mean
covid_serialint$summary_stats$mean
SORTIE
[1] 4.7Nous avons maintenant un paramètre épidémiologique que nous pouvons
réutiliser ! Étant donné que l’objet covid_serialint est un
lnorm ou une distribution log-normale, nous pouvons
remplacer le statistiques sommaires que nous
introduisons dans la base de données EpiNow2::LogNormal()
dans la fonction
R
generation_time <-
EpiNow2::LogNormal(
mean = covid_serialint$summary_stats$mean, # replaced!
sd = covid_serialint$summary_stats$sd, # replaced!
max = 20
)
Dans le prochain épisode, nous apprendrons à utiliser la fonction
EpiNow2 pour spécifier correctement les distributions et
estimer la transmissibilité. Ensuite, comment utiliser fonctions
de distribution pour obtenir une valeur maximale
(max) pour EpiNow2::LogNormal() et utiliser
epiparameter dans votre analyse.
Distributions logarithmiques normales
Si vous avez besoin de la distribution log-normale de la
distribution logarithmique au lieu des statistiques sommaires,
vous pouvez utiliser epiparameter::get_parameters():
R
covid_serialint_parameters <-
epiparameter::get_parameters(covid_serialint)
covid_serialint_parameters
SORTIE
meanlog sdlog
1.3862617 0.5679803 Cela permet d’obtenir un vecteur de classe
<numeric> prêt à être utilisé comme entrée pour
n’importe quel autre paquet !
Considérez que {EpiNow2} acceptent également les paramètres de
distribution comme données d’entrée. Exécutez
?EpiNow2::LogNormal pour lire les Distributions
de probabilités de référence.
Défis
L’intervalle sériel d’Ebola
Prenez 1 minute pour :
Accédez à l’intervalle de série Ebola avec la taille d’échantillon la plus élevée.
Réponse :
Qu’est-ce que le
sdde la distribution épidémiologique ?Quelle est la
sample_sizeutilisée dans cette étude ?
Le paramètre de gravité d’Ebola
Un paramètre de gravité tel que la durée de l’hospitalisation pourrait compléter les informations nécessaires sur la capacité d’accueil en cas d’épidémie (Cori et al.).
Pour Ebola :
- Qu’est-ce qui est rapporté ? estimation ponctuelle de la durée moyenne des soins de santé et de l’isolement des cas ?
Un délai informatif devrait mesurer le temps écoulé entre l’apparition des symptômes et la guérison ou le décès.
Trouver un moyen d’accéder à l’ensemble epiparameter
base de données et de trouver comment ce délai peut être stocké. Les
parameter_tbl() est un tableau de données.
R
# one way to get the list of all the available parameters
epiparameter_db(disease = "all") %>%
parameter_tbl() %>%
as_tibble() %>%
distinct(epi_name)
SORTIE
Returning 125 results that match the criteria (100 are parameterised).
Use subset to filter by entry variables or single_epiparameter to return a single entry.
To retrieve the citation for each use the 'get_citation' functionSORTIE
# A tibble: 13 × 1
epi_name
<chr>
1 incubation period
2 serial interval
3 generation time
4 onset to death
5 offspring distribution
6 hospitalisation to death
7 hospitalisation to discharge
8 notification to death
9 notification to discharge
10 onset to discharge
11 onset to hospitalisation
12 onset to ventilation
13 case fatality risk R
ebola_severity <- epiparameter_db(
disease = "ebola",
epi_name = "onset to discharge"
)
SORTIE
Returning 1 results that match the criteria (1 are parameterised).
Use subset to filter by entry variables or single_epiparameter to return a single entry.
To retrieve the citation for each use the 'get_citation' functionR
# point estimate
ebola_severity$summary_stats$mean
SORTIE
[1] 15.1Vérifiez que pour certains epiparameter vous disposerez également de l’élément incertitude autour de la estimation ponctuelle de chaque statistique sommaire :
R
# 95% confidence intervals
ebola_severity$summary_stats$mean_ci
SORTIE
[1] 95R
# limits of the confidence intervals
ebola_severity$summary_stats$mean_ci_limits
SORTIE
[1] 14.6 15.6La distribution zoo
Explorez cette shinyapp appelée Le zoo de la distribution!
Suivez les étapes suivantes pour reproduire la forme de la
distribution d’intervalles sériels COVID à partir de
epiparameter (covid_serialint objet) :
- Accédez au https://ben18785.shinyapps.io/distribution-zoo/ site web de l’application shiny,
- Allez dans le panneau de gauche,
- Gardez l’option Catégorie de distribution:
Continuous Univariate, - Sélectionnez un nouveau Type de distribution:
Log-Normal, - Déplacez le curseurs c’est-à-dire l’élément de
contrôle graphique qui vous permet d’ajuster une valeur en déplaçant une
poignée le long d’une piste ou d’une barre horizontale jusqu’à
l’emplacement du curseur.
covid_serialintparamètres.
Reproduisez ces éléments à l’aide de l’outil
distribution et tous ses éléments de liste :
[[2]], [[3]] et [[4]]. Explorez
comment la forme d’une distribution change lorsque ses paramètres
changent.
Partagez à propos de :
- Quelles sont les autres fonctionnalités du site web que vous trouvez utiles ?
Points clés
- Utilisez epiparameter pour accéder au catalogue des distributions de délais épidémiologiques.
- Utilisez cette fonction pour accéder au catalogue de la littérature
sur les distributions de délais épidémiologiques.
epiparameter_db()pour sélectionner une seule distribution de délais. - Utilisez cette option pour sélectionner les distributions à délais
unique.
parameter_tbl()pour obtenir une vue d’ensemble des distributions de délais multiples. - Réutiliser les estimations connues pour une maladie inconnue au début d’une épidémie lorsqu’il n’y a pas de données sur la recherche des contacts.

Comment fonctionne “single_epiparameter” ?
En regardant la documentation d’aide pour
?epiparameter::epiparameter_db():single_epiparameter = TRUEalors l’entrée paramétrée<epiparameter>avec l’entrée la plus grande taille d’échantillon sera renvoyée.Qu’est-ce qu’un paramétré
<epiparameter>? Regardez?is_parameterised.