Quantifier la transmission
Dernière mise à jour le 2025-11-11 | Modifier cette page
Vue d'ensemble
Questions
- Comment puis-je estimer le nombre de reproduction variable dans le temps (\(Rt\)) et le taux de croissance à partir d’une série chronologique de données de cas ?
- Comment quantifier l’hétérogénéité géographique à partir de ces paramètres de transmission ?
Objectifs
- Apprenez à estimer les paramètres de transmission à partir d’une
série chronologique de données sur les cas à l’aide du package
EpiNow2.
Conditions préalables
Les étudiants doivent se familiariser avec les concepts suivants avant de suivre ce tutoriel :
Statistiques: distributions de probabilités, principe de l’analyse bayésienne.
Théorie des épidémies: Nombre de reproduction effectif.
Science des données: la transformation et la visualisation des données. Vous pouvez revoir l’épisode sur Agréger et visualiser les données d’incidence.
Rappel : le nombre de reproduction effectif, \(R_t\)
Le nombre de reproduction de base, \(R_0\) est le nombre moyen de cas causés par un individu infectieux dans une population entièrement susceptible.
Mais dans une épidémie en cours, la population ne reste pas entièrement susceptible, car les personnes qui se remettent de l’infection sont généralement immunisées. En outre, il peut y avoir des changements de comportement ou d’autres facteurs qui affectent la transmission. Lorsque nous voulons surveiller les changements dans la transmission, nous nous intéressons donc davantage à la valeur du nombre de reproduction effectif, \(R_t\) qui représente le nombre moyen de cas causés par un individu infectieux dans la population au temps \(t\) compte tenu de l’état actuel de la population (y compris les niveaux d’immunité et les mesures de contrôle).
Introduction
L’intensité de la transmission d’une épidémie est quantifiée à l’aide de deux paramètres clés : le nombre de reproduction, qui renseigne sur la force de la transmission en indiquant le nombre de nouveaux cas attendus pour chaque cas existant, et le nombre de décès. taux de croissance qui renseigne sur la vitesse de transmission en indiquant la rapidité avec laquelle l’épidémie se propage ou décline (temps de doublement/dédoublement) au sein d’une population. Pour plus de détails sur la distinction entre la vitesse et la force de la transmission et les implications pour la lutte, consultez le site Dushoff & Park, 2021.
Pour estimer ces paramètres clés à l’aide des données relatives aux cas, nous devons tenir compte des délais entre les dates d’infections et les dates de cas déclarés. Lors d’une épidémie, les données ne sont généralement disponibles qu’à partir des dates déclarées. Nous devons donc utiliser des méthodes d’estimation pour tenir compte de ces retards lorsque nous essayons de comprendre les changements dans la transmission au fil du temps.
Dans les prochains tutoriels, nous nous concentrerons sur la manière d’utiliser les fonctions de EpiNow2 pour estimer les paramètres de transmission des données de cas. Nous ne couvrirons pas le contexte théorique des modèles ou du cadre d’inférence, pour plus de détails sur ces concepts, consultez la vignette..
Dans ce tutoriel, nous allons apprendre à utiliser le package
EpiNow2 pour estimer le nombre de reproduction variable
dans le temps. Nous obtiendrons les données d’entrée de
incidence2. Nous utiliserons le package
tidyr et dplyr pour organiser certains de
ses résultats, ggplot2 pour visualiser la distribution
des cas, et le tuyau %>% pour relier certaines de leurs
fonctions, alors appelons aussi le package tidyverse
:
R
library(EpiNow2)
library(incidence2)
library(tidyverse)
Le double point-virgule
Le double deux-points :: dans R vous permet d’appeler
une fonction spécifique d’un package sans charger l’ensemble du package
dans l’environnement actuel.
Par exemple, vous pouvez appeler une fonction spécifique d’un package
sans charger le package entier dans l’environnement actuel,
dplyr::filter(data, condition) utilise
filter() à partir du package dplyr.
Cela nous permet de nous souvenir des fonctions du package et d’éviter les conflits d’espace de noms.
Inférence bayésienne
Le package R EpiNow2 utilise une méthode d’inférence
bayésienne inférence bayésienne
pour estimer les nombres de reproduction et les durées d’infection sur
la base des dates déclarées. En d’autres termes, il estime la
transmission en fonction de la date à laquelle les personnes ont été
effectivement infectées (plutôt que de l’apparition des symptômes), en
tenant compte des retards dans les données observées. En revanche,
l’approche EpiEstim permet une estimation en temps réel
plus rapide et plus simple du nombre de reproductions en utilisant
uniquement les données de cas au fil du temps, reflétant la façon dont
la transmission change en fonction du moment où les symptômes
apparaissent.
Dans l’inférence bayésienne, nous utilisons les connaissances a priori (distributions a priori) et les données (via une fonction de vraisemblance) pour obtenir la probabilité a posteriori :
\(\text{Probabilité a posteriori} \propto \text{vraisemblance} \times \text{probabilité a priori}\)
Distributions de délais et données de cas
Données de cas
Pour illustrer les fonctions des EpiNow2 nous
utiliserons les données relatives au début de la pandémie de COVID-19 au
Royaume-Uni, mais uniquement pour les 90 premiers jours observés. Les
données sont disponibles dans le package R
incidence2.
R
dplyr::as_tibble(incidence2::covidregionaldataUK)
SORTIE
# A tibble: 6,370 × 13
date region region_code cases_new cases_total deaths_new deaths_total
<date> <chr> <chr> <dbl> <dbl> <dbl> <dbl>
1 2020-01-30 East Mi… E12000004 NA NA NA NA
2 2020-01-30 East of… E12000006 NA NA NA NA
3 2020-01-30 England E92000001 2 2 NA NA
4 2020-01-30 London E12000007 NA NA NA NA
5 2020-01-30 North E… E12000001 NA NA NA NA
6 2020-01-30 North W… E12000002 NA NA NA NA
7 2020-01-30 Norther… N92000002 NA NA NA NA
8 2020-01-30 Scotland S92000003 NA NA NA NA
9 2020-01-30 South E… E12000008 NA NA NA NA
10 2020-01-30 South W… E12000009 NA NA NA NA
# ℹ 6,360 more rows
# ℹ 6 more variables: recovered_new <dbl>, recovered_total <dbl>,
# hosp_new <dbl>, hosp_total <dbl>, tested_new <dbl>, tested_total <dbl>Pour utiliser les données, nous devons les formater de manière à ce qu’elles comportent deux colonnes :
-
datela date (en tant qu’objet date, voir?is.Date()), -
confirm: nombre de cas déclarés (confirmés) à cette date.
Utilisons tidyr et incidence2 pour cela :
R
cases_sliced <- incidence2::covidregionaldataUK %>%
dplyr::as_tibble() %>%
# Preprocess missing values
tidyr::replace_na(base::list(cases_new = 0)) %>%
# Compute the daily incidence
incidence2::incidence(
date_index = "date",
counts = "cases_new",
count_values_to = "confirm",
date_names_to = "date",
complete_dates = TRUE
) %>%
# Keep the first 90 dates
dplyr::slice_head(n = 90)
Avec incidence2::incidence() nous agrégeons des cas sur
différentes périodes de temps (intervalles, c’est-à-dire des
jours, des semaines ou des mois) ou par groupe. Nous pouvons également
obtenir les dates complètes pour tous les intervalles de dates par
catégorie de groupe à l’aide de la fonction
complete_dates = TRUE Explorez plus tard les incidence2::incidence()
manuel de référence
Pouvons-nous reproduire {incidence2} avec {dplyr}?
Nous pouvons obtenir un objet similaire à cases à partir
de l’objet incidence2::covidregionaldataUK à l’aide du
package dplyr.
R
incidence2::covidregionaldataUK %>%
dplyr::select(date, cases_new) %>%
dplyr::group_by(date) %>%
dplyr::summarise(confirm = sum(cases_new, na.rm = TRUE)) %>%
dplyr::ungroup() %>%
dplyr::slice_head(n = 90)
Cependant, incidence2::incidence() contient des
arguments pratiques comme complete_dates qui facilitent
l’obtention d’un objet d’incidence avec le même intervalle de dates pour
chaque groupe sans nécessiter de lignes de code supplémentaires ou un
package de séries temporelles.
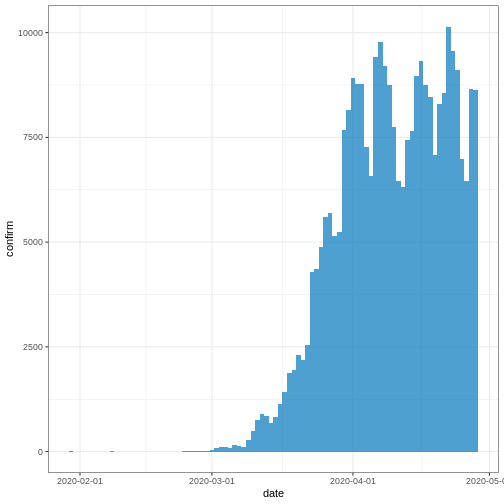
Dans une situation d’épidémie, il est probable que nous n’ayons accès qu’au début de l’ensemble des données d’entrée. Nous supposons donc que nous ne disposons que des 90 premiers jours de ces données.
R
plot(cases_sliced)
Pour transmettre les résultats de incidence2 à
EpiNow2, nous devons supprimer une colonne de l’objet
cases_filter :
R
# Drop column for {EpiNow2} input format
cases <- cases_sliced %>%
dplyr::select(-count_variable)
cases
SORTIE
# A tibble: 90 × 2
date confirm
<date> <dbl>
1 2020-01-30 3
2 2020-01-31 0
3 2020-02-01 0
4 2020-02-02 0
5 2020-02-03 0
6 2020-02-04 0
7 2020-02-05 2
8 2020-02-06 0
9 2020-02-07 0
10 2020-02-08 8
# ℹ 80 more rowsDistribution des délais
Nous supposons qu’il existe des délais entre le moment de l’infection et le moment où un cas est signalé. Nous spécifions ces délais sous forme de distributions pour tenir compte de l’incertitude des différences au niveau individuel. Le délai peut impliquer plusieurs types de processus. Un délai typique entre le moment de l’infection et la déclaration du cas peut consister en ce qui suit
le temps écoulé entre l’infection et l’apparition des symptômes (le période d’incubation) + délai entre l’apparition des symptômes et la notification du cas (délai de déclaration) .
La distribution des délais pour chacun de ces processus peut être estimée à partir de données ou obtenue à partir de la littérature. Nous pouvons exprimer l’incertitude quant aux paramètres corrects des distributions en supposant que les distributions ont fixe fixes ou qu’elles ont variables variables. Pour comprendre la différence entre fixe et variables considérons la période d’incubation.
Délais et données
Le nombre de retards et le type de retard sont des données flexibles qui dépendent des données. Les exemples ci-dessous montrent comment les délais peuvent être spécifiés pour différentes sources de données :
| Source de données | Délai (s) |
|---|---|
| Heure d’apparition des symptômes | Période d’incubation |
| Date de la déclaration du cas | Période d’incubation + délai entre l’apparition des symptômes et la notification du cas |
| Durée de l’hospitalisation | Période d’incubation + temps écoulé entre l’apparition des symptômes et l’hospitalisation |
Distribution de la période d’incubation
La distribution de la période d’incubation pour de nombreuses maladies peut généralement être obtenue à partir de la littérature. L’ensemble epiparameter contient une bibliothèque de paramètres épidémiologiques pour différentes maladies, obtenus à partir de la littérature.
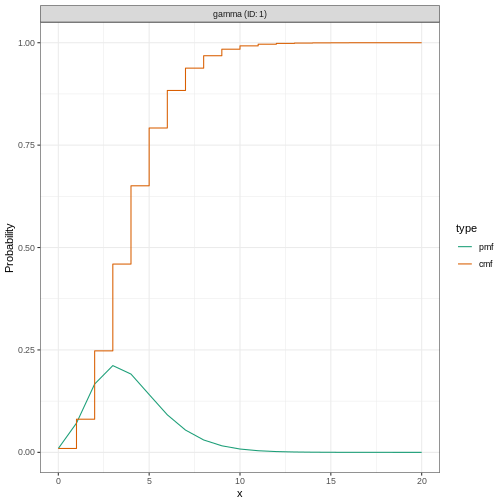
Nous spécifions une distribution gamma (fixe) avec moyenne \(\mu = 4\) et un écart-type \(\sigma = 2\) (forme = \(4\), échelle = \(1\)) en utilisant la fonction
Gamma() comme suit :
R
incubation_period_fixed <- EpiNow2::Gamma(
mean = 4,
sd = 2,
max = 20
)
incubation_period_fixed
SORTIE
- gamma distribution (max: 20):
shape:
4
rate:
1L’argument max est la valeur maximale que la
distribution peut prendre ; dans cet exemple, 20 jours.
Nous pouvons tracer les distributions générées par
EpiNow2 en utilisant plot().
R
plot(incubation_period_fixed)
Pourquoi une distribution gamma ?
La période d’incubation doit être une valeur positive. Nous devons donc spécifier une distribution en EpiNow2 qui ne concerne que les valeurs positives.
Gamma() prend en charge les distributions Gamma et
LogNormal() les distributions log-normales, qui sont des
distributions pour les valeurs positives uniquement.
Pour tous les types de retard, nous devrons utiliser des distributions pour les valeurs positives uniquement - nous ne voulons pas inclure les retards de jours négatifs dans notre analyse !
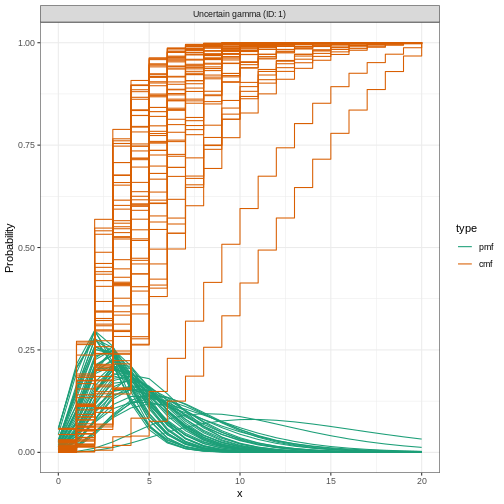
Prise en compte de l’incertitude de la distribution
Pour spécifier une distribution variable nous incluons l’incertitude autour de la moyenne \(\mu\) et de l’écart-type \(\sigma\) de notre distribution gamma. Si notre distribution de la période d’incubation a une moyenne \(\mu\) et un écart-type \(\sigma\) nous supposons que la moyenne (\(\mu\)) suit une distribution normale avec un écart type \(\sigma_{\mu}\):
\[\mbox{Normal}(\mu,\sigma_{\mu}^2)\]
et un écart-type (\(\sigma\)) suit une distribution normale avec un écart type \(\sigma_{\sigma}\):
\[\mbox{Normal}(\sigma,\sigma_{\sigma}^2).\]
Nous spécifions ceci en utilisant Normal() pour chaque
argument : la moyenne (\(\mu = 4\) avec
\(\sigma_{\mu} = 0.5\)) et l’écart-type
(\(\sigma = 2\) avec \(\sigma_{\sigma} = 0.5\)).
R
incubation_period_variable <- EpiNow2::Gamma(
mean = EpiNow2::Normal(mean = 4, sd = 0.5),
sd = EpiNow2::Normal(mean = 2, sd = 0.5),
max = 20
)
incubation_period_variable
SORTIE
- gamma distribution (max: 20):
shape:
- normal distribution:
mean:
4
sd:
0.61
rate:
- normal distribution:
mean:
1
sd:
0.31Traçons la distribution que nous venons de configurer :
R
plot(incubation_period_variable)
Retards dans l’établissement des rapports
Après la période d’incubation, il s’écoule un délai supplémentaire entre l’apparition des symptômes et la notification du cas : le délai de déclaration. Nous pouvons le spécifier comme une distribution fixe ou variable, ou estimer une distribution à partir de données.
Lorsque vous spécifiez une distribution, il est utile de visualiser la densité de probabilité pour voir le pic et l’étendue de la distribution. Dans cet example, nous allons utiliser une distribution log normale.
Si nous voulons supposer que le délai de déclaration moyen est de 2
jours (avec une incertitude de 0,5 jour) et que l’écart type est de 1
jour (avec une incertitude de 0,5 jour), nous pouvons spécifier une
distribution variable à l’aide de la commande LogNormal()
comme précédemment :
R
reporting_delay_variable <- EpiNow2::LogNormal(
meanlog = EpiNow2::Normal(mean = 2, sd = 0.5),
sdlog = EpiNow2::Normal(mean = 1, sd = 0.5),
max = 10
)
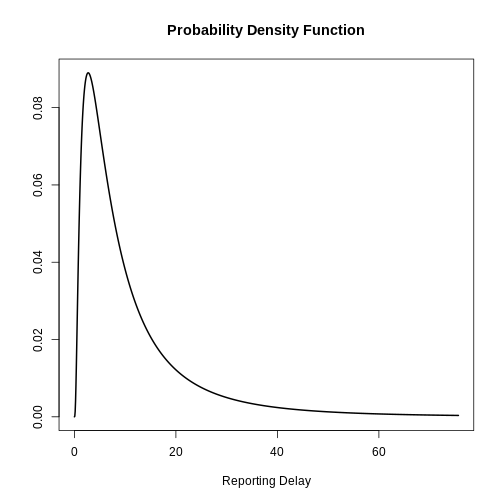
Visualisez une distribution log-normale en utilisant {epiparameter}
En utilisant epiparameter::epiparameter() nous pouvons
créer une distribution personnalisée. La distribution normale à
logarithme fixe aura l’aspect suivant :
R
library(epiparameter)
R
epiparameter::epiparameter(
disease = "covid",
epi_name = "reporting delay",
prob_distribution =
epiparameter::create_prob_distribution(
prob_distribution = "lnorm",
prob_distribution_params = c(
meanlog = 2,
sdlog = 1
)
)
) %>%
plot()
Comment obtenir le délai de reporting à partir des données ?
Si l’on dispose de données sur le délai entre l’apparition des
symptômes et la déclaration, on peut utiliser la fonction
estimate_delay() pour estimer une distribution log-normale
à partir d’un vecteur de délais. Le code ci-dessous illustre comment
utiliser la fonction estimate_delay() avec des données
synthétiques sur les délais.
R
library(tidyverse)
# Steps:
# - get Ebola data from package {outbreaks}
# - keep a subset of columns for this example only
# - calculate the time difference between two dates in linelist
# - extract the time difference as a vector class object
# - estimate the delay parameters using {EpiNow2}
outbreaks::ebola_sim_clean$linelist %>%
tibble::as_tibble() %>%
dplyr::select(case_id, date_of_onset, date_of_hospitalisation) %>%
dplyr::mutate(reporting_delay = date_of_hospitalisation - date_of_onset) %>%
dplyr::pull(reporting_delay) %>%
EpiNow2::estimate_delay(
samples = 1000,
bootstraps = 10
)
SORTIE
WARN [2025-11-11 01:32:57] dist_fit (chain: 1): The largest R-hat is 1.11, indicating chains have not mixed.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#r-hat -
WARN [2025-11-11 01:32:57] dist_fit (chain: 1): Bulk Effective Samples Size (ESS) is too low, indicating posterior means and medians may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#bulk-ess -
WARN [2025-11-11 01:32:57] dist_fit (chain: 1): Tail Effective Samples Size (ESS) is too low, indicating posterior variances and tail quantiles may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#tail-ess -
WARN [2025-11-11 01:33:00] dist_fit (chain: 1): The largest R-hat is 1.06, indicating chains have not mixed.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#r-hat -
WARN [2025-11-11 01:33:00] dist_fit (chain: 1): Bulk Effective Samples Size (ESS) is too low, indicating posterior means and medians may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#bulk-ess -
WARN [2025-11-11 01:33:00] dist_fit (chain: 1): Tail Effective Samples Size (ESS) is too low, indicating posterior variances and tail quantiles may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#tail-ess -
WARN [2025-11-11 01:33:02] dist_fit (chain: 1): Bulk Effective Samples Size (ESS) is too low, indicating posterior means and medians may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#bulk-ess -
WARN [2025-11-11 01:33:02] dist_fit (chain: 1): Tail Effective Samples Size (ESS) is too low, indicating posterior variances and tail quantiles may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#tail-ess -
WARN [2025-11-11 01:33:04] dist_fit (chain: 1): Bulk Effective Samples Size (ESS) is too low, indicating posterior means and medians may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#bulk-ess -
WARN [2025-11-11 01:33:04] dist_fit (chain: 1): Tail Effective Samples Size (ESS) is too low, indicating posterior variances and tail quantiles may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#tail-ess -
WARN [2025-11-11 01:33:06] dist_fit (chain: 1): The largest R-hat is 1.12, indicating chains have not mixed.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#r-hat -
WARN [2025-11-11 01:33:06] dist_fit (chain: 1): Bulk Effective Samples Size (ESS) is too low, indicating posterior means and medians may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#bulk-ess -
WARN [2025-11-11 01:33:06] dist_fit (chain: 1): Tail Effective Samples Size (ESS) is too low, indicating posterior variances and tail quantiles may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#tail-ess -
WARN [2025-11-11 01:33:08] dist_fit (chain: 1): The largest R-hat is 1.15, indicating chains have not mixed.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#r-hat -
WARN [2025-11-11 01:33:08] dist_fit (chain: 1): Bulk Effective Samples Size (ESS) is too low, indicating posterior means and medians may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#bulk-ess -
WARN [2025-11-11 01:33:08] dist_fit (chain: 1): Tail Effective Samples Size (ESS) is too low, indicating posterior variances and tail quantiles may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#tail-ess -
WARN [2025-11-11 01:33:11] dist_fit (chain: 1): The largest R-hat is 1.11, indicating chains have not mixed.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#r-hat -
WARN [2025-11-11 01:33:11] dist_fit (chain: 1): Bulk Effective Samples Size (ESS) is too low, indicating posterior means and medians may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#bulk-ess -
WARN [2025-11-11 01:33:11] dist_fit (chain: 1): Tail Effective Samples Size (ESS) is too low, indicating posterior variances and tail quantiles may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#tail-ess -
WARN [2025-11-11 01:33:13] dist_fit (chain: 1): The largest R-hat is 1.1, indicating chains have not mixed.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#r-hat -
WARN [2025-11-11 01:33:13] dist_fit (chain: 1): Bulk Effective Samples Size (ESS) is too low, indicating posterior means and medians may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#bulk-ess -
WARN [2025-11-11 01:33:13] dist_fit (chain: 1): Tail Effective Samples Size (ESS) is too low, indicating posterior variances and tail quantiles may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#tail-ess -
WARN [2025-11-11 01:33:15] dist_fit (chain: 1): Bulk Effective Samples Size (ESS) is too low, indicating posterior means and medians may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#bulk-ess -
WARN [2025-11-11 01:33:15] dist_fit (chain: 1): Tail Effective Samples Size (ESS) is too low, indicating posterior variances and tail quantiles may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#tail-ess -
WARN [2025-11-11 01:33:17] dist_fit (chain: 1): Bulk Effective Samples Size (ESS) is too low, indicating posterior means and medians may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#bulk-ess -
WARN [2025-11-11 01:33:17] dist_fit (chain: 1): Tail Effective Samples Size (ESS) is too low, indicating posterior variances and tail quantiles may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#tail-ess - SORTIE
- lognormal distribution (max: 22):
meanlog:
- normal distribution:
mean:
0.21
sd:
0.16
sdlog:
- normal distribution:
mean:
1
sd:
0.12Temps de génération
Nous devons également spécifier une distribution pour le temps de génération. Nous utiliserons ici une distribution log-normale avec une moyenne de 3,6 et un écart-type de 3,1 (Ganyani et al. 2020).
R
generation_time_variable <- EpiNow2::LogNormal(
mean = EpiNow2::Normal(mean = 3.6, sd = 0.5),
sd = EpiNow2::Normal(mean = 3.1, sd = 0.5),
max = 20
)
Trouver des estimations
La fonction epinow() est une “enveloppe” pour la
fonction estimate_infections() utilisée pour estimer les
cas par date d’infection. La distribution du temps de génération et la
distribution des délais doivent être transmises à l’aide des fonctions
generation_time_opts() et delay_opts()
respectivement.
Il existe de nombreuses autres entrées qui peuvent être transmises à
epinow() voir ?EpiNow2::epinow() pour plus de
détails. Une entrée optionnelle consiste à spécifier un a priori
log-normal pour le nombre de reproduction effectif \(R_t\) au début de l’épidémie. Nous
spécifions une moyenne de 2 jours et un écart-type de 2 jours comme
arguments de la commande prior dans
rt_opts():
R
# define Rt prior distribution
rt_prior <- EpiNow2::rt_opts(prior = EpiNow2::LogNormal(mean = 2, sd = 2))
Inférence bayésienne à l’aide de Stan
L’inférence bayésienne est réalisée à l’aide de méthodes MCMC avec le
programme Stan. Les fonctions Stan
disposent d’un certain nombre d’entrées par défaut, notamment le nombre
de chaînes et le nombre d’échantillons par chaîne (voir
?EpiNow2::stan_opts()).
Pour réduire le temps de calcul, nous pouvons exécuter les chaînes en
parallèle. Pour ce faire, nous devons définir le nombre de cœurs à
utiliser. Par défaut, 4 chaînes MCMC sont exécutées (voir
stan_opts()$chains), nous pouvons donc définir un nombre
égal de cœurs à utiliser en parallèle comme suit :
R
withr::local_options(base::list(mc.cores = 4))
Pour connaître le nombre maximal de cœurs disponibles sur votre
machine, utilisez la commande parallel::detectCores().
Liste de vérification
Note : Dans le code ci-dessous _fixed
les distributions sont utilisées à la place de _variable
(distributions de retard avec incertitude). Cela permet d’accélérer le
temps de calcul. Il est généralement recommandé d’utiliser des
distributions variables qui tiennent compte d’une incertitude
supplémentaire.
R
# fixed alternatives
generation_time_fixed <- EpiNow2::LogNormal(
mean = 3.6,
sd = 3.1,
max = 20
)
reporting_delay_fixed <- EpiNow2::LogNormal(
mean = 2,
sd = 1,
max = 10
)
Vous êtes maintenant prêt à exécuter EpiNow2::epinow()
pour estimer le nombre de reproduction variable dans le temps pour les
90 premiers jours :
R
estimates <- EpiNow2::epinow(
# reported cases
data = cases,
# delays
generation_time = EpiNow2::generation_time_opts(generation_time_fixed),
delays = EpiNow2::delay_opts(incubation_period_fixed + reporting_delay_fixed),
# prior
rt = rt_prior
)
N’attendez pas que cela se poursuive
Pour les besoins de ce tutoriel, nous pouvons optionnellement
utiliser EpiNow2::stan_opts() pour réduire le temps de
calcul. Nous pouvons spécifier un nombre fixe de
samples = 1000 et chains = 2 aux
stan l’argument de la EpiNow2::epinow() de la
fonction. Cette opération devrait prendre environ 3 minutes.
<Nous pouvons optionnellement définir
stan = stan_opts(method = "vb") pour utiliser une méthode
d’échantillonnage approximative. Cela devrait prendre moins d’une
minute. –>
R
# you can add the `stan` argument
EpiNow2::epinow(
...,
stan = EpiNow2::stan_opts(samples = 1000, chains = 3)
)
Rappelez-vous : En utilisant un nombre approprié d’ échantillons et de chaînes est cruciale pour garantir la convergence et obtenir des estimations fiables dans les calculs bayésiens utilisant Stan. L’obtention de résultats plus précis se fait au détriment de la vitesse.
Résultats
Nous pouvons extraire et visualiser des estimations du nombre de reproductions effectives au fil du temps :
R
estimates$plots$R
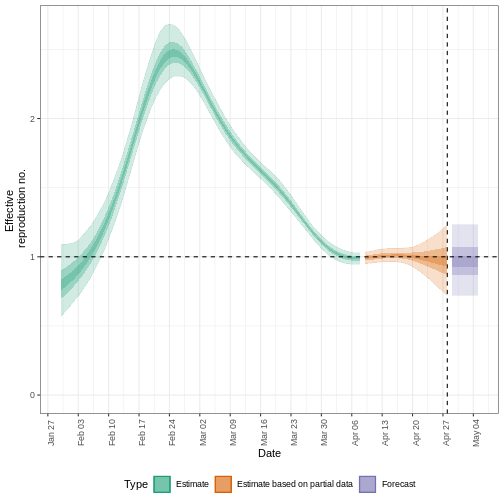
L’incertitude des estimations augmente avec le temps. Cela s’explique par le fait que les estimations sont fondées sur des données antérieures, c’est-à-dire sur les périodes de retard. Cette différence d’incertitude est classée dans les catégories suivantes Estimation (vert) utilise toutes les données et Estimation basée sur des données partielles (orange) des estimations basées sur moins de données (parce que les infections qui se sont produites à l’époque sont plus susceptibles de ne pas avoir encore été observées) et qui ont donc des intervalles de plus en plus larges vers la date du dernier point de données. Enfin, les Prévision (violet) est une projection dans le temps.
Nous pouvons également visualiser l’estimation du taux de croissance dans le temps :
R
estimates$plots$growth_rate
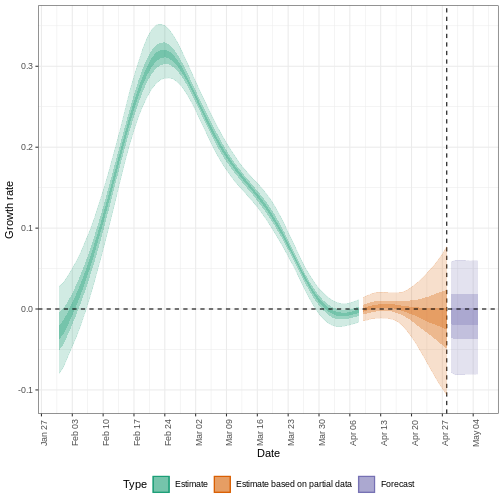
Pour extraire un résumé des principaux paramètres de transmission à dernière date* dans les données :
R
summary(estimates)
SORTIE
measure estimate
<char> <char>
1: New infections per day 7821 (4823 -- 13140)
2: Expected change in reports Likely decreasing
3: Effective reproduction no. 0.96 (0.75 -- 1.2)
4: Rate of growth -0.012 (-0.099 -- 0.079)
5: Doubling/halving time (days) -56 (8.8 -- -7)Ces estimations étant basées sur des données partielles, elles présentent un large intervalle d’incertitude.
Le résumé de notre analyse montre que le changement attendu dans les déclarations est de Likely decreasing avec l’estimation des nouvelles infections 7821 (4823 – 13140).
Le nombre effectif de reproduction \(R_t\) (à la dernière date des données) est de 0.96 (0.75 – 1.2).
Le taux de croissance exponentiel du nombre de cas est de -0.012 (-0.099 – 0.079).
Le temps de doublement (le temps nécessaire pour que le nombre de cas double) est de -56 (8.8 – -7).
Expected change in reports
Un facteur décrivant l’évolution attendue des déclarations sur la base de la probabilité a posteriori que \(R_t < 1\).
| Probabilité (\(p\)) | Changement attendu (\(p\)) |
|---|---|
| p < 0.05$ | Augmentation |
| 0,05$ p< 0,4$ | Augmentation probable |
| 0,4$ p< 0,6$ | Stable |
| 0,6$ p < 0,95$ | Vraisemblablement décroissante |
| 0,95$ p $ | Décroissante |
Intervalles crédibles
En tout EpiNow2 les régions ombrées reflètent les intervalles de crédibilité de 90 %, 50 % et 20 %, du plus clair au plus foncé.
Liste de vérification
EpiNow2 peut être utilisé pour estimer les paramètres de
transmission à partir des données relatives aux cas, à tout moment de
l’évolution d’une épidémie. La fiabilité de ces estimations dépend de la
qualité des données et du choix approprié des distributions de délais.
Dans le prochain tutoriel, nous apprendrons à faire des prévisions et
nous étudierons certaines des options d’inférence supplémentaires
disponibles dans le logiciel EpiNow2.
Défi
Défi
Quantifier l’hétérogénéité géographique
Les données relatives au début de la pandémie de COVID-19 au Royaume-Uni proviennent du package R incidence2 comprennent la région dans laquelle les cas ont été enregistrés. Pour obtenir des estimations régionales du nombre effectif de reproductions et de cas, nous devons formater les données de manière à ce qu’elles comportent trois colonnes :
-
datela date, -
region: la région, -
confirmle nombre de déclarations de maladies (confirmées) pour une région à une date donnée.
Générer des estimations régionales de Rt à partir des
incidence2::covidregionaldataUK à partir de la base de
données :
- utiliser incidence2 pour convertir les données
agrégées en données d’incidence par la variable
region, - conserver les 90 premières dates pour toutes les régions,
- estimer le Rt par région en utilisant le temps de génération défini et les retards dans cet épisode.
R
regional_cases <- incidence2::covidregionaldataUK %>%
# use {tidyr} to preprocess missing values
tidyr::replace_na(base::list(cases_new = 0))
Pour manipuler les données, vous pouvez
R
regional_cases <- incidence2::covidregionaldataUK %>%
# use {tidyr} to preprocess missing values
tidyr::replace_na(base::list(cases_new = 0)) %>%
# use {incidence2} to convert aggregated data to incidence data
incidence2::incidence(
date_index = "date",
groups = "region",
counts = "cases_new",
count_values_to = "confirm",
date_names_to = "date",
complete_dates = TRUE
) %>%
dplyr::select(-count_variable) %>%
dplyr::filter(date < ymd(20200301))
Pour apprendre à faire l’estimation régionale de Rt, lisez la section
de la vignette “Get started” sur regional_epinow() à
l’adresse https://epiforecasts.io/EpiNow2/articles/EpiNow2.html#regional_epinow
Pour obtenir des estimations régionales, nous utilisons les mêmes
arguments de epinow() dans la fonction
regional_epinow():
R
estimates_regional <- EpiNow2::regional_epinow(
# cases
data = regional_cases,
# delays
generation_time = EpiNow2::generation_time_opts(generation_time_fixed),
delays = EpiNow2::delay_opts(incubation_period_fixed + reporting_delay_fixed),
# prior
rt = rt_prior
)
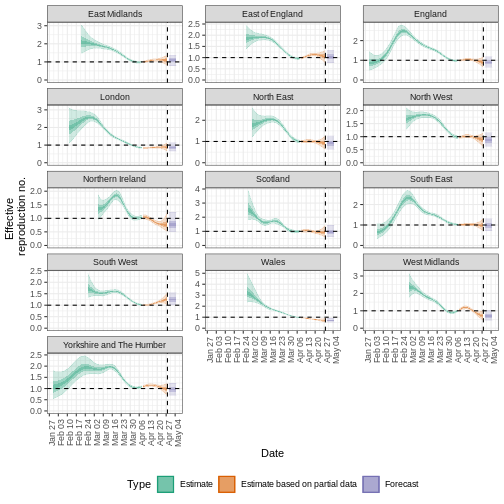
Visualisez les résultats avec :
R
estimates_regional$summary$summarised_results$table
estimates_regional$summary$plots$R
Points clés
- Les paramètres de transmission peuvent être estimés à partir des données du cas après prise en compte des retards.
- L’incertitude peut être prise en compte dans la distribution des retards.
