Utilizar distribuciones de retraso en el análisis
Última actualización: 2024-11-19 | Mejora esta página
Hoja de ruta
Preguntas
- ¿Cómo reutilizar los retrasos almacenados en el paquete
{epiparameter}con mi flujo de análisis existente?
Objetivos
- Utilizar funciones de distribución para distribuciones continuas y
discretas almacenadas como objetos
<epidist>. - Convertir una distribución continua en discreta con
{epiparameter}. - Conectar las salidas de
{epiparameter}con entradas de EpiNow2.
Requisitos previos
- Completar el tutorial Cuantificar la transmisión
Este episodio requiere que estés familiarizado con:
Ciencia de datos : Programación básica con R.
Estadística : Distribuciones de probabilidad.
Teoría epidémica Parámetros epidemiológicos, periodos de tiempo, número reproductivo efectivo.
Introducción
{epiparameter} nos ayuda a elegir un conjunto
específico de parámetros epidemiológicos de la bibliografía, en lugar de
copiarlos/pegarlos a mano:
R
covid_serialint <-
epiparameter::epidist_db(
disease = "covid",
epi_dist = "serial",
author = "Nishiura",
single_epidist = TRUE
)
¡Ahora tenemos un parámetro epidemiológico que podemos utilizar en
nuestro análisis! En el bloque de abajo hemos sustituido uno de los
parámetros de estadísticas de resumen por
EpiNow2::LogNormal()
R
generation_time <-
EpiNow2::LogNormal(
mean = covid_serialint$summary_stats$mean, # replaced!
sd = covid_serialint$summary_stats$sd, # replaced!
max = 20
)
En este episodio, utilizaremos las funciones de
distribución que {epiparameter} proporciona para
obtener un valor máximo (max) para este y cualquier otro
paquete aguas abajo en tu flujo de análisis.
Carguemos los paquetes {epiparameter} y
EpiNow2. En EpiNow2 estableceremos 4
núcleos para utilizarlos en cálculos paralelos. Utilizaremos el operador
pipe %>%, algunos verbos de
dplyr y ggplot2, así que llamemos también
al paquete tidyverse:
R
library(epiparameter)
library(EpiNow2)
library(tidyverse)
withr::local_options(list(mc.cores = 4))
El doble punto
El doble punto :: en R te permite llamar a una función
específica de un paquete sin cargar todo el paquete en el entorno
actual.
Por ejemplo dplyr::filter(data, condition) utiliza
filter() del paquete dplyr.
Esto nos ayuda a recordar las funciones del paquete y a evitar conflictos con los nombres de las funciones.
Funciones de distribución
En R, todas las distribuciones estadísticas tienen funciones para acceder a lo siguiente:
-
density(): Función de densidad de probabilidad (PDF, por sus siglas en inglés), -
cdf(): Función de distribución acumulada (CDF, por sus siglas en inglés), -
quantile(): Cuantil y -
generate(): Generar valores aleatorios de la distribución dada.
Funciones para la distribución Normal
Si lo necesitas, ¡lee en detalle acerca de las funciones de probabilidad de R para la distribución normal, cada una de sus definiciones e identifica en qué parte de una distribución se encuentran!
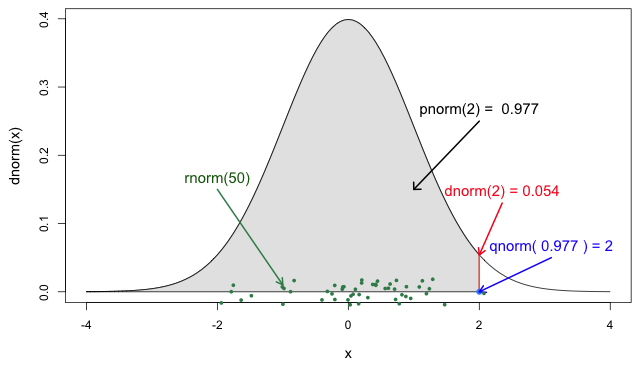
Si consultas ?stats::Distributions, cada tipo de
distribución tiene un conjunto único de funciones. Sin embargo,
¡{epiparameter} te ofrece las mismas cuatro funciones para
acceder a cada uno de los valores anteriores para cualquier objeto
<epidist> que quieras!
R
# Grafica esto para tener una imagen de referencia
plot(covid_serialint, day_range = 0:20)
R
# El valor de densidad cuando el cuantil tiene un valor de 10 (días)
density(covid_serialint, at = 10)
SALIDA
[1] 0.01911607R
# La probabilidad acumulada cuando el cuantil tiene un valor de 10 (días)
cdf(covid_serialint, q = 10)
SALIDA
[1] 0.9466605R
# El valor del cuantil (día) cuando la probabilidad acumulada es 60%
quantile(covid_serialint, p = 0.6)
SALIDA
[1] 4.618906R
# Generar 10 valores aleatorios (días) dada una familia de distribuciones y
# sus parámetros
generate(covid_serialint, times = 10)
SALIDA
[1] 5.702350 8.846805 4.247009 10.232628 7.698895 4.088730 12.172251
[8] 4.556690 5.119388 2.796879Ventana para el rastreo de contactos y el intervalo en serie
El intervalo serial es importante en la optimización del rastreo de contactos, ya que proporciona una ventana temporal para la contención de la propagación de una enfermedad (Fine, 2003). A partir del intervalo serial, podemos evaluar la necesidad de ampliar el número de días previos a tener en cuenta para iniciar el rastreo de contactos e incluir más contactos retrospectivos (Davis et al., 2020).
Con el intervalo de serie COVID-19 (covid_serialint)
calcula:
- ¿Cuántos más casos atrasados se podrían captar si el método de rastreo de contactos considerara los contactos de hasta 6 días antes del inicio en comparación con los de 2 días antes del inicio?
En la Figura 5 de las funciones
de probabilidad de R para la distribución normal, la sección
sombreada representa una probabilidad acumulada de 0.997
para el valor del cuantil en x = 2.
R
plot(covid_serialint)
R
cdf(covid_serialint, q = 2)
SALIDA
[1] 0.1111729R
cdf(covid_serialint, q = 6)
SALIDA
[1] 0.7623645Dado el intervalo de serie COVID-19:
Un método de rastreo de contactos que considere los contactos hasta 2 días antes del inicio captará alrededor del 11,1% de los casos retrospectivos.
Si este periodo se amplía a 6 días antes del inicio, se podría incluir el 76,2% de los contactos retrospectivos.
intercambiamos la pregunta entre días y probabilidad acumulada a:
- Al considerar los casos secundarios, ¿cuántos días después del inicio de los síntomas de los casos primarios podemos esperar que se produzca un 55% de inicio de los síntomas?
R
quantile(covid_serialint, p = 0.55)
Una interpretación podría ser
- El 55% del inicio de los síntomas de los casos secundarios ocurrirá después de 4,2 días del inicio de los síntomas de los casos primarios.
Discretizar una distribución continua
¡Nos acercamos al final! EpiNow2::LogNormal() todavía
necesita un valor máximo (max).
Una forma de hacerlo es obtener el valor del cuantil del percentil 99
de la distribución o la probabilidad acumulada de 0.99 .
Para ello, necesitamos acceder al conjunto de funciones de distribución
de nuestro objeto <epidist>.
Podemos utilizar el conjunto de funciones de distribución de una
distribución continua (como arriba). Sin embargo, estos valores
serán continuos. Podemos discretizar la
distribución continua almacenada en nuestro objeto
<epidist> para obtener valores discretos a partir de
una distribución continua.
Cuando usamos epiparameter::discretise() sobre la
distribución continua, obtenemos una distribución
discreta (o discretizada):
R
covid_serialint_discrete <-
epiparameter::discretise(covid_serialint)
covid_serialint_discrete
SALIDA
Disease: COVID-19
Pathogen: SARS-CoV-2
Epi Distribution: serial interval
Study: Nishiura H, Linton N, Akhmetzhanov A (2020). "Serial interval of novel
coronavirus (COVID-19) infections." _International Journal of
Infectious Diseases_. doi:10.1016/j.ijid.2020.02.060
<https://doi.org/10.1016/j.ijid.2020.02.060>.
Distribution: discrete lnorm
Parameters:
meanlog: 1.386
sdlog: 0.568Identificamos este cambio en la línea de salida
Distribution: del objeto <epidist>.
Comprueba de nuevo esta línea:
Distribution: discrete lnormMientras que para una distribución continua trazamos la Función de densidad de probabilidad (PDF) para una distribución discreta, trazamos la función de masa de probabilidad (PMF):
R
# continua
plot(covid_serialint)
# discreta
plot(covid_serialint_discrete)
Para obtener finalmente un valor máximo (max), accedamos
al valor del cuantil del percentil 99 o cuando la probabilidad acumulada
es 0.99 usando prob_dist$q de forma similar a
como accedemos a los valores de estadísticas de resumen
(summary_stats).
R
covid_serialint_discrete_max <-
quantile(covid_serialint_discrete, p = 0.99)
Duración del periodo de cuarentena e incubación
El periodo de incubación es un retraso útil para evaluar la duración de la vigilancia activa o la cuarentena (Lauer et al., 2020). Del mismo modo, los retrasos desde la aparición de los síntomas hasta la recuperación (o la muerte) determinarán la duración necesaria de la asistencia sanitaria y el aislamiento del caso (Cori et al., 2017).
Calcula:
- ¿En qué plazo exacto de tiempo el 99% de las personas que presentan síntomas de COVID-19 después de la infección los presentan?
¿Qué distribución del retraso mide el tiempo entre la infección y la aparición de los síntomas?
¡Las funciones de probabilidad discretas para
<epidist> son las mismas que utilizamos para las
continuas!
R
# Grafica esto para tener una imagen de referencia
plot(covid_serialint_discrete, day_range = 0:20)
# El valor de la densidad cuando el cuantil tiene un valor de 10 (días)
density(covid_serialint_discrete, at = 10)
# La probabilidad acumulada cuando el cuantil tiene un valor de 10 (días)
cdf(covid_serialint_discrete, q = 10)
# El valor del cuantil (días) cuando la probabilidad acumulada es 60%
quantile(covid_serialint_discrete, p = 0.6)
# Generar valores aleatorios
generate(covid_serialint_discrete, times = 10)
R
covid_incubation <-
epiparameter::epidist_db(
disease = "covid",
epi_dist = "incubation",
single_epidist = TRUE
)
SALIDA
Using Linton N, Kobayashi T, Yang Y, Hayashi K, Akhmetzhanov A, Jung S, Yuan
B, Kinoshita R, Nishiura H (2020). "Incubation Period and Other
Epidemiological Characteristics of 2019 Novel Coronavirus Infections
with Right Truncation: A Statistical Analysis of Publicly Available
Case Data." _Journal of Clinical Medicine_. doi:10.3390/jcm9020538
<https://doi.org/10.3390/jcm9020538>..
To retrieve the citation use the 'get_citation' functionR
covid_incubation_discrete <- epiparameter::discretise(covid_incubation)
quantile(covid_incubation_discrete, p = 0.99)
SALIDA
[1] 19El 99% de los que desarrollan síntomas de COVID-19 lo harán en los 16 días posteriores a la infección.
Ahora, ¿es esperable este resultado en términos epidemiológicos?
A partir de un valor máximo con quantile() podemos crear
una secuencia de valores de cuantiles como un vector numérico y calcular
density() para cada uno:
R
# Crear la visualización para una distribución discreta
# a partir de un valor máximo de la distribución
quantile(covid_serialint_discrete, p = 0.99) %>%
# Generar un vector de cuantiles
# como una secuencia para cada número natural
seq(1L, to = ., by = 1L) %>%
# Convertir el vector numérico en un data.frame (tibble)
as_tibble_col(column_name = "quantile_values") %>%
mutate(
# Calcular los valores de densidad
# para cada cuantul en la función de densidad
density_values =
density(
x = covid_serialint_discrete,
at = quantile_values
)
) %>%
# Graficar
ggplot(
aes(
x = quantile_values,
y = density_values
)
) +
geom_col()
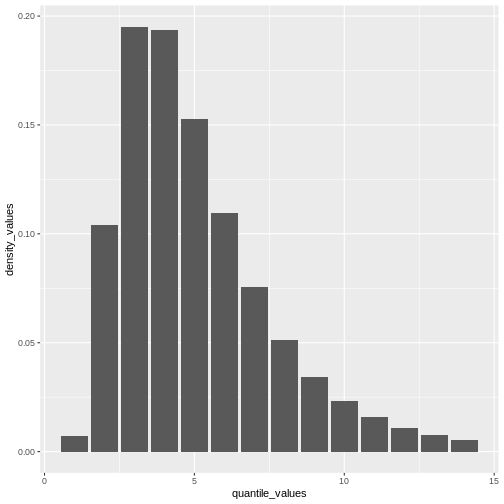
Recuerda: En las infecciones con transmisión presintomática, los intervalos seriales pueden tener valores negativos (Nishiura et al., 2020). ¡Cuando utilizamos el intervalo serial para aproximar el tiempo de generación necesitamos hacer esta distribución sólo con valores positivos!
Plug-in {epiparameter} a {EpiNow2}
¡Ahora podemos introducirlo todo en la función
EpiNow2::LogNormal()!
- Las estadísticas de resumen: media
(
mean) y desviación estándar (sd) de la distribución, - un valor máximo (
max), - el nombre de la distribución (
distribution).
Cuando utilices EpiNow2::LogNormal() para definir una
distribución log normal como la del intervalo serial
del COVID-19 (covid_serialint) podemos especificar la media
(mean) y la desviación estándar (sd) como
parámetros. Alternativamente, para obtener los parámetros “naturales” de
una distribución log normal podemos convertir sus estadísticos de
resumen en parámetros de distribución denominados meanlog y
sdlog. Con {epiparameter} podemos obtener
directamente los parámetros de la distribución utilizando
epiparameter::get_parameters():
R
covid_serialint_parameters <-
epiparameter::get_parameters(covid_serialint)
Entonces, tenemos:
R
serial_interval_covid <-
EpiNow2::LogNormal(
meanlog = covid_serialint_parameters["meanlog"],
sdlog = covid_serialint_parameters["sdlog"],
max = covid_serialint_discrete_max
)
serial_interval_covid
SALIDA
- lognormal distribution (max: 14):
meanlog:
1.4
sdlog:
0.57Suponiendo un escenario con COVID-19, utilicemos los primeros 60 días
del conjunto de datos example_confirmed del paquete
EpiNow2 como casos
reportados(reported_cases) y el recientemente creado
intervalo serial COVID (serial_interval_covid) como
entradas para estimar el número reproductivo variable en el tiempo
utilizando EpiNow2::epinow().
R
epinow_estimates_cg <- epinow(
# casos
data = example_confirmed[1:60],
# retrasos
generation_time = generation_time_opts(serial_interval_covid)
)
SALIDA
WARN [2024-11-19 02:33:14] epinow: There were 2 divergent transitions after warmup. See
https://mc-stan.org/misc/warnings.html#divergent-transitions-after-warmup
to find out why this is a problem and how to eliminate them. -
WARN [2024-11-19 02:33:14] epinow: Examine the pairs() plot to diagnose sampling problems
- R
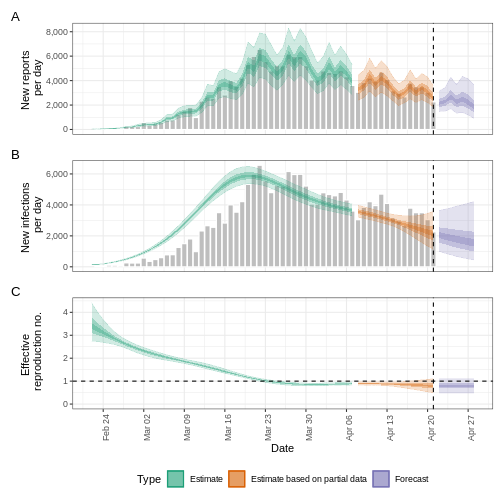
base::plot(epinow_estimates_cg)
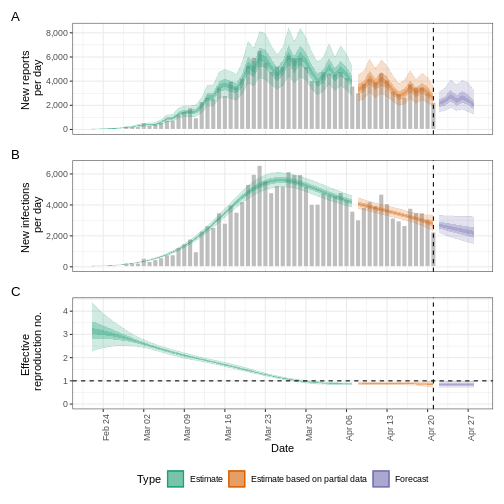
plot() incluye los casos estimados por fecha de
infección, que se reconstruyen a partir de los casos notificados y los
retrasos.
Advertencia
Utilizar el intervalo serial en lugar del tiempo de generación es una alternativa que puede propagar sesgos en tus estimaciones, más aún en enfermedades con transmisión presintomática reportada. (Chung Lau et al., 2021)
Ajuste por retrasos en la notificación
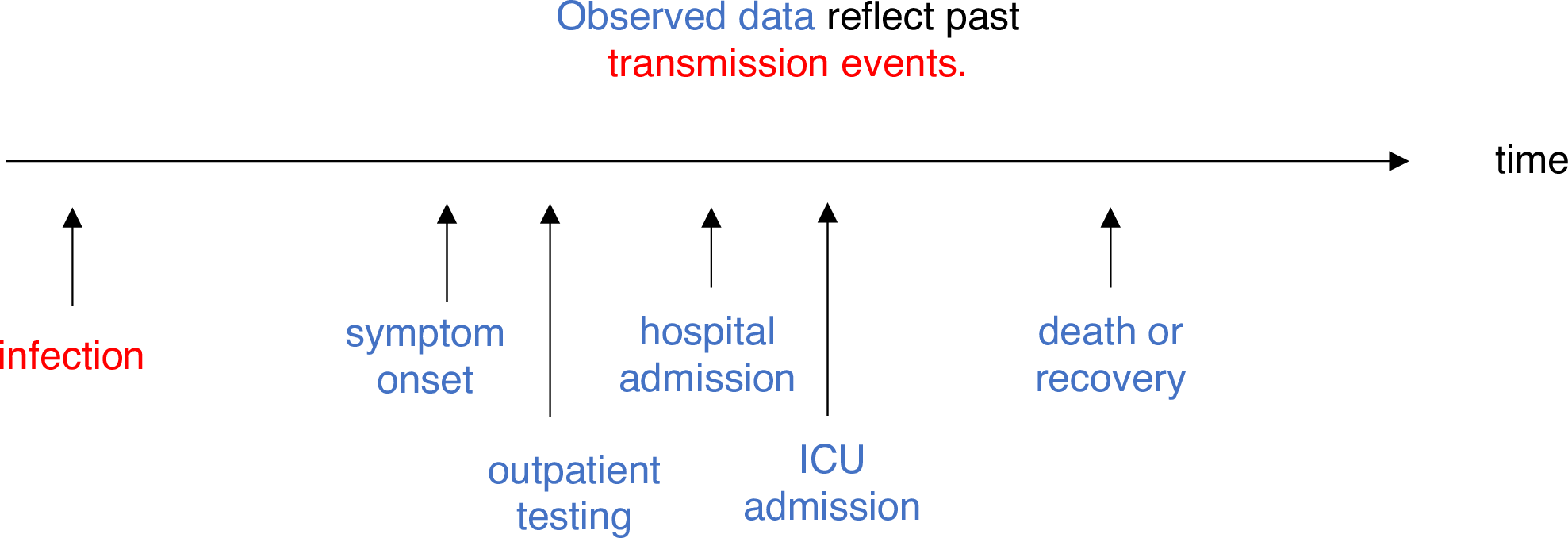
Estimar \(R_t\) requiere datos sobre el número diario de nuevas infecciones. Debido a los retrasos en el desarrollo de cargas víricas detectables, la aparición de síntomas, la búsqueda de atención sanitaria y la notificación, estas cifras no están fácilmente disponibles. Todas las observaciones reflejan eventos de transmisión de algún momento del pasado. En otras palabras, si \(d\) es el tiempo transcurrido desde la infección hasta la observación, las observaciones en el momento \(t\) informan a \(R_{t−d}\) no \(R_t\). (Gostic et al., 2020)
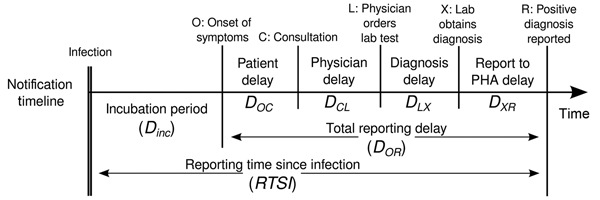
La distribución del retraso podría inferirse
conjuntamente con los tiempos de infección subyacentes o estimarse como
la suma del periodo de
incubación y la distribución de los retrasos desde el inicio de los
síntomas hasta la observación a partir de los datos (retraso en la notificación). En
EpiNow2 podemos especificar estas dos distribuciones de
retraso complementarias con el argumento delays.
Utiliza un periodo de incubación de COVID-19 para estimar Rt
Estima el número reproductivo variable en el tiempo para los primeros
60 días del conjunto de datos example_confirmed de
EpiNow2. Accede a un periodo de incubación para COVID-19
a partir de {epiparameter} para utilizarlo como retraso de
notificación.
Utiliza el último cálculo de epinow() usando el
argumento delays y la función auxiliar
delay_opts().
El argumento delays y la función auxiliar
delay_opts() son análogos al argumento
generation_time y la función auxiliar
generation_time_opts().
R
epinow_estimates <- epinow(
# casos
reported_cases = example_confirmed[1:60],
# retrasos
generation_time = generation_time_opts(covid_serial_interval),
delays = delay_opts(covid_incubation_time)
)
R
# Tiempo de generación ---------------------------------------------------------
# Intervalo serial covid
covid_serialint <-
epiparameter::epidist_db(
disease = "covid",
epi_dist = "serial",
author = "Nishiura",
single_epidist = TRUE
)
# adaptar epidist para epinow2
covid_serialint_discrete_max <- covid_serialint %>%
epiparameter::discretise() %>%
quantile(p = 0.99)
covid_serialint_parameters <-
epiparameter::get_parameters(covid_serialint)
covid_serial_interval <-
EpiNow2::LogNormal(
meanlog = covid_serialint_parameters["meanlog"],
sdlog = covid_serialint_parameters["sdlog"],
max = covid_serialint_discrete_max
)
# Periodo de incubación -------------------------------------------------------
# Periodo de incubación
covid_incubation <- epiparameter::epidist_db(
disease = "covid",
epi_dist = "incubation",
author = "Natalie",
single_epidist = TRUE
)
# Adaptar epiparameter para epinow2
covid_incubation_discrete_max <- covid_incubation %>%
epiparameter::discretise() %>%
quantile(p = 0.99)
covid_incubation_parameters <-
epiparameter::get_parameters(covid_incubation)
covid_incubation_time <-
EpiNow2::LogNormal(
meanlog = covid_incubation_parameters["meanlog"],
sdlog = covid_incubation_parameters["sdlog"],
max = covid_incubation_discrete_max
)
# epinow ------------------------------------------------------------------
# usar epinow
epinow_estimates_cgi <- epinow(
# casos
data = example_confirmed[1:60],
# retrasos
generation_time = generation_time_opts(covid_serial_interval),
delays = delay_opts(covid_incubation_time)
)
SALIDA
WARN [2024-11-19 02:34:51] epinow: There were 11 divergent transitions after warmup. See
https://mc-stan.org/misc/warnings.html#divergent-transitions-after-warmup
to find out why this is a problem and how to eliminate them. -
WARN [2024-11-19 02:34:51] epinow: Examine the pairs() plot to diagnose sampling problems
- R
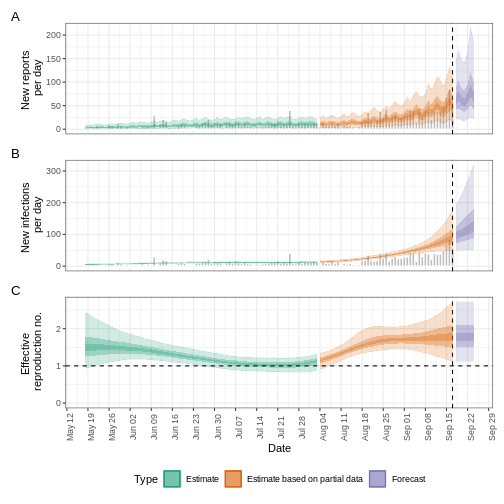
base::plot(epinow_estimates_cgi)
Intenta complementar el argumento delays con un retraso
de notificación como el objeto reporting_delay_fixed del
episodio anterior.
¿Cuánto ha cambiado?
Tras añadir el periodo de incubación, debata acerca de:
- ¿Cambia la tendencia del ajuste del modelo en la sección estimación (“Estimate”)?
- ¿Ha cambiado la incertidumbre?
- ¿Cómo explicaría o interpretaría estos cambios?
Compara todas las figuras generadas con EpiNow2 anteriormente.
Desafíos
Un consejo para completar código
Si escribimos el [ ] luego del objeto
covid_serialint_parameters[ ], dentro de [ ]
podemos utilizar el la tecla Tab en el teclado ↹ para usar la
función
de completado de código
Usar esto permite acceder rápidamente a
covid_serialint_parameters["meanlog"] y
covid_serialint_parameters["sdlog"].
¡Te invitamos a probarlo en otros bloques de código y en la consola de R!
Número de reproducción efectiva del ébola ajustado por retrasos en la notificación
Descarga y lee el conjunto de datos de ébola:
- Estime el número reproductivo efectivo utilizando EpiNow2
- Ajuste la estimación según los retrasos de notificación disponibles
en
{epiparameter} - ¿Por qué eligió ese parámetro?
Para calcular el \(R_t\) utilizando EpiNow2 necesitamos
- Los datos de incidencia agregada con los casos confirmados por día, y
- La distribución del tiempo de generación.
- Opcionalmente, informar las distribuciones de retrasos cuando estén disponibles (por ejemplo, el periodo de incubación).
Para obtener las distribuciones de retrasos utilizando
{epiparameter} podemos utilizar funciones como:
epiparameter::epidist_db()epiparameter::parameter_tbl()discretise()quantile()
R
# Leer datos
# e.j.: Si la ruta al archivo es data/raw-data/ebola_cases.csv entonces:
ebola_confirmed <-
read_csv(here::here("data", "raw-data", "ebola_cases.csv"))
# Listar las distribuciones
epiparameter::epidist_db(disease = "ebola") %>%
epiparameter::parameter_tbl()
R
# Tiempo de generación ---------------------------------------------------------
# Filtrar una distribución para el tiempo de generación
ebola_serial <- epiparameter::epidist_db(
disease = "ebola",
epi_dist = "serial",
single_epidist = TRUE
)
# adaptar epiparameter para epinow2
ebola_serial_discrete <- epiparameter::discretise(ebola_serial)
serial_interval_ebola <-
EpiNow2::Gamma(
mean = ebola_serial$summary_stats$mean,
sd = ebola_serial$summary_stats$sd,
max = quantile(ebola_serial_discrete, p = 0.99)
)
# Tiempo de incubación -------------------------------------------------------
# Filtrar una distribución para el retraso del periodo de incubación
ebola_incubation <- epiparameter::epidist_db(
disease = "ebola",
epi_dist = "incubation",
single_epidist = TRUE
)
# adaptar epiparameter para epinow2
ebola_incubation_discrete <- epiparameter::discretise(ebola_incubation)
incubation_period_ebola <-
EpiNow2::Gamma(
mean = ebola_incubation$summary_stats$mean,
sd = ebola_incubation$summary_stats$sd,
max = quantile(ebola_serial_discrete, p = 0.99)
)
# epinow ------------------------------------------------------------------
# Usar epinow
epinow_estimates_egi <- epinow(
# casos
data = ebola_confirmed,
# retrasos
generation_time = generation_time_opts(serial_interval_ebola),
delays = delay_opts(incubation_period_ebola)
)
SALIDA
WARN [2024-11-19 02:37:24] epinow: There were 181 divergent transitions after warmup. See
https://mc-stan.org/misc/warnings.html#divergent-transitions-after-warmup
to find out why this is a problem and how to eliminate them. -
WARN [2024-11-19 02:37:24] epinow: Examine the pairs() plot to diagnose sampling problems
-
WARN [2024-11-19 02:37:26] epinow: The largest R-hat is NA, indicating chains have not mixed.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#r-hat -
WARN [2024-11-19 02:37:27] epinow: Bulk Effective Samples Size (ESS) is too low, indicating posterior means and medians may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#bulk-ess -
WARN [2024-11-19 02:37:28] epinow: Tail Effective Samples Size (ESS) is too low, indicating posterior variances and tail quantiles may be unreliable.
Running the chains for more iterations may help. See
https://mc-stan.org/misc/warnings.html#tail-ess - R
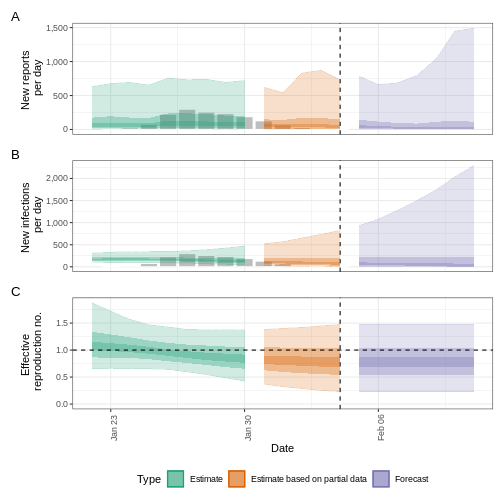
plot(epinow_estimates_egi)
¿Qué hacer con las distribuciones de Weibull?
Utiliza el conjunto de datos
influenza_england_1978_school del paquete
outbreaks para calcular el número reproductivo efectivo
mediante EpiNow2, ajustando por los retrasos de
notificación disponibles en {epiparameter}.
EpiNow2::NonParametric() acepta Funciones de Masa de
Probabilidad (PMF) de cualquier familia de distribuciones. Lee la guía
de referencia sobre Distribuciones
de probabilidad.
R
# ¿Qué parámetros hay disponibles para Influenza?
epiparameter::epidist_db(disease = "influenza") %>%
epiparameter::parameter_tbl() %>%
count(epi_distribution)
SALIDA
# Parameter table:
# A data frame: 3 × 2
epi_distribution n
<chr> <int>
1 generation time 1
2 incubation period 15
3 serial interval 1R
# Tiempo de generación -------------------------------------------------------
# Leer tiempo de generación
influenza_generation <-
epiparameter::epidist_db(
disease = "influenza",
epi_dist = "generation"
)
influenza_generation
SALIDA
Disease: Influenza
Pathogen: Influenza-A-H1N1
Epi Distribution: generation time
Study: Lessler J, Reich N, Cummings D, New York City Department of Health and
Mental Hygiene Swine Influenza Investigation Team (2009). "Outbreak of
2009 Pandemic Influenza A (H1N1) at a New York City School." _The New
England Journal of Medicine_. doi:10.1056/NEJMoa0906089
<https://doi.org/10.1056/NEJMoa0906089>.
Distribution: weibull
Parameters:
shape: 2.360
scale: 3.180R
# EpiNow2 permite usar distribuciones Gamma o LogNormal
# Se puede introducir una PMF
influenza_generation_discrete <-
epiparameter::discretise(influenza_generation)
influenza_generation_max <-
quantile(influenza_generation_discrete, p = 0.99)
influenza_generation_pmf <-
density(
influenza_generation_discrete,
at = 1:influenza_generation_max
)
influenza_generation_pmf
SALIDA
[1] 0.06312336 0.22134988 0.29721220 0.23896828 0.12485164 0.04309454R
# EpiNow2::NonParametric() también puede recibir valores de PMF
generation_time_influenza <-
EpiNow2::NonParametric(
pmf = influenza_generation_pmf
)
# Periodo de incubación -------------------------------------------------------
# Leer el periodo de incubación
influenza_incubation <-
epiparameter::epidist_db(
disease = "influenza",
epi_dist = "incubation",
single_epidist = TRUE
)
# Discretizar el periodo de incubación
influenza_incubation_discrete <-
epiparameter::discretise(influenza_incubation)
influenza_incubation_max <-
quantile(influenza_incubation_discrete, p = 0.99)
influenza_incubation_pmf <-
density(
influenza_incubation_discrete,
at = 1:influenza_incubation_max
)
influenza_incubation_pmf
SALIDA
[1] 0.05749151 0.16687705 0.22443092 0.21507632 0.16104546 0.09746609 0.04841928R
# EpiNow2::NonParametric() también puede recibit valores de PMF
incubation_time_influenza <-
EpiNow2::NonParametric(
pmf = influenza_incubation_pmf
)
# epinow ------------------------------------------------------------------
# Leer datos
influenza_cleaned <-
outbreaks::influenza_england_1978_school %>%
select(date, confirm = in_bed)
# Usar epinow
epinow_estimates_igi <- epinow(
# casos
data = influenza_cleaned,
# retrasos
generation_time = generation_time_opts(generation_time_influenza),
delays = delay_opts(incubation_time_influenza)
)
plot(epinow_estimates_igi)
Próximos pasos
¿Cómo obtener parámetros de distribución a partir de distribuciones estadísticas?
¿Cómo obtener la media y la desviación típica de un tiempo de
generación con sólo parámetros de distribución pero sin
estadísticas de resumen como mean o sd para
EpiNow2::Gamma() o EpiNow2::LogNormal()?
¡Mira en {epiparameter} la viñeta extracción
y conversión de parámetros y sus casos
de uso !
¿Cómo estimar las distribuciones de retraso de la Enfermedad X?
Consulta este excelente tutorial sobre la estimación del intervalo serial y el período de incubación de la Enfermedad X, teniendo en cuenta la censura por medio de inferencia bayesiana con paquetes como rstan y coarseDataTools.
- Tutorial en Inglés: https://rpubs.com/tracelac/diseaseX
- Tutorial en Español: https://epiverse-trace.github.io/epimodelac/EnfermedadX.html
Luego, después de obtener tus valores estimados,
¡puedes crear manualmente tus propios objetos con clase
<epidist> por medio de
epiparameter::epidist()! Echa un vistazo a su guía
de referencia sobre “Crear un objeto <epidist>”
¡!
Por último, echa un vistazo al último paquete de R
{epidist} que proporciona métodos para abordar los
principales retos de la estimación de distribuciones, como el
truncamiento, la censura por intervalos y los sesgos dinámicos.
Puntos Clave
- Utilizar funciones de distribución con
<epidist>para obtener estadísticas de resumen y parámetros informativos de las intervenciones de salud pública, como la Ventana de rastreo de contactos y la Duración de la cuarentena. - Utilizar
discretise()para convertir distribuciones de retraso continuas en discretas. - Utilizar
{epiparameter}para obtener los retrasos de información necesarios en las estimaciones de transmisibilidad.
